Mol:FL3FACGS0027
From Metabolomics.JP
(Difference between revisions)
| Line 1: | Line 1: | ||
| − | + | ||
| − | + | ||
| − | Copyright: ARM project http://www.metabolome.jp/ | + | Copyright: ARM project http://www.metabolome.jp/ |
| − | 43 47 0 0 0 0 0 0 0 0999 V2000 | + | 43 47 0 0 0 0 0 0 0 0999 V2000 |
| − | 0.2085 0.8253 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.2085 0.8253 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.2085 0.3045 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.2085 0.3045 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.6596 0.0440 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.6596 0.0440 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.1106 0.3045 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.1106 0.3045 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.1106 0.8253 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.1106 0.8253 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.6596 1.0857 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.6596 1.0857 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.5617 0.0440 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.5617 0.0440 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.0128 0.3045 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.0128 0.3045 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.0128 0.8253 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.0128 0.8253 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.5617 1.0857 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.5617 1.0857 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.7334 -0.3239 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.7334 -0.3239 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.4637 1.0856 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.4637 1.0856 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.9234 0.8202 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.9234 0.8202 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.3831 1.0856 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.3831 1.0856 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.3831 1.6165 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.3831 1.6165 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.9234 1.8819 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.9234 1.8819 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.4637 1.6165 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.4637 1.6165 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.3110 1.1252 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.3110 1.1252 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.6596 -0.4757 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.6596 -0.4757 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.9775 1.9596 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.9775 1.9596 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.9234 2.5067 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.9234 2.5067 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.9404 0.4349 0.0000 C 0 0 2 0 0 0 0 0 0 0 0 0 | + | -2.9404 0.4349 0.0000 C 0 0 2 0 0 0 0 0 0 0 0 0 |
| − | -2.4248 -0.2457 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0 | + | -2.4248 -0.2457 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0 |
| − | -1.6823 0.0431 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0 | + | -1.6823 0.0431 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0 |
| − | -0.9659 0.0508 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0 | + | -0.9659 0.0508 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0 |
| − | -1.4865 0.5715 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.4865 0.5715 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.2450 0.2992 0.0000 C 0 0 3 0 0 0 0 0 0 0 0 0 | + | -2.2450 0.2992 0.0000 C 0 0 3 0 0 0 0 0 0 0 0 0 |
| − | -3.5170 0.1021 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.5170 0.1021 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.1367 -0.6207 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.1367 -0.6207 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.9949 -0.4244 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.9949 -0.4244 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.7332 -3.3561 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.7332 -3.3561 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.6772 -2.3723 0.0000 C 0 0 2 0 0 0 0 0 0 0 0 0 | + | -1.6772 -2.3723 0.0000 C 0 0 2 0 0 0 0 0 0 0 0 0 |
| − | -0.8244 -2.4148 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0 | + | -0.8244 -2.4148 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0 |
| − | -0.5683 -1.6605 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0 | + | -0.5683 -1.6605 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0 |
| − | -0.1138 -1.1070 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0 | + | -0.1138 -1.1070 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0 |
| − | -0.8473 -1.1708 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.8473 -1.1708 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.1263 -1.9269 0.0000 C 0 0 3 0 0 0 0 0 0 0 0 0 | + | -1.1263 -1.9269 0.0000 C 0 0 3 0 0 0 0 0 0 0 0 0 |
| − | -0.5646 -2.7245 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.5646 -2.7245 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.2521 -1.7939 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.2521 -1.7939 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.8540 0.9061 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.8540 0.9061 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.9260 1.7280 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.9260 1.7280 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.8822 -1.5426 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.8822 -1.5426 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.8022 -1.9342 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.8022 -1.9342 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1 2 1 0 0 0 0 | + | 1 2 1 0 0 0 0 |
| − | 2 3 2 0 0 0 0 | + | 2 3 2 0 0 0 0 |
| − | 3 4 1 0 0 0 0 | + | 3 4 1 0 0 0 0 |
| − | 4 5 2 0 0 0 0 | + | 4 5 2 0 0 0 0 |
| − | 5 6 1 0 0 0 0 | + | 5 6 1 0 0 0 0 |
| − | 6 1 2 0 0 0 0 | + | 6 1 2 0 0 0 0 |
| − | 4 7 1 0 0 0 0 | + | 4 7 1 0 0 0 0 |
| − | 7 8 1 0 0 0 0 | + | 7 8 1 0 0 0 0 |
| − | 8 9 2 0 0 0 0 | + | 8 9 2 0 0 0 0 |
| − | 9 10 1 0 0 0 0 | + | 9 10 1 0 0 0 0 |
| − | 10 5 1 0 0 0 0 | + | 10 5 1 0 0 0 0 |
| − | 7 11 2 0 0 0 0 | + | 7 11 2 0 0 0 0 |
| − | 9 12 1 0 0 0 0 | + | 9 12 1 0 0 0 0 |
| − | 12 13 2 0 0 0 0 | + | 12 13 2 0 0 0 0 |
| − | 13 14 1 0 0 0 0 | + | 13 14 1 0 0 0 0 |
| − | 14 15 2 0 0 0 0 | + | 14 15 2 0 0 0 0 |
| − | 15 16 1 0 0 0 0 | + | 15 16 1 0 0 0 0 |
| − | 16 17 2 0 0 0 0 | + | 16 17 2 0 0 0 0 |
| − | 17 12 1 0 0 0 0 | + | 17 12 1 0 0 0 0 |
| − | 18 1 1 0 0 0 0 | + | 18 1 1 0 0 0 0 |
| − | 3 19 1 0 0 0 0 | + | 3 19 1 0 0 0 0 |
| − | 15 20 1 0 0 0 0 | + | 15 20 1 0 0 0 0 |
| − | 16 21 1 0 0 0 0 | + | 16 21 1 0 0 0 0 |
| − | 22 23 1 1 0 0 0 | + | 22 23 1 1 0 0 0 |
| − | 23 24 1 1 0 0 0 | + | 23 24 1 1 0 0 0 |
| − | 25 24 1 1 0 0 0 | + | 25 24 1 1 0 0 0 |
| − | 25 26 1 0 0 0 0 | + | 25 26 1 0 0 0 0 |
| − | 26 27 1 0 0 0 0 | + | 26 27 1 0 0 0 0 |
| − | 27 22 1 0 0 0 0 | + | 27 22 1 0 0 0 0 |
| − | 22 28 1 0 0 0 0 | + | 22 28 1 0 0 0 0 |
| − | 23 29 1 0 0 0 0 | + | 23 29 1 0 0 0 0 |
| − | 24 30 1 0 0 0 0 | + | 24 30 1 0 0 0 0 |
| − | 32 33 1 1 0 0 0 | + | 32 33 1 1 0 0 0 |
| − | 33 34 1 1 0 0 0 | + | 33 34 1 1 0 0 0 |
| − | 35 34 1 1 0 0 0 | + | 35 34 1 1 0 0 0 |
| − | 35 36 1 0 0 0 0 | + | 35 36 1 0 0 0 0 |
| − | 36 37 1 0 0 0 0 | + | 36 37 1 0 0 0 0 |
| − | 37 32 1 0 0 0 0 | + | 37 32 1 0 0 0 0 |
| − | 33 38 1 0 0 0 0 | + | 33 38 1 0 0 0 0 |
| − | 34 39 1 0 0 0 0 | + | 34 39 1 0 0 0 0 |
| − | 31 32 1 0 0 0 0 | + | 31 32 1 0 0 0 0 |
| − | 35 30 1 0 0 0 0 | + | 35 30 1 0 0 0 0 |
| − | 25 18 1 0 0 0 0 | + | 25 18 1 0 0 0 0 |
| − | 27 40 1 0 0 0 0 | + | 27 40 1 0 0 0 0 |
| − | 40 41 1 0 0 0 0 | + | 40 41 1 0 0 0 0 |
| − | 37 42 1 0 0 0 0 | + | 37 42 1 0 0 0 0 |
| − | 42 43 1 0 0 0 0 | + | 42 43 1 0 0 0 0 |
| − | M STY 1 2 SUP | + | M STY 1 2 SUP |
| − | M SLB 1 2 2 | + | M SLB 1 2 2 |
| − | M SAL 2 2 42 43 | + | M SAL 2 2 42 43 |
| − | M SBL 2 1 46 | + | M SBL 2 1 46 |
| − | M SMT 2 CH2OH | + | M SMT 2 CH2OH |
| − | M SVB 2 46 -2.6841 -0.7103 | + | M SVB 2 46 -2.6841 -0.7103 |
| − | M STY 1 1 SUP | + | M STY 1 1 SUP |
| − | M SLB 1 1 1 | + | M SLB 1 1 1 |
| − | M SAL 1 2 40 41 | + | M SAL 1 2 40 41 |
| − | M SBL 1 1 44 | + | M SBL 1 1 44 |
| − | M SMT 1 CH2OH | + | M SMT 1 CH2OH |
| − | M SVB 1 44 -2.5405 1.1066 | + | M SVB 1 44 -2.5405 1.1066 |
| − | S SKP 8 | + | S SKP 8 |
| − | ID FL3FACGS0027 | + | ID FL3FACGS0027 |
| − | KNApSAcK_ID C00004286 | + | KNApSAcK_ID C00004286 |
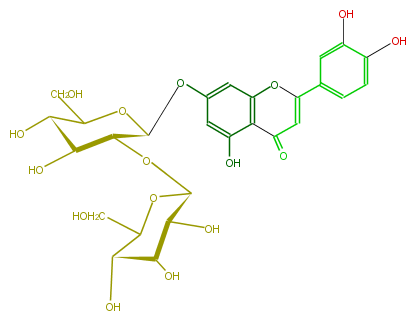
| − | NAME Luteolin 7-allosyl-(1->2)-glucoside | + | NAME Luteolin 7-allosyl-(1->2)-glucoside |
| − | CAS_RN 113471-89-9 | + | CAS_RN 113471-89-9 |
| − | FORMULA C27H30O16 | + | FORMULA C27H30O16 |
| − | EXACTMASS 610.153384912 | + | EXACTMASS 610.153384912 |
| − | AVERAGEMASS 610.5175 | + | AVERAGEMASS 610.5175 |
| − | SMILES c(c1)c(c(O)cc1C(=C5)Oc(c(C(=O)5)2)cc(O[C@H](O4)[C@H]([C@@H](O)[C@H](C4CO)O)O[C@H](O3)[C@H]([C@@H](O)[C@H](C3CO)O)O)cc2O)O | + | SMILES c(c1)c(c(O)cc1C(=C5)Oc(c(C(=O)5)2)cc(O[C@H](O4)[C@H]([C@@H](O)[C@H](C4CO)O)O[C@H](O3)[C@H]([C@@H](O)[C@H](C3CO)O)O)cc2O)O |
M END | M END | ||
| − | |||
Latest revision as of 09:00, 14 March 2009
Copyright: ARM project http://www.metabolome.jp/
43 47 0 0 0 0 0 0 0 0999 V2000
0.2085 0.8253 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.2085 0.3045 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.6596 0.0440 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.1106 0.3045 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.1106 0.8253 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.6596 1.0857 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.5617 0.0440 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.0128 0.3045 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.0128 0.8253 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.5617 1.0857 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
1.7334 -0.3239 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
2.4637 1.0856 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.9234 0.8202 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.3831 1.0856 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.3831 1.6165 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.9234 1.8819 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.4637 1.6165 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.3110 1.1252 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
0.6596 -0.4757 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
3.9775 1.9596 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
2.9234 2.5067 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-2.9404 0.4349 0.0000 C 0 0 2 0 0 0 0 0 0 0 0 0
-2.4248 -0.2457 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0
-1.6823 0.0431 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0
-0.9659 0.0508 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0
-1.4865 0.5715 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-2.2450 0.2992 0.0000 C 0 0 3 0 0 0 0 0 0 0 0 0
-3.5170 0.1021 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-3.1367 -0.6207 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-0.9949 -0.4244 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-1.7332 -3.3561 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-1.6772 -2.3723 0.0000 C 0 0 2 0 0 0 0 0 0 0 0 0
-0.8244 -2.4148 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0
-0.5683 -1.6605 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0
-0.1138 -1.1070 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0
-0.8473 -1.1708 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-1.1263 -1.9269 0.0000 C 0 0 3 0 0 0 0 0 0 0 0 0
-0.5646 -2.7245 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
0.2521 -1.7939 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-2.8540 0.9061 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.9260 1.7280 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-1.8822 -1.5426 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.8022 -1.9342 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
1 2 1 0 0 0 0
2 3 2 0 0 0 0
3 4 1 0 0 0 0
4 5 2 0 0 0 0
5 6 1 0 0 0 0
6 1 2 0 0 0 0
4 7 1 0 0 0 0
7 8 1 0 0 0 0
8 9 2 0 0 0 0
9 10 1 0 0 0 0
10 5 1 0 0 0 0
7 11 2 0 0 0 0
9 12 1 0 0 0 0
12 13 2 0 0 0 0
13 14 1 0 0 0 0
14 15 2 0 0 0 0
15 16 1 0 0 0 0
16 17 2 0 0 0 0
17 12 1 0 0 0 0
18 1 1 0 0 0 0
3 19 1 0 0 0 0
15 20 1 0 0 0 0
16 21 1 0 0 0 0
22 23 1 1 0 0 0
23 24 1 1 0 0 0
25 24 1 1 0 0 0
25 26 1 0 0 0 0
26 27 1 0 0 0 0
27 22 1 0 0 0 0
22 28 1 0 0 0 0
23 29 1 0 0 0 0
24 30 1 0 0 0 0
32 33 1 1 0 0 0
33 34 1 1 0 0 0
35 34 1 1 0 0 0
35 36 1 0 0 0 0
36 37 1 0 0 0 0
37 32 1 0 0 0 0
33 38 1 0 0 0 0
34 39 1 0 0 0 0
31 32 1 0 0 0 0
35 30 1 0 0 0 0
25 18 1 0 0 0 0
27 40 1 0 0 0 0
40 41 1 0 0 0 0
37 42 1 0 0 0 0
42 43 1 0 0 0 0
M STY 1 2 SUP
M SLB 1 2 2
M SAL 2 2 42 43
M SBL 2 1 46
M SMT 2 CH2OH
M SVB 2 46 -2.6841 -0.7103
M STY 1 1 SUP
M SLB 1 1 1
M SAL 1 2 40 41
M SBL 1 1 44
M SMT 1 CH2OH
M SVB 1 44 -2.5405 1.1066
S SKP 8
ID FL3FACGS0027
KNApSAcK_ID C00004286
NAME Luteolin 7-allosyl-(1->2)-glucoside
CAS_RN 113471-89-9
FORMULA C27H30O16
EXACTMASS 610.153384912
AVERAGEMASS 610.5175
SMILES c(c1)c(c(O)cc1C(=C5)Oc(c(C(=O)5)2)cc(O[C@H](O4)[C@H]([C@@H](O)[C@H](C4CO)O)O[C@H](O3)[C@H]([C@@H](O)[C@H](C3CO)O)O)cc2O)O
M END