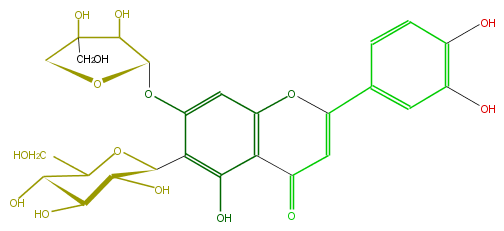
Mol:FL3FACDS0022
From Metabolomics.JP
(Difference between revisions)
| Line 1: | Line 1: | ||
| − | + | ||
| − | + | ||
| − | Copyright: ARM project http://www.metabolome.jp/ | + | Copyright: ARM project http://www.metabolome.jp/ |
| − | 41 45 0 0 0 0 0 0 0 0999 V2000 | + | 41 45 0 0 0 0 0 0 0 0999 V2000 |
| − | -0.9933 0.0216 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.9933 0.0216 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.9933 -0.8034 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.9933 -0.8034 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.2788 -1.2159 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.2788 -1.2159 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.4357 -0.8034 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.4357 -0.8034 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.4357 0.0216 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.4357 0.0216 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.2788 0.4341 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.2788 0.4341 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.1501 -1.2159 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.1501 -1.2159 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.8646 -0.8034 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.8646 -0.8034 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.8646 0.0216 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.8646 0.0216 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.1501 0.4341 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.1501 0.4341 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.1501 -2.0040 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.1501 -2.0040 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.2788 -2.0404 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.2788 -2.0404 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.7377 0.5664 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.7377 0.5664 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.4905 0.1317 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.4905 0.1317 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 4.2434 0.5664 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 4.2434 0.5664 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 4.2434 1.4357 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 4.2434 1.4357 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.4905 1.8703 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.4905 1.8703 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.7377 1.4357 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.7377 1.4357 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 4.9959 1.8701 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 4.9959 1.8701 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.7074 0.4339 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.7074 0.4339 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.8306 -1.2371 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.8306 -1.2371 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.9989 -1.7820 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.9989 -1.7820 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.4620 -1.2270 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.4620 -1.2270 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.5647 -1.0962 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.5647 -1.0962 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.3297 -0.6978 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.3297 -0.6978 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.9313 -1.2155 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.9313 -1.2155 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -4.4130 -1.7397 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -4.4130 -1.7397 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.7670 -1.9548 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.7670 -1.9548 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.5082 -1.4825 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.5082 -1.4825 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.3127 1.5438 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.3127 1.5438 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.1170 1.5438 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.1170 1.5438 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.7886 1.0939 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.7886 1.0939 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.7439 0.6453 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.7439 0.6453 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.7136 1.0496 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.7136 1.0496 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.1170 2.0199 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.1170 2.0199 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.3127 2.0404 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.3127 2.0404 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 4.9959 0.1320 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 4.9959 0.1320 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.7125 -0.7715 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.7125 -0.7715 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -4.9959 -0.7214 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -4.9959 -0.7214 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.0939 1.1337 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.0939 1.1337 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.9618 1.7414 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.9618 1.7414 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1 2 1 0 0 0 0 | + | 1 2 1 0 0 0 0 |
| − | 2 3 2 0 0 0 0 | + | 2 3 2 0 0 0 0 |
| − | 3 4 1 0 0 0 0 | + | 3 4 1 0 0 0 0 |
| − | 4 5 2 0 0 0 0 | + | 4 5 2 0 0 0 0 |
| − | 5 6 1 0 0 0 0 | + | 5 6 1 0 0 0 0 |
| − | 6 1 2 0 0 0 0 | + | 6 1 2 0 0 0 0 |
| − | 4 7 1 0 0 0 0 | + | 4 7 1 0 0 0 0 |
| − | 7 8 1 0 0 0 0 | + | 7 8 1 0 0 0 0 |
| − | 8 9 2 0 0 0 0 | + | 8 9 2 0 0 0 0 |
| − | 9 10 1 0 0 0 0 | + | 9 10 1 0 0 0 0 |
| − | 10 5 1 0 0 0 0 | + | 10 5 1 0 0 0 0 |
| − | 7 11 2 0 0 0 0 | + | 7 11 2 0 0 0 0 |
| − | 3 12 1 0 0 0 0 | + | 3 12 1 0 0 0 0 |
| − | 13 14 2 0 0 0 0 | + | 13 14 2 0 0 0 0 |
| − | 14 15 1 0 0 0 0 | + | 14 15 1 0 0 0 0 |
| − | 15 16 2 0 0 0 0 | + | 15 16 2 0 0 0 0 |
| − | 16 17 1 0 0 0 0 | + | 16 17 1 0 0 0 0 |
| − | 17 18 2 0 0 0 0 | + | 17 18 2 0 0 0 0 |
| − | 18 13 1 0 0 0 0 | + | 18 13 1 0 0 0 0 |
| − | 9 13 1 0 0 0 0 | + | 9 13 1 0 0 0 0 |
| − | 16 19 1 0 0 0 0 | + | 16 19 1 0 0 0 0 |
| − | 1 20 1 0 0 0 0 | + | 1 20 1 0 0 0 0 |
| − | 21 22 1 1 0 0 0 | + | 21 22 1 1 0 0 0 |
| − | 22 23 1 1 0 0 0 | + | 22 23 1 1 0 0 0 |
| − | 24 23 1 1 0 0 0 | + | 24 23 1 1 0 0 0 |
| − | 24 25 1 0 0 0 0 | + | 24 25 1 0 0 0 0 |
| − | 25 26 1 0 0 0 0 | + | 25 26 1 0 0 0 0 |
| − | 26 21 1 0 0 0 0 | + | 26 21 1 0 0 0 0 |
| − | 21 27 1 0 0 0 0 | + | 21 27 1 0 0 0 0 |
| − | 22 28 1 0 0 0 0 | + | 22 28 1 0 0 0 0 |
| − | 23 29 1 0 0 0 0 | + | 23 29 1 0 0 0 0 |
| − | 24 2 1 0 0 0 0 | + | 24 2 1 0 0 0 0 |
| − | 30 31 1 0 0 0 0 | + | 30 31 1 0 0 0 0 |
| − | 31 32 1 0 0 0 0 | + | 31 32 1 0 0 0 0 |
| − | 32 33 1 1 0 0 0 | + | 32 33 1 1 0 0 0 |
| − | 34 33 1 1 0 0 0 | + | 34 33 1 1 0 0 0 |
| − | 34 30 1 0 0 0 0 | + | 34 30 1 0 0 0 0 |
| − | 31 35 1 0 0 0 0 | + | 31 35 1 0 0 0 0 |
| − | 30 36 1 0 0 0 0 | + | 30 36 1 0 0 0 0 |
| − | 20 34 1 0 0 0 0 | + | 20 34 1 0 0 0 0 |
| − | 15 37 1 0 0 0 0 | + | 15 37 1 0 0 0 0 |
| − | 38 39 1 0 0 0 0 | + | 38 39 1 0 0 0 0 |
| − | 26 38 1 0 0 0 0 | + | 26 38 1 0 0 0 0 |
| − | 40 41 1 0 0 0 0 | + | 40 41 1 0 0 0 0 |
| − | 31 40 1 0 0 0 0 | + | 31 40 1 0 0 0 0 |
| − | M STY 1 1 SUP | + | M STY 1 1 SUP |
| − | M SLB 1 1 1 | + | M SLB 1 1 1 |
| − | M SAL 1 2 38 39 | + | M SAL 1 2 38 39 |
| − | M SBL 1 1 43 | + | M SBL 1 1 43 |
| − | M SMT 1 ^ CH2OH | + | M SMT 1 ^ CH2OH |
| − | M SBV 1 43 0.7812 -0.4439 | + | M SBV 1 43 0.7812 -0.4439 |
| − | M STY 1 2 SUP | + | M STY 1 2 SUP |
| − | M SLB 1 2 2 | + | M SLB 1 2 2 |
| − | M SAL 2 2 40 41 | + | M SAL 2 2 40 41 |
| − | M SBL 2 1 45 | + | M SBL 2 1 45 |
| − | M SMT 2 CH2OH | + | M SMT 2 CH2OH |
| − | M SBV 2 45 -0.0231 0.4101 | + | M SBV 2 45 -0.0231 0.4101 |
| − | S SKP 5 | + | S SKP 5 |
| − | ID FL3FACDS0022 | + | ID FL3FACDS0022 |
| − | FORMULA C26H28O15 | + | FORMULA C26H28O15 |
| − | EXACTMASS 580.1428202259999 | + | EXACTMASS 580.1428202259999 |
| − | AVERAGEMASS 580.49152 | + | AVERAGEMASS 580.49152 |
| − | SMILES O(C5)C(C(C5(O)CO)O)Oc(c(C(C(O)4)OC(C(O)C(O)4)CO)3)cc(c2c3O)OC(=CC2=O)c(c1)ccc(O)c1O | + | SMILES O(C5)C(C(C5(O)CO)O)Oc(c(C(C(O)4)OC(C(O)C(O)4)CO)3)cc(c2c3O)OC(=CC2=O)c(c1)ccc(O)c1O |
M END | M END | ||
| − | |||
Latest revision as of 09:00, 14 March 2009
Copyright: ARM project http://www.metabolome.jp/
41 45 0 0 0 0 0 0 0 0999 V2000
-0.9933 0.0216 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.9933 -0.8034 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.2788 -1.2159 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.4357 -0.8034 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.4357 0.0216 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.2788 0.4341 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.1501 -1.2159 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.8646 -0.8034 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.8646 0.0216 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.1501 0.4341 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
1.1501 -2.0040 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-0.2788 -2.0404 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
2.7377 0.5664 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.4905 0.1317 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
4.2434 0.5664 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
4.2434 1.4357 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.4905 1.8703 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.7377 1.4357 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
4.9959 1.8701 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-1.7074 0.4339 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-3.8306 -1.2371 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.9989 -1.7820 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.4620 -1.2270 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.5647 -1.0962 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.3297 -0.6978 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-2.9313 -1.2155 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-4.4130 -1.7397 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-3.7670 -1.9548 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-1.5082 -1.4825 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-2.3127 1.5438 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-3.1170 1.5438 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-3.7886 1.0939 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.7439 0.6453 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-1.7136 1.0496 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-3.1170 2.0199 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-2.3127 2.0404 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
4.9959 0.1320 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-3.7125 -0.7715 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-4.9959 -0.7214 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-3.0939 1.1337 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-3.9618 1.7414 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
1 2 1 0 0 0 0
2 3 2 0 0 0 0
3 4 1 0 0 0 0
4 5 2 0 0 0 0
5 6 1 0 0 0 0
6 1 2 0 0 0 0
4 7 1 0 0 0 0
7 8 1 0 0 0 0
8 9 2 0 0 0 0
9 10 1 0 0 0 0
10 5 1 0 0 0 0
7 11 2 0 0 0 0
3 12 1 0 0 0 0
13 14 2 0 0 0 0
14 15 1 0 0 0 0
15 16 2 0 0 0 0
16 17 1 0 0 0 0
17 18 2 0 0 0 0
18 13 1 0 0 0 0
9 13 1 0 0 0 0
16 19 1 0 0 0 0
1 20 1 0 0 0 0
21 22 1 1 0 0 0
22 23 1 1 0 0 0
24 23 1 1 0 0 0
24 25 1 0 0 0 0
25 26 1 0 0 0 0
26 21 1 0 0 0 0
21 27 1 0 0 0 0
22 28 1 0 0 0 0
23 29 1 0 0 0 0
24 2 1 0 0 0 0
30 31 1 0 0 0 0
31 32 1 0 0 0 0
32 33 1 1 0 0 0
34 33 1 1 0 0 0
34 30 1 0 0 0 0
31 35 1 0 0 0 0
30 36 1 0 0 0 0
20 34 1 0 0 0 0
15 37 1 0 0 0 0
38 39 1 0 0 0 0
26 38 1 0 0 0 0
40 41 1 0 0 0 0
31 40 1 0 0 0 0
M STY 1 1 SUP
M SLB 1 1 1
M SAL 1 2 38 39
M SBL 1 1 43
M SMT 1 ^ CH2OH
M SBV 1 43 0.7812 -0.4439
M STY 1 2 SUP
M SLB 1 2 2
M SAL 2 2 40 41
M SBL 2 1 45
M SMT 2 CH2OH
M SBV 2 45 -0.0231 0.4101
S SKP 5
ID FL3FACDS0022
FORMULA C26H28O15
EXACTMASS 580.1428202259999
AVERAGEMASS 580.49152
SMILES O(C5)C(C(C5(O)CO)O)Oc(c(C(C(O)4)OC(C(O)C(O)4)CO)3)cc(c2c3O)OC(=CC2=O)c(c1)ccc(O)c1O
M END