Mol:FL3FACDS0019
From Metabolomics.JP
(Difference between revisions)
| Line 1: | Line 1: | ||
| − | + | ||
| − | + | ||
| − | Copyright: ARM project http://www.metabolome.jp/ | + | Copyright: ARM project http://www.metabolome.jp/ |
| − | 66 72 0 0 0 0 0 0 0 0999 V2000 | + | 66 72 0 0 0 0 0 0 0 0999 V2000 |
| − | -1.7407 0.5702 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.7407 0.5702 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.7407 -0.2548 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.7407 -0.2548 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.0261 -0.6673 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.0261 -0.6673 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.3116 -0.2548 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.3116 -0.2548 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.3116 0.5702 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.3116 0.5702 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.0261 0.9828 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.0261 0.9828 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.4030 -0.6673 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.4030 -0.6673 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.1175 -0.2548 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.1175 -0.2548 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.1175 0.5702 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.1175 0.5702 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.4030 0.9828 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.4030 0.9828 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.4030 -1.3107 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.4030 -1.3107 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.4549 0.9827 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.4549 0.9827 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.0261 -1.4921 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.0261 -1.4921 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.9113 1.0091 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.9113 1.0091 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.6642 0.5745 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.6642 0.5745 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.4172 1.0091 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.4172 1.0091 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.4172 1.8786 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.4172 1.8786 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.6642 2.3134 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.6642 2.3134 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.9113 1.8786 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.9113 1.8786 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.9971 2.3334 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.9971 2.3334 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -4.1371 -0.2134 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -4.1371 -0.2134 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.6603 -0.8427 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.6603 -0.8427 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.9737 -0.5757 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.9737 -0.5757 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.3114 -0.5685 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.3114 -0.5685 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.7926 -0.0871 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.7926 -0.0871 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.3932 -0.4041 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.3932 -0.4041 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -4.8523 -0.4050 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -4.8523 -0.4050 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -4.3160 -0.8427 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -4.3160 -0.8427 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.2729 -0.9254 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.2729 -0.9254 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.9630 5.3431 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.9630 5.3431 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.1851 4.7539 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.1851 4.7539 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.5152 3.9053 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.5152 3.9053 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.5240 3.0865 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.5240 3.0865 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 4.1192 3.6815 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 4.1192 3.6815 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.7273 4.4239 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.7273 4.4239 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.7894 5.9907 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.7894 5.9907 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.1851 5.5104 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.1851 5.5104 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.1091 3.2018 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.1091 3.2018 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.5598 -1.7589 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.5598 -1.7589 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.1968 -2.1266 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.1968 -2.1266 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.8869 -2.1473 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.8869 -2.1473 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.8869 -3.1623 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.8869 -3.1623 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.3279 -3.5515 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.3279 -3.5515 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.3279 -4.3592 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.3279 -4.3592 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.6284 -4.7632 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.6284 -4.7632 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.0712 -4.3592 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.0712 -4.3592 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.0712 -3.5515 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.0712 -3.5515 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.6284 -3.1476 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.6284 -3.1476 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.6284 -5.5707 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.6284 -5.5707 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.7977 -4.8193 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.7977 -4.8193 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 4.1697 0.5747 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 4.1697 0.5747 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.0022 -4.2619 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.0022 -4.2619 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.6157 -4.9312 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.6157 -4.9312 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.3588 -4.7188 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.3588 -4.7188 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.1063 -4.9312 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.1063 -4.9312 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.4928 -4.2619 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.4928 -4.2619 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.7498 -4.4742 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.7498 -4.4742 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.2846 -4.4542 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.2846 -4.4542 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.2423 -3.9816 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.2423 -3.9816 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 4.0503 -4.5839 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 4.0503 -4.5839 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.9552 0.1578 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.9552 0.1578 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -5.1814 0.5402 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -5.1814 0.5402 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.8158 -4.9312 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.8158 -4.9312 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.8158 -5.9907 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.8158 -5.9907 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 4.1579 4.8545 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 4.1579 4.8545 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 5.1814 4.5802 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 5.1814 4.5802 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1 2 1 0 0 0 0 | + | 1 2 1 0 0 0 0 |
| − | 2 3 2 0 0 0 0 | + | 2 3 2 0 0 0 0 |
| − | 3 4 1 0 0 0 0 | + | 3 4 1 0 0 0 0 |
| − | 4 5 2 0 0 0 0 | + | 4 5 2 0 0 0 0 |
| − | 5 6 1 0 0 0 0 | + | 5 6 1 0 0 0 0 |
| − | 6 1 2 0 0 0 0 | + | 6 1 2 0 0 0 0 |
| − | 4 7 1 0 0 0 0 | + | 4 7 1 0 0 0 0 |
| − | 7 8 1 0 0 0 0 | + | 7 8 1 0 0 0 0 |
| − | 8 9 2 0 0 0 0 | + | 8 9 2 0 0 0 0 |
| − | 9 10 1 0 0 0 0 | + | 9 10 1 0 0 0 0 |
| − | 10 5 1 0 0 0 0 | + | 10 5 1 0 0 0 0 |
| − | 7 11 2 0 0 0 0 | + | 7 11 2 0 0 0 0 |
| − | 1 12 1 0 0 0 0 | + | 1 12 1 0 0 0 0 |
| − | 3 13 1 0 0 0 0 | + | 3 13 1 0 0 0 0 |
| − | 14 15 2 0 0 0 0 | + | 14 15 2 0 0 0 0 |
| − | 15 16 1 0 0 0 0 | + | 15 16 1 0 0 0 0 |
| − | 16 17 2 0 0 0 0 | + | 16 17 2 0 0 0 0 |
| − | 17 18 1 0 0 0 0 | + | 17 18 1 0 0 0 0 |
| − | 18 19 2 0 0 0 0 | + | 18 19 2 0 0 0 0 |
| − | 19 14 1 0 0 0 0 | + | 19 14 1 0 0 0 0 |
| − | 14 9 1 0 0 0 0 | + | 14 9 1 0 0 0 0 |
| − | 17 20 1 0 0 0 0 | + | 17 20 1 0 0 0 0 |
| − | 21 22 1 1 0 0 0 | + | 21 22 1 1 0 0 0 |
| − | 22 23 1 1 0 0 0 | + | 22 23 1 1 0 0 0 |
| − | 24 23 1 1 0 0 0 | + | 24 23 1 1 0 0 0 |
| − | 24 25 1 0 0 0 0 | + | 24 25 1 0 0 0 0 |
| − | 25 26 1 0 0 0 0 | + | 25 26 1 0 0 0 0 |
| − | 26 21 1 0 0 0 0 | + | 26 21 1 0 0 0 0 |
| − | 21 27 1 0 0 0 0 | + | 21 27 1 0 0 0 0 |
| − | 22 28 1 0 0 0 0 | + | 22 28 1 0 0 0 0 |
| − | 23 29 1 0 0 0 0 | + | 23 29 1 0 0 0 0 |
| − | 24 2 1 0 0 0 0 | + | 24 2 1 0 0 0 0 |
| − | 30 31 1 1 0 0 0 | + | 30 31 1 1 0 0 0 |
| − | 31 32 1 1 0 0 0 | + | 31 32 1 1 0 0 0 |
| − | 33 32 1 1 0 0 0 | + | 33 32 1 1 0 0 0 |
| − | 33 34 1 0 0 0 0 | + | 33 34 1 0 0 0 0 |
| − | 34 35 1 0 0 0 0 | + | 34 35 1 0 0 0 0 |
| − | 35 30 1 0 0 0 0 | + | 35 30 1 0 0 0 0 |
| − | 30 36 1 0 0 0 0 | + | 30 36 1 0 0 0 0 |
| − | 31 37 1 0 0 0 0 | + | 31 37 1 0 0 0 0 |
| − | 32 38 1 0 0 0 0 | + | 32 38 1 0 0 0 0 |
| − | 33 20 1 0 0 0 0 | + | 33 20 1 0 0 0 0 |
| − | 29 39 1 0 0 0 0 | + | 29 39 1 0 0 0 0 |
| − | 39 40 2 0 0 0 0 | + | 39 40 2 0 0 0 0 |
| − | 39 41 1 0 0 0 0 | + | 39 41 1 0 0 0 0 |
| − | 41 42 2 0 0 0 0 | + | 41 42 2 0 0 0 0 |
| − | 42 43 1 0 0 0 0 | + | 42 43 1 0 0 0 0 |
| − | 43 44 2 0 0 0 0 | + | 43 44 2 0 0 0 0 |
| − | 44 45 1 0 0 0 0 | + | 44 45 1 0 0 0 0 |
| − | 45 46 2 0 0 0 0 | + | 45 46 2 0 0 0 0 |
| − | 46 47 1 0 0 0 0 | + | 46 47 1 0 0 0 0 |
| − | 47 48 2 0 0 0 0 | + | 47 48 2 0 0 0 0 |
| − | 48 43 1 0 0 0 0 | + | 48 43 1 0 0 0 0 |
| − | 45 49 1 0 0 0 0 | + | 45 49 1 0 0 0 0 |
| − | 50 46 1 0 0 0 0 | + | 50 46 1 0 0 0 0 |
| − | 16 51 1 0 0 0 0 | + | 16 51 1 0 0 0 0 |
| − | 52 53 1 0 0 0 0 | + | 52 53 1 0 0 0 0 |
| − | 53 54 1 1 0 0 0 | + | 53 54 1 1 0 0 0 |
| − | 54 55 1 1 0 0 0 | + | 54 55 1 1 0 0 0 |
| − | 56 55 1 1 0 0 0 | + | 56 55 1 1 0 0 0 |
| − | 56 57 1 0 0 0 0 | + | 56 57 1 0 0 0 0 |
| − | 57 52 1 0 0 0 0 | + | 57 52 1 0 0 0 0 |
| − | 52 58 1 0 0 0 0 | + | 52 58 1 0 0 0 0 |
| − | 57 59 1 0 0 0 0 | + | 57 59 1 0 0 0 0 |
| − | 56 60 1 0 0 0 0 | + | 56 60 1 0 0 0 0 |
| − | 53 50 1 0 0 0 0 | + | 53 50 1 0 0 0 0 |
| − | 61 62 1 0 0 0 0 | + | 61 62 1 0 0 0 0 |
| − | 26 61 1 0 0 0 0 | + | 26 61 1 0 0 0 0 |
| − | 63 64 1 0 0 0 0 | + | 63 64 1 0 0 0 0 |
| − | 55 63 1 0 0 0 0 | + | 55 63 1 0 0 0 0 |
| − | 65 66 1 0 0 0 0 | + | 65 66 1 0 0 0 0 |
| − | 35 65 1 0 0 0 0 | + | 35 65 1 0 0 0 0 |
| − | M STY 1 1 SUP | + | M STY 1 1 SUP |
| − | M SLB 1 1 1 | + | M SLB 1 1 1 |
| − | M SAL 1 2 61 62 | + | M SAL 1 2 61 62 |
| − | M SBL 1 1 68 | + | M SBL 1 1 68 |
| − | M SMT 1 ^ CH2OH | + | M SMT 1 ^ CH2OH |
| − | M SBV 1 68 0.5619 -0.5619 | + | M SBV 1 68 0.5619 -0.5619 |
| − | M STY 1 2 SUP | + | M STY 1 2 SUP |
| − | M SLB 1 2 2 | + | M SLB 1 2 2 |
| − | M SAL 2 2 63 64 | + | M SAL 2 2 63 64 |
| − | M SBL 2 1 70 | + | M SBL 2 1 70 |
| − | M SMT 2 CH2OH | + | M SMT 2 CH2OH |
| − | M SBV 2 70 -0.7095 0.0000 | + | M SBV 2 70 -0.7095 0.0000 |
| − | M STY 1 3 SUP | + | M STY 1 3 SUP |
| − | M SLB 1 3 3 | + | M SLB 1 3 3 |
| − | M SAL 3 2 65 66 | + | M SAL 3 2 65 66 |
| − | M SBL 3 1 72 | + | M SBL 3 1 72 |
| − | M SMT 3 CH2OH | + | M SMT 3 CH2OH |
| − | M SBV 3 72 -0.4306 -0.4306 | + | M SBV 3 72 -0.4306 -0.4306 |
| − | S SKP 5 | + | S SKP 5 |
| − | ID FL3FACDS0019 | + | ID FL3FACDS0019 |
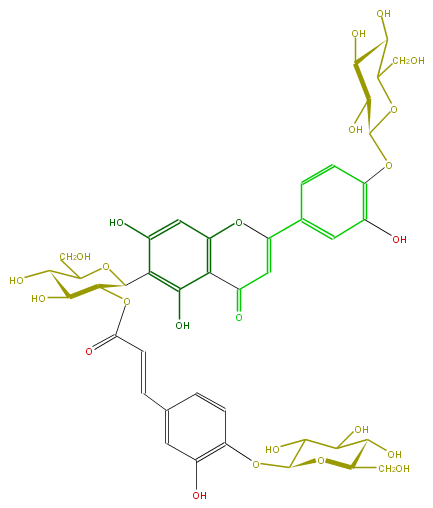
| − | FORMULA C42H46O24 | + | FORMULA C42H46O24 |
| − | EXACTMASS 934.2379024 | + | EXACTMASS 934.2379024 |
| − | AVERAGEMASS 934.8002399999999 | + | AVERAGEMASS 934.8002399999999 |
| − | SMILES c(c47)(c(c(O)cc4OC(=CC7=O)c(c5)ccc(OC(O6)C(C(O)C(O)C6CO)O)c5O)C(O1)C(OC(=O)C=Cc(c3)ccc(c3O)OC(O2)C(O)C(C(O)C2CO)O)C(O)C(O)C1CO)O | + | SMILES c(c47)(c(c(O)cc4OC(=CC7=O)c(c5)ccc(OC(O6)C(C(O)C(O)C6CO)O)c5O)C(O1)C(OC(=O)C=Cc(c3)ccc(c3O)OC(O2)C(O)C(C(O)C2CO)O)C(O)C(O)C1CO)O |
M END | M END | ||
| − | |||
Latest revision as of 09:00, 14 March 2009
Copyright: ARM project http://www.metabolome.jp/
66 72 0 0 0 0 0 0 0 0999 V2000
-1.7407 0.5702 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.7407 -0.2548 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.0261 -0.6673 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.3116 -0.2548 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.3116 0.5702 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.0261 0.9828 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.4030 -0.6673 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.1175 -0.2548 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.1175 0.5702 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.4030 0.9828 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
0.4030 -1.3107 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-2.4549 0.9827 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-1.0261 -1.4921 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
1.9113 1.0091 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.6642 0.5745 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.4172 1.0091 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.4172 1.8786 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.6642 2.3134 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.9113 1.8786 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.9971 2.3334 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-4.1371 -0.2134 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-3.6603 -0.8427 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.9737 -0.5757 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.3114 -0.5685 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.7926 -0.0871 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-3.3932 -0.4041 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-4.8523 -0.4050 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-4.3160 -0.8427 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-2.2729 -0.9254 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
3.9630 5.3431 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.1851 4.7539 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.5152 3.9053 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.5240 3.0865 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
4.1192 3.6815 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
3.7273 4.4239 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.7894 5.9907 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
3.1851 5.5104 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
3.1091 3.2018 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-2.5598 -1.7589 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-3.1968 -2.1266 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-1.8869 -2.1473 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.8869 -3.1623 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.3279 -3.5515 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.3279 -4.3592 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.6284 -4.7632 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.0712 -4.3592 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.0712 -3.5515 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.6284 -3.1476 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.6284 -5.5707 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
0.7977 -4.8193 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
4.1697 0.5747 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
2.0022 -4.2619 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.6157 -4.9312 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.3588 -4.7188 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
3.1063 -4.9312 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.4928 -4.2619 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.7498 -4.4742 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.2846 -4.4542 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
3.2423 -3.9816 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
4.0503 -4.5839 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-3.9552 0.1578 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-5.1814 0.5402 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
3.8158 -4.9312 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.8158 -5.9907 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
4.1579 4.8545 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
5.1814 4.5802 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
1 2 1 0 0 0 0
2 3 2 0 0 0 0
3 4 1 0 0 0 0
4 5 2 0 0 0 0
5 6 1 0 0 0 0
6 1 2 0 0 0 0
4 7 1 0 0 0 0
7 8 1 0 0 0 0
8 9 2 0 0 0 0
9 10 1 0 0 0 0
10 5 1 0 0 0 0
7 11 2 0 0 0 0
1 12 1 0 0 0 0
3 13 1 0 0 0 0
14 15 2 0 0 0 0
15 16 1 0 0 0 0
16 17 2 0 0 0 0
17 18 1 0 0 0 0
18 19 2 0 0 0 0
19 14 1 0 0 0 0
14 9 1 0 0 0 0
17 20 1 0 0 0 0
21 22 1 1 0 0 0
22 23 1 1 0 0 0
24 23 1 1 0 0 0
24 25 1 0 0 0 0
25 26 1 0 0 0 0
26 21 1 0 0 0 0
21 27 1 0 0 0 0
22 28 1 0 0 0 0
23 29 1 0 0 0 0
24 2 1 0 0 0 0
30 31 1 1 0 0 0
31 32 1 1 0 0 0
33 32 1 1 0 0 0
33 34 1 0 0 0 0
34 35 1 0 0 0 0
35 30 1 0 0 0 0
30 36 1 0 0 0 0
31 37 1 0 0 0 0
32 38 1 0 0 0 0
33 20 1 0 0 0 0
29 39 1 0 0 0 0
39 40 2 0 0 0 0
39 41 1 0 0 0 0
41 42 2 0 0 0 0
42 43 1 0 0 0 0
43 44 2 0 0 0 0
44 45 1 0 0 0 0
45 46 2 0 0 0 0
46 47 1 0 0 0 0
47 48 2 0 0 0 0
48 43 1 0 0 0 0
45 49 1 0 0 0 0
50 46 1 0 0 0 0
16 51 1 0 0 0 0
52 53 1 0 0 0 0
53 54 1 1 0 0 0
54 55 1 1 0 0 0
56 55 1 1 0 0 0
56 57 1 0 0 0 0
57 52 1 0 0 0 0
52 58 1 0 0 0 0
57 59 1 0 0 0 0
56 60 1 0 0 0 0
53 50 1 0 0 0 0
61 62 1 0 0 0 0
26 61 1 0 0 0 0
63 64 1 0 0 0 0
55 63 1 0 0 0 0
65 66 1 0 0 0 0
35 65 1 0 0 0 0
M STY 1 1 SUP
M SLB 1 1 1
M SAL 1 2 61 62
M SBL 1 1 68
M SMT 1 ^ CH2OH
M SBV 1 68 0.5619 -0.5619
M STY 1 2 SUP
M SLB 1 2 2
M SAL 2 2 63 64
M SBL 2 1 70
M SMT 2 CH2OH
M SBV 2 70 -0.7095 0.0000
M STY 1 3 SUP
M SLB 1 3 3
M SAL 3 2 65 66
M SBL 3 1 72
M SMT 3 CH2OH
M SBV 3 72 -0.4306 -0.4306
S SKP 5
ID FL3FACDS0019
FORMULA C42H46O24
EXACTMASS 934.2379024
AVERAGEMASS 934.8002399999999
SMILES c(c47)(c(c(O)cc4OC(=CC7=O)c(c5)ccc(OC(O6)C(C(O)C(O)C6CO)O)c5O)C(O1)C(OC(=O)C=Cc(c3)ccc(c3O)OC(O2)C(O)C(C(O)C2CO)O)C(O)C(O)C1CO)O
M END