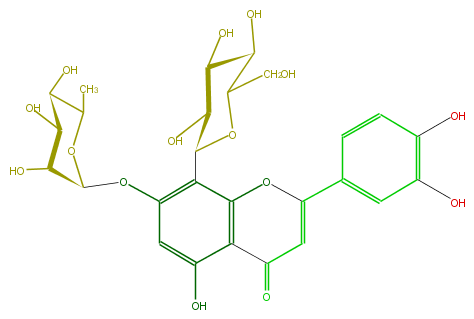
Mol:FL3FACDS0004
From Metabolomics.JP
(Difference between revisions)
| Line 1: | Line 1: | ||
| − | + | ||
| − | + | ||
| − | Copyright: ARM project http://www.metabolome.jp/ | + | Copyright: ARM project http://www.metabolome.jp/ |
| − | 42 46 0 0 0 0 0 0 0 0999 V2000 | + | 42 46 0 0 0 0 0 0 0 0999 V2000 |
| − | -1.5659 -0.8577 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.5659 -0.8577 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.5659 -1.6827 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.5659 -1.6827 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.8514 -2.0951 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.8514 -2.0951 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.1370 -1.6827 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.1370 -1.6827 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.1370 -0.8577 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.1370 -0.8577 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.8514 -0.4452 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.8514 -0.4452 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.5775 -2.0951 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.5775 -2.0951 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.2919 -1.6827 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.2919 -1.6827 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.2919 -0.8577 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.2919 -0.8577 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.5775 -0.4452 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.5775 -0.4452 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.5775 -2.7384 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.5775 -2.7384 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.2801 -0.4453 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.2801 -0.4453 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.0856 -0.4188 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.0856 -0.4188 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.8385 -0.8536 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.8385 -0.8536 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.5914 -0.4188 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.5914 -0.4188 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.5914 0.4505 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.5914 0.4505 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.8385 0.8851 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.8385 0.8851 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.0856 0.4505 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.0856 0.4505 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 4.3438 0.8849 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 4.3438 0.8849 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.3269 2.1214 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.3269 2.1214 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.6055 1.8337 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.6055 1.8337 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.5856 0.9236 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.5856 0.9236 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.8573 0.1512 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.8573 0.1512 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.0947 0.5067 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.0947 0.5067 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.2089 1.3382 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.2089 1.3382 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.2539 2.9199 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.2539 2.9199 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.3113 2.5457 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.3113 2.5457 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.3318 0.3732 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.3318 0.3732 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 4.3438 -0.8533 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 4.3438 -0.8533 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.8514 -2.9199 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.8514 -2.9199 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.7754 -0.1877 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.7754 -0.1877 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.1008 -0.5771 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.1008 -0.5771 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.3149 0.1718 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.3149 0.1718 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.1008 0.9253 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.1008 0.9253 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.7754 1.3148 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.7754 1.3148 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.5614 0.5658 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.5614 0.5658 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.4262 1.8055 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.4262 1.8055 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.1008 1.4113 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.1008 1.4113 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -4.1667 1.0364 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -4.1667 1.0364 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -4.3438 -0.2180 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -4.3438 -0.2180 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.5863 1.7171 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.5863 1.7171 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.2674 0.9054 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.2674 0.9054 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1 2 1 0 0 0 0 | + | 1 2 1 0 0 0 0 |
| − | 2 3 2 0 0 0 0 | + | 2 3 2 0 0 0 0 |
| − | 3 4 1 0 0 0 0 | + | 3 4 1 0 0 0 0 |
| − | 4 5 2 0 0 0 0 | + | 4 5 2 0 0 0 0 |
| − | 5 6 1 0 0 0 0 | + | 5 6 1 0 0 0 0 |
| − | 6 1 2 0 0 0 0 | + | 6 1 2 0 0 0 0 |
| − | 4 7 1 0 0 0 0 | + | 4 7 1 0 0 0 0 |
| − | 7 8 1 0 0 0 0 | + | 7 8 1 0 0 0 0 |
| − | 8 9 2 0 0 0 0 | + | 8 9 2 0 0 0 0 |
| − | 9 10 1 0 0 0 0 | + | 9 10 1 0 0 0 0 |
| − | 10 5 1 0 0 0 0 | + | 10 5 1 0 0 0 0 |
| − | 7 11 2 0 0 0 0 | + | 7 11 2 0 0 0 0 |
| − | 1 12 1 0 0 0 0 | + | 1 12 1 0 0 0 0 |
| − | 13 14 2 0 0 0 0 | + | 13 14 2 0 0 0 0 |
| − | 14 15 1 0 0 0 0 | + | 14 15 1 0 0 0 0 |
| − | 15 16 2 0 0 0 0 | + | 15 16 2 0 0 0 0 |
| − | 16 17 1 0 0 0 0 | + | 16 17 1 0 0 0 0 |
| − | 17 18 2 0 0 0 0 | + | 17 18 2 0 0 0 0 |
| − | 18 13 1 0 0 0 0 | + | 18 13 1 0 0 0 0 |
| − | 13 9 1 0 0 0 0 | + | 13 9 1 0 0 0 0 |
| − | 16 19 1 0 0 0 0 | + | 16 19 1 0 0 0 0 |
| − | 20 21 1 1 0 0 0 | + | 20 21 1 1 0 0 0 |
| − | 21 22 1 1 0 0 0 | + | 21 22 1 1 0 0 0 |
| − | 23 22 1 1 0 0 0 | + | 23 22 1 1 0 0 0 |
| − | 23 24 1 0 0 0 0 | + | 23 24 1 0 0 0 0 |
| − | 24 25 1 0 0 0 0 | + | 24 25 1 0 0 0 0 |
| − | 25 20 1 0 0 0 0 | + | 25 20 1 0 0 0 0 |
| − | 20 26 1 0 0 0 0 | + | 20 26 1 0 0 0 0 |
| − | 21 27 1 0 0 0 0 | + | 21 27 1 0 0 0 0 |
| − | 22 28 1 0 0 0 0 | + | 22 28 1 0 0 0 0 |
| − | 6 23 1 0 0 0 0 | + | 6 23 1 0 0 0 0 |
| − | 15 29 1 0 0 0 0 | + | 15 29 1 0 0 0 0 |
| − | 3 30 1 0 0 0 0 | + | 3 30 1 0 0 0 0 |
| − | 32 31 1 1 0 0 0 | + | 32 31 1 1 0 0 0 |
| − | 32 33 1 0 0 0 0 | + | 32 33 1 0 0 0 0 |
| − | 33 34 1 0 0 0 0 | + | 33 34 1 0 0 0 0 |
| − | 34 35 1 0 0 0 0 | + | 34 35 1 0 0 0 0 |
| − | 35 36 1 1 0 0 0 | + | 35 36 1 1 0 0 0 |
| − | 36 31 1 1 0 0 0 | + | 36 31 1 1 0 0 0 |
| − | 35 37 1 0 0 0 0 | + | 35 37 1 0 0 0 0 |
| − | 34 38 1 0 0 0 0 | + | 34 38 1 0 0 0 0 |
| − | 36 39 1 0 0 0 0 | + | 36 39 1 0 0 0 0 |
| − | 31 40 1 0 0 0 0 | + | 31 40 1 0 0 0 0 |
| − | 32 12 1 0 0 0 0 | + | 32 12 1 0 0 0 0 |
| − | 41 42 1 0 0 0 0 | + | 41 42 1 0 0 0 0 |
| − | 25 41 1 0 0 0 0 | + | 25 41 1 0 0 0 0 |
| − | M STY 1 1 SUP | + | M STY 1 1 SUP |
| − | M SLB 1 1 1 | + | M SLB 1 1 1 |
| − | M SAL 1 2 41 42 | + | M SAL 1 2 41 42 |
| − | M SBL 1 1 46 | + | M SBL 1 1 46 |
| − | M SMT 1 CH2OH | + | M SMT 1 CH2OH |
| − | M SBV 1 46 -0.7952 -0.3788 | + | M SBV 1 46 -0.7952 -0.3788 |
| − | S SKP 5 | + | S SKP 5 |
| − | ID FL3FACDS0004 | + | ID FL3FACDS0004 |
| − | FORMULA C27H30O15 | + | FORMULA C27H30O15 |
| − | EXACTMASS 594.15847029 | + | EXACTMASS 594.15847029 |
| − | AVERAGEMASS 594.5181 | + | AVERAGEMASS 594.5181 |
| − | SMILES c(O3)(c(C(C5O)OC(C(C5O)O)CO)1)c(C(C=C3c(c4)ccc(O)c4O)=O)c(cc(OC(O2)C(C(C(C2C)O)O)O)1)O | + | SMILES c(O3)(c(C(C5O)OC(C(C5O)O)CO)1)c(C(C=C3c(c4)ccc(O)c4O)=O)c(cc(OC(O2)C(C(C(C2C)O)O)O)1)O |
M END | M END | ||
| − | |||
Latest revision as of 09:00, 14 March 2009
Copyright: ARM project http://www.metabolome.jp/
42 46 0 0 0 0 0 0 0 0999 V2000
-1.5659 -0.8577 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.5659 -1.6827 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.8514 -2.0951 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.1370 -1.6827 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.1370 -0.8577 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.8514 -0.4452 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.5775 -2.0951 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.2919 -1.6827 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.2919 -0.8577 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.5775 -0.4452 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
0.5775 -2.7384 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-2.2801 -0.4453 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
2.0856 -0.4188 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.8385 -0.8536 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.5914 -0.4188 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.5914 0.4505 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.8385 0.8851 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.0856 0.4505 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
4.3438 0.8849 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
0.3269 2.1214 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.6055 1.8337 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.5856 0.9236 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.8573 0.1512 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.0947 0.5067 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-0.2089 1.3382 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.2539 2.9199 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-0.3113 2.5457 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-1.3318 0.3732 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
4.3438 -0.8533 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-0.8514 -2.9199 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-3.7754 -0.1877 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-3.1008 -0.5771 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-3.3149 0.1718 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-3.1008 0.9253 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-3.7754 1.3148 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-3.5614 0.5658 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-3.4262 1.8055 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-3.1008 1.4113 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-4.1667 1.0364 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-4.3438 -0.2180 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
0.5863 1.7171 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.2674 0.9054 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
1 2 1 0 0 0 0
2 3 2 0 0 0 0
3 4 1 0 0 0 0
4 5 2 0 0 0 0
5 6 1 0 0 0 0
6 1 2 0 0 0 0
4 7 1 0 0 0 0
7 8 1 0 0 0 0
8 9 2 0 0 0 0
9 10 1 0 0 0 0
10 5 1 0 0 0 0
7 11 2 0 0 0 0
1 12 1 0 0 0 0
13 14 2 0 0 0 0
14 15 1 0 0 0 0
15 16 2 0 0 0 0
16 17 1 0 0 0 0
17 18 2 0 0 0 0
18 13 1 0 0 0 0
13 9 1 0 0 0 0
16 19 1 0 0 0 0
20 21 1 1 0 0 0
21 22 1 1 0 0 0
23 22 1 1 0 0 0
23 24 1 0 0 0 0
24 25 1 0 0 0 0
25 20 1 0 0 0 0
20 26 1 0 0 0 0
21 27 1 0 0 0 0
22 28 1 0 0 0 0
6 23 1 0 0 0 0
15 29 1 0 0 0 0
3 30 1 0 0 0 0
32 31 1 1 0 0 0
32 33 1 0 0 0 0
33 34 1 0 0 0 0
34 35 1 0 0 0 0
35 36 1 1 0 0 0
36 31 1 1 0 0 0
35 37 1 0 0 0 0
34 38 1 0 0 0 0
36 39 1 0 0 0 0
31 40 1 0 0 0 0
32 12 1 0 0 0 0
41 42 1 0 0 0 0
25 41 1 0 0 0 0
M STY 1 1 SUP
M SLB 1 1 1
M SAL 1 2 41 42
M SBL 1 1 46
M SMT 1 CH2OH
M SBV 1 46 -0.7952 -0.3788
S SKP 5
ID FL3FACDS0004
FORMULA C27H30O15
EXACTMASS 594.15847029
AVERAGEMASS 594.5181
SMILES c(O3)(c(C(C5O)OC(C(C5O)O)CO)1)c(C(C=C3c(c4)ccc(O)c4O)=O)c(cc(OC(O2)C(C(C(C2C)O)O)O)1)O
M END