Mol:FL3FACCS0057
From Metabolomics.JP
(Difference between revisions)
| Line 1: | Line 1: | ||
| − | + | ||
| − | + | ||
| − | Copyright: ARM project http://www.metabolome.jp/ | + | Copyright: ARM project http://www.metabolome.jp/ |
| − | 35 38 0 0 0 0 0 0 0 0999 V2000 | + | 35 38 0 0 0 0 0 0 0 0999 V2000 |
| − | 1.6838 0.1537 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.6838 0.1537 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.9693 0.5662 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.9693 0.5662 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.2549 0.1537 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.2549 0.1537 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.2549 -0.6713 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.2549 -0.6713 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.9693 -1.0838 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.9693 -1.0838 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.6838 -0.6713 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.6838 -0.6713 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.4596 0.5662 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.4596 0.5662 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.1741 0.1537 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.1741 0.1537 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.1741 -0.6713 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.1741 -0.6713 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.4596 -1.0838 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.4596 -1.0838 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.9693 -1.6559 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.9693 -1.6559 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.4852 0.6163 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.4852 0.6163 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.1997 0.2038 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.1997 0.2038 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.9141 0.6163 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.9141 0.6163 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.9141 1.4413 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.9141 1.4413 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.1997 1.8538 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.1997 1.8538 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.4852 1.4413 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.4852 1.4413 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 4.5976 1.8359 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 4.5976 1.8359 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.4596 -1.8538 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.4596 -1.8538 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.8645 0.5523 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.8645 0.5523 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 4.5757 0.2344 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 4.5757 0.2344 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -4.0273 -0.3079 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -4.0273 -0.3079 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.6147 -1.0226 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.6147 -1.0226 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.8164 -0.8134 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.8164 -0.8134 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.0229 -1.0403 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.0229 -1.0403 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.4355 -0.3255 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.4355 -0.3255 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.2339 -0.5346 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.2339 -0.5346 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.3908 0.0512 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.3908 0.0512 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -4.5106 -0.7911 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -4.5106 -0.7911 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -4.0634 -0.7635 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -4.0634 -0.7635 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.0347 -1.5613 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.0347 -1.5613 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -4.0863 0.3917 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -4.0863 0.3917 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -4.0905 1.0340 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -4.0905 1.0340 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -4.5976 1.3268 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -4.5976 1.3268 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.5847 1.3261 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.5847 1.3261 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1 2 1 0 0 0 0 | + | 1 2 1 0 0 0 0 |
| − | 2 3 1 0 0 0 0 | + | 2 3 1 0 0 0 0 |
| − | 3 4 2 0 0 0 0 | + | 3 4 2 0 0 0 0 |
| − | 4 5 1 0 0 0 0 | + | 4 5 1 0 0 0 0 |
| − | 5 6 1 0 0 0 0 | + | 5 6 1 0 0 0 0 |
| − | 6 1 2 0 0 0 0 | + | 6 1 2 0 0 0 0 |
| − | 3 7 1 0 0 0 0 | + | 3 7 1 0 0 0 0 |
| − | 7 8 2 0 0 0 0 | + | 7 8 2 0 0 0 0 |
| − | 8 9 1 0 0 0 0 | + | 8 9 1 0 0 0 0 |
| − | 9 10 2 0 0 0 0 | + | 9 10 2 0 0 0 0 |
| − | 10 4 1 0 0 0 0 | + | 10 4 1 0 0 0 0 |
| − | 5 11 2 0 0 0 0 | + | 5 11 2 0 0 0 0 |
| − | 1 12 1 0 0 0 0 | + | 1 12 1 0 0 0 0 |
| − | 12 13 2 0 0 0 0 | + | 12 13 2 0 0 0 0 |
| − | 13 14 1 0 0 0 0 | + | 13 14 1 0 0 0 0 |
| − | 14 15 2 0 0 0 0 | + | 14 15 2 0 0 0 0 |
| − | 15 16 1 0 0 0 0 | + | 15 16 1 0 0 0 0 |
| − | 16 17 2 0 0 0 0 | + | 16 17 2 0 0 0 0 |
| − | 17 12 1 0 0 0 0 | + | 17 12 1 0 0 0 0 |
| − | 15 18 1 0 0 0 0 | + | 15 18 1 0 0 0 0 |
| − | 10 19 1 0 0 0 0 | + | 10 19 1 0 0 0 0 |
| − | 8 20 1 0 0 0 0 | + | 8 20 1 0 0 0 0 |
| − | 14 21 1 0 0 0 0 | + | 14 21 1 0 0 0 0 |
| − | 22 23 1 1 0 0 0 | + | 22 23 1 1 0 0 0 |
| − | 23 24 1 1 0 0 0 | + | 23 24 1 1 0 0 0 |
| − | 25 24 1 1 0 0 0 | + | 25 24 1 1 0 0 0 |
| − | 25 26 1 0 0 0 0 | + | 25 26 1 0 0 0 0 |
| − | 26 27 1 0 0 0 0 | + | 26 27 1 0 0 0 0 |
| − | 27 22 1 0 0 0 0 | + | 27 22 1 0 0 0 0 |
| − | 27 28 1 0 0 0 0 | + | 27 28 1 0 0 0 0 |
| − | 22 29 1 0 0 0 0 | + | 22 29 1 0 0 0 0 |
| − | 23 30 1 0 0 0 0 | + | 23 30 1 0 0 0 0 |
| − | 25 9 1 0 0 0 0 | + | 25 9 1 0 0 0 0 |
| − | 31 24 1 0 0 0 0 | + | 31 24 1 0 0 0 0 |
| − | 32 33 1 0 0 0 0 | + | 32 33 1 0 0 0 0 |
| − | 32 28 1 0 0 0 0 | + | 32 28 1 0 0 0 0 |
| − | 33 34 2 0 0 0 0 | + | 33 34 2 0 0 0 0 |
| − | 33 35 1 0 0 0 0 | + | 33 35 1 0 0 0 0 |
| − | S SKP 8 | + | S SKP 8 |
| − | ID FL3FACCS0057 | + | ID FL3FACCS0057 |
| − | KNApSAcK_ID C00014099 | + | KNApSAcK_ID C00014099 |
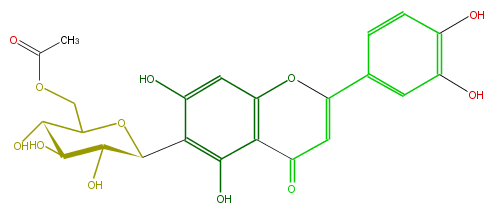
| − | NAME Isoorientin 6''-O-acetate | + | NAME Isoorientin 6''-O-acetate |
| − | CAS_RN - | + | CAS_RN - |
| − | FORMULA C23H22O12 | + | FORMULA C23H22O12 |
| − | EXACTMASS 490.111126168 | + | EXACTMASS 490.111126168 |
| − | AVERAGEMASS 490.41358 | + | AVERAGEMASS 490.41358 |
| − | SMILES C(C(=O)3)=C(c(c4)ccc(c4O)O)Oc(c31)cc(c(C(C(O)2)OC(COC(C)=O)C(C(O)2)O)c(O)1)O | + | SMILES C(C(=O)3)=C(c(c4)ccc(c4O)O)Oc(c31)cc(c(C(C(O)2)OC(COC(C)=O)C(C(O)2)O)c(O)1)O |
M END | M END | ||
| − | |||
Latest revision as of 09:00, 14 March 2009
Copyright: ARM project http://www.metabolome.jp/
35 38 0 0 0 0 0 0 0 0999 V2000
1.6838 0.1537 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.9693 0.5662 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
0.2549 0.1537 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.2549 -0.6713 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.9693 -1.0838 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.6838 -0.6713 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.4596 0.5662 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.1741 0.1537 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.1741 -0.6713 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.4596 -1.0838 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.9693 -1.6559 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
2.4852 0.6163 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.1997 0.2038 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.9141 0.6163 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.9141 1.4413 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.1997 1.8538 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.4852 1.4413 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
4.5976 1.8359 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-0.4596 -1.8538 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-1.8645 0.5523 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
4.5757 0.2344 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-4.0273 -0.3079 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-3.6147 -1.0226 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.8164 -0.8134 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.0229 -1.0403 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.4355 -0.3255 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-3.2339 -0.5346 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-3.3908 0.0512 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-4.5106 -0.7911 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-4.0634 -0.7635 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-3.0347 -1.5613 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-4.0863 0.3917 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-4.0905 1.0340 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-4.5976 1.3268 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-3.5847 1.3261 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1 2 1 0 0 0 0
2 3 1 0 0 0 0
3 4 2 0 0 0 0
4 5 1 0 0 0 0
5 6 1 0 0 0 0
6 1 2 0 0 0 0
3 7 1 0 0 0 0
7 8 2 0 0 0 0
8 9 1 0 0 0 0
9 10 2 0 0 0 0
10 4 1 0 0 0 0
5 11 2 0 0 0 0
1 12 1 0 0 0 0
12 13 2 0 0 0 0
13 14 1 0 0 0 0
14 15 2 0 0 0 0
15 16 1 0 0 0 0
16 17 2 0 0 0 0
17 12 1 0 0 0 0
15 18 1 0 0 0 0
10 19 1 0 0 0 0
8 20 1 0 0 0 0
14 21 1 0 0 0 0
22 23 1 1 0 0 0
23 24 1 1 0 0 0
25 24 1 1 0 0 0
25 26 1 0 0 0 0
26 27 1 0 0 0 0
27 22 1 0 0 0 0
27 28 1 0 0 0 0
22 29 1 0 0 0 0
23 30 1 0 0 0 0
25 9 1 0 0 0 0
31 24 1 0 0 0 0
32 33 1 0 0 0 0
32 28 1 0 0 0 0
33 34 2 0 0 0 0
33 35 1 0 0 0 0
S SKP 8
ID FL3FACCS0057
KNApSAcK_ID C00014099
NAME Isoorientin 6''-O-acetate
CAS_RN -
FORMULA C23H22O12
EXACTMASS 490.111126168
AVERAGEMASS 490.41358
SMILES C(C(=O)3)=C(c(c4)ccc(c4O)O)Oc(c31)cc(c(C(C(O)2)OC(COC(C)=O)C(C(O)2)O)c(O)1)O
M END