Mol:FL3FACCS0002
From Metabolomics.JP
(Difference between revisions)
| Line 1: | Line 1: | ||
| − | + | ||
| − | + | ||
| − | Copyright: ARM project http://www.metabolome.jp/ | + | Copyright: ARM project http://www.metabolome.jp/ |
| − | 32 35 0 0 0 0 0 0 0 0999 V2000 | + | 32 35 0 0 0 0 0 0 0 0999 V2000 |
| − | -1.8333 -0.8365 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.8333 -0.8365 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.8333 -1.4144 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.8333 -1.4144 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.3328 -1.7033 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.3328 -1.7033 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.8324 -1.4144 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.8324 -1.4144 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.8324 -0.8365 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.8324 -0.8365 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.3328 -0.5476 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.3328 -0.5476 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.3319 -1.7033 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.3319 -1.7033 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.1685 -1.4144 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.1685 -1.4144 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.1685 -0.8365 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.1685 -0.8365 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.3319 -0.5476 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.3319 -0.5476 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.6686 -0.5478 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.6686 -0.5478 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.1990 -0.8540 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.1990 -0.8540 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.7293 -0.5478 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.7293 -0.5478 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.7293 0.0646 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.7293 0.0646 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.1990 0.3708 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.1990 0.3708 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.6686 0.0646 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.6686 0.0646 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.3328 -2.2808 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.3328 -2.2808 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.2592 -0.4106 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.2592 -0.4106 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.3319 -2.2805 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.3319 -2.2805 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.2592 0.3706 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.2592 0.3706 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.3383 0.2154 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.3383 0.2154 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.6568 0.3980 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.6568 0.3980 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.6568 1.1035 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.6568 1.1035 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.2939 1.7085 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.2939 1.7085 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.9754 1.5259 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.9754 1.5259 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.9754 0.8204 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.9754 0.8204 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.0288 1.2717 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.0288 1.2717 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.4119 2.1487 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.4119 2.1487 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.7896 -0.0975 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.7896 -0.0975 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.2592 -0.8537 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.2592 -0.8537 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.6417 1.6974 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.6417 1.6974 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.2251 2.2808 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.2251 2.2808 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1 2 1 0 0 0 0 | + | 1 2 1 0 0 0 0 |
| − | 2 3 2 0 0 0 0 | + | 2 3 2 0 0 0 0 |
| − | 3 4 1 0 0 0 0 | + | 3 4 1 0 0 0 0 |
| − | 4 5 2 0 0 0 0 | + | 4 5 2 0 0 0 0 |
| − | 5 6 1 0 0 0 0 | + | 5 6 1 0 0 0 0 |
| − | 6 1 2 0 0 0 0 | + | 6 1 2 0 0 0 0 |
| − | 4 7 1 0 0 0 0 | + | 4 7 1 0 0 0 0 |
| − | 7 8 1 0 0 0 0 | + | 7 8 1 0 0 0 0 |
| − | 8 9 2 0 0 0 0 | + | 8 9 2 0 0 0 0 |
| − | 9 10 1 0 0 0 0 | + | 9 10 1 0 0 0 0 |
| − | 10 5 1 0 0 0 0 | + | 10 5 1 0 0 0 0 |
| − | 9 11 1 0 0 0 0 | + | 9 11 1 0 0 0 0 |
| − | 11 12 2 0 0 0 0 | + | 11 12 2 0 0 0 0 |
| − | 12 13 1 0 0 0 0 | + | 12 13 1 0 0 0 0 |
| − | 13 14 2 0 0 0 0 | + | 13 14 2 0 0 0 0 |
| − | 14 15 1 0 0 0 0 | + | 14 15 1 0 0 0 0 |
| − | 15 16 2 0 0 0 0 | + | 15 16 2 0 0 0 0 |
| − | 16 11 1 0 0 0 0 | + | 16 11 1 0 0 0 0 |
| − | 3 17 1 0 0 0 0 | + | 3 17 1 0 0 0 0 |
| − | 1 18 1 0 0 0 0 | + | 1 18 1 0 0 0 0 |
| − | 7 19 2 0 0 0 0 | + | 7 19 2 0 0 0 0 |
| − | 14 20 1 0 0 0 0 | + | 14 20 1 0 0 0 0 |
| − | 21 22 1 1 0 0 0 | + | 21 22 1 1 0 0 0 |
| − | 22 23 1 1 0 0 0 | + | 22 23 1 1 0 0 0 |
| − | 24 23 1 1 0 0 0 | + | 24 23 1 1 0 0 0 |
| − | 24 25 1 0 0 0 0 | + | 24 25 1 0 0 0 0 |
| − | 25 26 1 0 0 0 0 | + | 25 26 1 0 0 0 0 |
| − | 26 21 1 0 0 0 0 | + | 26 21 1 0 0 0 0 |
| − | 6 21 1 0 0 0 0 | + | 6 21 1 0 0 0 0 |
| − | 23 27 1 0 0 0 0 | + | 23 27 1 0 0 0 0 |
| − | 24 28 1 0 0 0 0 | + | 24 28 1 0 0 0 0 |
| − | 22 29 1 0 0 0 0 | + | 22 29 1 0 0 0 0 |
| − | 13 30 1 0 0 0 0 | + | 13 30 1 0 0 0 0 |
| − | 25 31 1 0 0 0 0 | + | 25 31 1 0 0 0 0 |
| − | 31 32 1 0 0 0 0 | + | 31 32 1 0 0 0 0 |
| − | M STY 1 1 SUP | + | M STY 1 1 SUP |
| − | M SLB 1 1 1 | + | M SLB 1 1 1 |
| − | M SAL 1 2 31 32 | + | M SAL 1 2 31 32 |
| − | M SBL 1 1 34 | + | M SBL 1 1 34 |
| − | M SMT 1 CH2OH | + | M SMT 1 CH2OH |
| − | M SBV 1 34 -8.3120 3.3569 | + | M SBV 1 34 -8.3120 3.3569 |
| − | S SKP 8 | + | S SKP 8 |
| − | ID FL3FACCS0002 | + | ID FL3FACCS0002 |
| − | KNApSAcK_ID C00001078 | + | KNApSAcK_ID C00001078 |
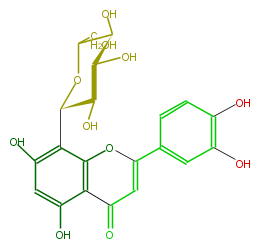
| − | NAME Orientin;8-beta-D-Glucopyranosyl-3',4',5,7-tetrahydroxyflavone;Luteolin 8-C-beta-D-glucopyranoside;Lutexin;2-(3,4-Dihydroxyphenyl)-8-beta-D-glucopyranosyl-5,7-dihydroxy-4H-1-benzopyran-4-one | + | NAME Orientin;8-beta-D-Glucopyranosyl-3',4',5,7-tetrahydroxyflavone;Luteolin 8-C-beta-D-glucopyranoside;Lutexin;2-(3,4-Dihydroxyphenyl)-8-beta-D-glucopyranosyl-5,7-dihydroxy-4H-1-benzopyran-4-one |
| − | CAS_RN 28608-75-5 | + | CAS_RN 28608-75-5 |
| − | FORMULA C21H20O11 | + | FORMULA C21H20O11 |
| − | EXACTMASS 448.100561482 | + | EXACTMASS 448.100561482 |
| − | AVERAGEMASS 448.3769 | + | AVERAGEMASS 448.3769 |
| − | SMILES C(C(O)1)(c(c(O)4)c(O2)c(c(O)c4)C(=O)C=C2c(c3)cc(c(O)c3)O)OC(CO)C(O)C1O | + | SMILES C(C(O)1)(c(c(O)4)c(O2)c(c(O)c4)C(=O)C=C2c(c3)cc(c(O)c3)O)OC(CO)C(O)C1O |
M END | M END | ||
| − | |||
Latest revision as of 09:00, 14 March 2009
Copyright: ARM project http://www.metabolome.jp/
32 35 0 0 0 0 0 0 0 0999 V2000
-1.8333 -0.8365 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.8333 -1.4144 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.3328 -1.7033 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.8324 -1.4144 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.8324 -0.8365 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.3328 -0.5476 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.3319 -1.7033 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.1685 -1.4144 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.1685 -0.8365 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.3319 -0.5476 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
0.6686 -0.5478 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.1990 -0.8540 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.7293 -0.5478 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.7293 0.0646 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.1990 0.3708 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.6686 0.0646 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.3328 -2.2808 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-2.2592 -0.4106 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-0.3319 -2.2805 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
2.2592 0.3706 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-1.3383 0.2154 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.6568 0.3980 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.6568 1.1035 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.2939 1.7085 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.9754 1.5259 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.9754 0.8204 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-0.0288 1.2717 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-0.4119 2.1487 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-0.7896 -0.0975 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
2.2592 -0.8537 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-0.6417 1.6974 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.2251 2.2808 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
1 2 1 0 0 0 0
2 3 2 0 0 0 0
3 4 1 0 0 0 0
4 5 2 0 0 0 0
5 6 1 0 0 0 0
6 1 2 0 0 0 0
4 7 1 0 0 0 0
7 8 1 0 0 0 0
8 9 2 0 0 0 0
9 10 1 0 0 0 0
10 5 1 0 0 0 0
9 11 1 0 0 0 0
11 12 2 0 0 0 0
12 13 1 0 0 0 0
13 14 2 0 0 0 0
14 15 1 0 0 0 0
15 16 2 0 0 0 0
16 11 1 0 0 0 0
3 17 1 0 0 0 0
1 18 1 0 0 0 0
7 19 2 0 0 0 0
14 20 1 0 0 0 0
21 22 1 1 0 0 0
22 23 1 1 0 0 0
24 23 1 1 0 0 0
24 25 1 0 0 0 0
25 26 1 0 0 0 0
26 21 1 0 0 0 0
6 21 1 0 0 0 0
23 27 1 0 0 0 0
24 28 1 0 0 0 0
22 29 1 0 0 0 0
13 30 1 0 0 0 0
25 31 1 0 0 0 0
31 32 1 0 0 0 0
M STY 1 1 SUP
M SLB 1 1 1
M SAL 1 2 31 32
M SBL 1 1 34
M SMT 1 CH2OH
M SBV 1 34 -8.3120 3.3569
S SKP 8
ID FL3FACCS0002
KNApSAcK_ID C00001078
NAME Orientin;8-beta-D-Glucopyranosyl-3',4',5,7-tetrahydroxyflavone;Luteolin 8-C-beta-D-glucopyranoside;Lutexin;2-(3,4-Dihydroxyphenyl)-8-beta-D-glucopyranosyl-5,7-dihydroxy-4H-1-benzopyran-4-one
CAS_RN 28608-75-5
FORMULA C21H20O11
EXACTMASS 448.100561482
AVERAGEMASS 448.3769
SMILES C(C(O)1)(c(c(O)4)c(O2)c(c(O)c4)C(=O)C=C2c(c3)cc(c(O)c3)O)OC(CO)C(O)C1O
M END