Mol:FL3FAAGS0058
From Metabolomics.JP
(Difference between revisions)
| Line 1: | Line 1: | ||
| − | + | ||
| − | + | ||
| − | Copyright: ARM project http://www.metabolome.jp/ | + | Copyright: ARM project http://www.metabolome.jp/ |
| − | 42 46 0 0 0 0 0 0 0 0999 V2000 | + | 42 46 0 0 0 0 0 0 0 0999 V2000 |
| − | 0.3853 0.1971 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.3853 0.1971 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.3853 -0.6279 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.3853 -0.6279 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.0998 -1.0404 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.0998 -1.0404 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.8142 -0.6279 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.8142 -0.6279 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.8142 0.1971 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.8142 0.1971 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.0998 0.6096 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.0998 0.6096 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.5287 -1.0404 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.5287 -1.0404 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.2432 -0.6279 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.2432 -0.6279 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.2432 0.1971 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.2432 0.1971 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.5287 0.6096 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.5287 0.6096 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.9576 0.6096 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.9576 0.6096 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 4.6721 0.1971 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 4.6721 0.1971 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 5.3866 0.6096 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 5.3866 0.6096 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 5.3866 1.4346 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 5.3866 1.4346 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 4.6721 1.8471 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 4.6721 1.8471 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.9576 1.4346 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.9576 1.4346 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.5287 -1.8654 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.5287 -1.8654 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.0998 -1.8654 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.0998 -1.8654 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.3292 0.6096 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.3292 0.6096 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 6.1329 1.8654 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 6.1329 1.8654 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.8503 0.5285 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.8503 0.5285 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.3440 -0.1232 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.3440 -0.1232 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.5817 0.1930 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.5817 0.1930 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.7646 0.0766 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.7646 0.0766 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.2709 0.7283 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.2709 0.7283 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.0333 0.4122 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.0333 0.4122 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.2537 0.9355 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.2537 0.9355 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.6794 1.2580 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.6794 1.2580 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.2280 0.0301 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.2280 0.0301 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.7392 -0.0736 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.7392 -0.0736 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.1445 -0.3697 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.1445 -0.3697 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -5.7552 -0.0327 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -5.7552 -0.0327 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -5.2489 -0.6844 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -5.2489 -0.6844 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -4.4866 -0.3682 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -4.4866 -0.3682 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.6695 -0.4846 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.6695 -0.4846 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -4.1758 0.1672 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -4.1758 0.1672 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -4.9382 -0.1490 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -4.9382 -0.1490 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -5.1587 0.3743 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -5.1587 0.3743 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -5.5844 0.6969 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -5.5844 0.6969 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -6.1329 -0.5311 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -6.1329 -0.5311 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -5.6442 -0.6348 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -5.6442 -0.6348 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -4.0494 -0.9309 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -4.0494 -0.9309 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1 2 2 0 0 0 0 | + | 1 2 2 0 0 0 0 |
| − | 2 3 1 0 0 0 0 | + | 2 3 1 0 0 0 0 |
| − | 3 4 2 0 0 0 0 | + | 3 4 2 0 0 0 0 |
| − | 4 5 1 0 0 0 0 | + | 4 5 1 0 0 0 0 |
| − | 5 6 2 0 0 0 0 | + | 5 6 2 0 0 0 0 |
| − | 6 1 1 0 0 0 0 | + | 6 1 1 0 0 0 0 |
| − | 4 7 1 0 0 0 0 | + | 4 7 1 0 0 0 0 |
| − | 7 8 1 0 0 0 0 | + | 7 8 1 0 0 0 0 |
| − | 8 9 2 0 0 0 0 | + | 8 9 2 0 0 0 0 |
| − | 9 10 1 0 0 0 0 | + | 9 10 1 0 0 0 0 |
| − | 10 5 1 0 0 0 0 | + | 10 5 1 0 0 0 0 |
| − | 9 11 1 0 0 0 0 | + | 9 11 1 0 0 0 0 |
| − | 11 12 2 0 0 0 0 | + | 11 12 2 0 0 0 0 |
| − | 12 13 1 0 0 0 0 | + | 12 13 1 0 0 0 0 |
| − | 13 14 2 0 0 0 0 | + | 13 14 2 0 0 0 0 |
| − | 14 15 1 0 0 0 0 | + | 14 15 1 0 0 0 0 |
| − | 15 16 2 0 0 0 0 | + | 15 16 2 0 0 0 0 |
| − | 16 11 1 0 0 0 0 | + | 16 11 1 0 0 0 0 |
| − | 7 17 2 0 0 0 0 | + | 7 17 2 0 0 0 0 |
| − | 3 18 1 0 0 0 0 | + | 3 18 1 0 0 0 0 |
| − | 1 19 1 0 0 0 0 | + | 1 19 1 0 0 0 0 |
| − | 14 20 1 0 0 0 0 | + | 14 20 1 0 0 0 0 |
| − | 21 22 1 1 0 0 0 | + | 21 22 1 1 0 0 0 |
| − | 22 23 1 1 0 0 0 | + | 22 23 1 1 0 0 0 |
| − | 24 23 1 1 0 0 0 | + | 24 23 1 1 0 0 0 |
| − | 24 25 1 0 0 0 0 | + | 24 25 1 0 0 0 0 |
| − | 25 26 1 0 0 0 0 | + | 25 26 1 0 0 0 0 |
| − | 26 21 1 0 0 0 0 | + | 26 21 1 0 0 0 0 |
| − | 26 27 1 0 0 0 0 | + | 26 27 1 0 0 0 0 |
| − | 27 28 1 0 0 0 0 | + | 27 28 1 0 0 0 0 |
| − | 21 29 1 0 0 0 0 | + | 21 29 1 0 0 0 0 |
| − | 22 30 1 0 0 0 0 | + | 22 30 1 0 0 0 0 |
| − | 23 31 1 0 0 0 0 | + | 23 31 1 0 0 0 0 |
| − | 32 33 1 1 0 0 0 | + | 32 33 1 1 0 0 0 |
| − | 33 34 1 1 0 0 0 | + | 33 34 1 1 0 0 0 |
| − | 35 34 1 1 0 0 0 | + | 35 34 1 1 0 0 0 |
| − | 35 36 1 0 0 0 0 | + | 35 36 1 0 0 0 0 |
| − | 36 37 1 0 0 0 0 | + | 36 37 1 0 0 0 0 |
| − | 37 32 1 0 0 0 0 | + | 37 32 1 0 0 0 0 |
| − | 37 38 1 0 0 0 0 | + | 37 38 1 0 0 0 0 |
| − | 38 39 1 0 0 0 0 | + | 38 39 1 0 0 0 0 |
| − | 32 40 1 0 0 0 0 | + | 32 40 1 0 0 0 0 |
| − | 33 41 1 0 0 0 0 | + | 33 41 1 0 0 0 0 |
| − | 34 42 1 0 0 0 0 | + | 34 42 1 0 0 0 0 |
| − | 35 29 1 0 0 0 0 | + | 35 29 1 0 0 0 0 |
| − | 19 24 1 0 0 0 0 | + | 19 24 1 0 0 0 0 |
| − | S SKP 8 | + | S SKP 8 |
| − | ID FL3FAAGS0058 | + | ID FL3FAAGS0058 |
| − | KNApSAcK_ID C00013608 | + | KNApSAcK_ID C00013608 |
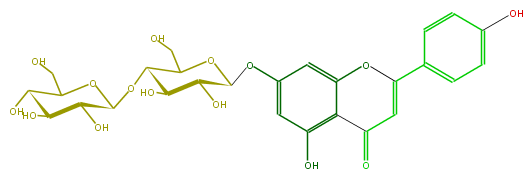
| − | NAME Apigenin 7-cellobioside;5,7,4'-Trihydroxyflavone 7-cellobioside;7-[(4-O-beta-D-Glucopyranosyl-beta-D-glucopyranosyl)oxy]-5-hydroxy-2-(4-hydroxyphenyl)-4H-1-benzopyran-4-one | + | NAME Apigenin 7-cellobioside;5,7,4'-Trihydroxyflavone 7-cellobioside;7-[(4-O-beta-D-Glucopyranosyl-beta-D-glucopyranosyl)oxy]-5-hydroxy-2-(4-hydroxyphenyl)-4H-1-benzopyran-4-one |
| − | CAS_RN 211096-97-8 | + | CAS_RN 211096-97-8 |
| − | FORMULA C27H30O15 | + | FORMULA C27H30O15 |
| − | EXACTMASS 594.15847029 | + | EXACTMASS 594.15847029 |
| − | AVERAGEMASS 594.5181 | + | AVERAGEMASS 594.5181 |
| − | SMILES C(C=4)(=O)c(c(OC4c(c5)ccc(c5)O)3)c(O)cc(c3)OC(O1)C(C(O)C(OC(O2)C(O)C(O)C(O)C2CO)C1CO)O | + | SMILES C(C=4)(=O)c(c(OC4c(c5)ccc(c5)O)3)c(O)cc(c3)OC(O1)C(C(O)C(OC(O2)C(O)C(O)C(O)C2CO)C1CO)O |
M END | M END | ||
| − | |||
Latest revision as of 09:00, 14 March 2009
Copyright: ARM project http://www.metabolome.jp/
42 46 0 0 0 0 0 0 0 0999 V2000
0.3853 0.1971 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.3853 -0.6279 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.0998 -1.0404 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.8142 -0.6279 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.8142 0.1971 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.0998 0.6096 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.5287 -1.0404 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.2432 -0.6279 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.2432 0.1971 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.5287 0.6096 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
3.9576 0.6096 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
4.6721 0.1971 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
5.3866 0.6096 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
5.3866 1.4346 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
4.6721 1.8471 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.9576 1.4346 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.5287 -1.8654 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
1.0998 -1.8654 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-0.3292 0.6096 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
6.1329 1.8654 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-2.8503 0.5285 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.3440 -0.1232 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.5817 0.1930 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.7646 0.0766 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.2709 0.7283 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-2.0333 0.4122 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.2537 0.9355 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.6794 1.2580 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-3.2280 0.0301 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-2.7392 -0.0736 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-1.1445 -0.3697 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-5.7552 -0.0327 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-5.2489 -0.6844 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-4.4866 -0.3682 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-3.6695 -0.4846 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-4.1758 0.1672 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-4.9382 -0.1490 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-5.1587 0.3743 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-5.5844 0.6969 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-6.1329 -0.5311 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-5.6442 -0.6348 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-4.0494 -0.9309 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
1 2 2 0 0 0 0
2 3 1 0 0 0 0
3 4 2 0 0 0 0
4 5 1 0 0 0 0
5 6 2 0 0 0 0
6 1 1 0 0 0 0
4 7 1 0 0 0 0
7 8 1 0 0 0 0
8 9 2 0 0 0 0
9 10 1 0 0 0 0
10 5 1 0 0 0 0
9 11 1 0 0 0 0
11 12 2 0 0 0 0
12 13 1 0 0 0 0
13 14 2 0 0 0 0
14 15 1 0 0 0 0
15 16 2 0 0 0 0
16 11 1 0 0 0 0
7 17 2 0 0 0 0
3 18 1 0 0 0 0
1 19 1 0 0 0 0
14 20 1 0 0 0 0
21 22 1 1 0 0 0
22 23 1 1 0 0 0
24 23 1 1 0 0 0
24 25 1 0 0 0 0
25 26 1 0 0 0 0
26 21 1 0 0 0 0
26 27 1 0 0 0 0
27 28 1 0 0 0 0
21 29 1 0 0 0 0
22 30 1 0 0 0 0
23 31 1 0 0 0 0
32 33 1 1 0 0 0
33 34 1 1 0 0 0
35 34 1 1 0 0 0
35 36 1 0 0 0 0
36 37 1 0 0 0 0
37 32 1 0 0 0 0
37 38 1 0 0 0 0
38 39 1 0 0 0 0
32 40 1 0 0 0 0
33 41 1 0 0 0 0
34 42 1 0 0 0 0
35 29 1 0 0 0 0
19 24 1 0 0 0 0
S SKP 8
ID FL3FAAGS0058
KNApSAcK_ID C00013608
NAME Apigenin 7-cellobioside;5,7,4'-Trihydroxyflavone 7-cellobioside;7-[(4-O-beta-D-Glucopyranosyl-beta-D-glucopyranosyl)oxy]-5-hydroxy-2-(4-hydroxyphenyl)-4H-1-benzopyran-4-one
CAS_RN 211096-97-8
FORMULA C27H30O15
EXACTMASS 594.15847029
AVERAGEMASS 594.5181
SMILES C(C=4)(=O)c(c(OC4c(c5)ccc(c5)O)3)c(O)cc(c3)OC(O1)C(C(O)C(OC(O2)C(O)C(O)C(O)C2CO)C1CO)O
M END