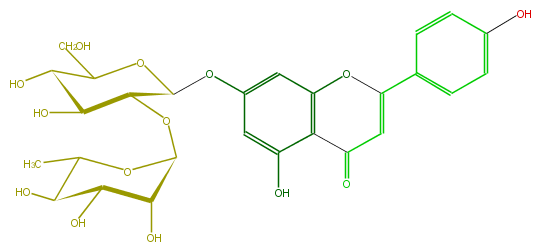
Mol:FL3FAAGS0021
From Metabolomics.JP
(Difference between revisions)
| Line 1: | Line 1: | ||
| − | + | ||
| − | + | ||
| − | Copyright: ARM project http://www.metabolome.jp/ | + | Copyright: ARM project http://www.metabolome.jp/ |
| − | 41 45 0 0 0 0 0 0 0 0999 V2000 | + | 41 45 0 0 0 0 0 0 0 0999 V2000 |
| − | -0.5439 0.6495 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.5439 0.6495 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.5439 -0.1600 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.5439 -0.1600 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.1572 -0.5648 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.1572 -0.5648 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.8582 -0.1600 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.8582 -0.1600 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.8582 0.6495 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.8582 0.6495 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.1572 1.0541 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.1572 1.0541 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.5593 -0.5648 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.5593 -0.5648 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.2603 -0.1600 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.2603 -0.1600 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.2603 0.6495 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.2603 0.6495 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.5593 1.0541 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.5593 1.0541 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.5593 -1.1958 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.5593 -1.1958 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.9610 1.0540 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.9610 1.0540 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.6755 0.6416 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.6755 0.6416 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 4.3900 1.0540 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 4.3900 1.0540 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 4.3900 1.8790 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 4.3900 1.8790 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.6755 2.2915 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.6755 2.2915 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.9610 1.8790 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.9610 1.8790 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.2446 1.0540 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.2446 1.0540 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.1572 -1.3739 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.1572 -1.3739 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 5.1042 2.2915 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 5.1042 2.2915 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -4.4823 1.1209 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -4.4823 1.1209 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.8324 0.2631 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.8324 0.2631 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.8968 0.6270 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.8968 0.6270 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.9940 0.6367 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.9940 0.6367 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.6500 1.2929 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.6500 1.2929 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.6058 0.9498 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.6058 0.9498 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -5.1042 0.8650 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -5.1042 0.8650 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -4.6171 0.2537 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -4.6171 0.2537 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.1132 0.0983 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.1132 0.0983 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.9069 -0.6615 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.9069 -0.6615 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -4.4176 -1.5461 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -4.4176 -1.5461 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.4354 -1.2653 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.4354 -1.2653 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.4472 -1.5461 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.4472 -1.5461 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.9363 -0.6615 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.9363 -0.6615 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.9186 -0.9421 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.9186 -0.9421 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.9974 -1.9924 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.9974 -1.9924 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -4.9853 -1.3563 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -4.9853 -1.3563 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -4.7527 -0.7823 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -4.7527 -0.7823 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.4472 -2.2915 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.4472 -2.2915 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -4.2656 1.6358 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -4.2656 1.6358 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.3652 1.1160 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.3652 1.1160 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1 2 1 0 0 0 0 | + | 1 2 1 0 0 0 0 |
| − | 2 3 2 0 0 0 0 | + | 2 3 2 0 0 0 0 |
| − | 3 4 1 0 0 0 0 | + | 3 4 1 0 0 0 0 |
| − | 4 5 2 0 0 0 0 | + | 4 5 2 0 0 0 0 |
| − | 5 6 1 0 0 0 0 | + | 5 6 1 0 0 0 0 |
| − | 6 1 2 0 0 0 0 | + | 6 1 2 0 0 0 0 |
| − | 4 7 1 0 0 0 0 | + | 4 7 1 0 0 0 0 |
| − | 7 8 1 0 0 0 0 | + | 7 8 1 0 0 0 0 |
| − | 8 9 2 0 0 0 0 | + | 8 9 2 0 0 0 0 |
| − | 9 10 1 0 0 0 0 | + | 9 10 1 0 0 0 0 |
| − | 10 5 1 0 0 0 0 | + | 10 5 1 0 0 0 0 |
| − | 7 11 2 0 0 0 0 | + | 7 11 2 0 0 0 0 |
| − | 9 12 1 0 0 0 0 | + | 9 12 1 0 0 0 0 |
| − | 12 13 2 0 0 0 0 | + | 12 13 2 0 0 0 0 |
| − | 13 14 1 0 0 0 0 | + | 13 14 1 0 0 0 0 |
| − | 14 15 2 0 0 0 0 | + | 14 15 2 0 0 0 0 |
| − | 15 16 1 0 0 0 0 | + | 15 16 1 0 0 0 0 |
| − | 16 17 2 0 0 0 0 | + | 16 17 2 0 0 0 0 |
| − | 17 12 1 0 0 0 0 | + | 17 12 1 0 0 0 0 |
| − | 1 18 1 0 0 0 0 | + | 1 18 1 0 0 0 0 |
| − | 3 19 1 0 0 0 0 | + | 3 19 1 0 0 0 0 |
| − | 15 20 1 0 0 0 0 | + | 15 20 1 0 0 0 0 |
| − | 21 22 1 1 0 0 0 | + | 21 22 1 1 0 0 0 |
| − | 22 23 1 1 0 0 0 | + | 22 23 1 1 0 0 0 |
| − | 24 23 1 1 0 0 0 | + | 24 23 1 1 0 0 0 |
| − | 24 25 1 0 0 0 0 | + | 24 25 1 0 0 0 0 |
| − | 25 26 1 0 0 0 0 | + | 25 26 1 0 0 0 0 |
| − | 26 21 1 0 0 0 0 | + | 26 21 1 0 0 0 0 |
| − | 21 27 1 0 0 0 0 | + | 21 27 1 0 0 0 0 |
| − | 22 28 1 0 0 0 0 | + | 22 28 1 0 0 0 0 |
| − | 23 29 1 0 0 0 0 | + | 23 29 1 0 0 0 0 |
| − | 30 31 1 0 0 0 0 | + | 30 31 1 0 0 0 0 |
| − | 31 32 1 1 0 0 0 | + | 31 32 1 1 0 0 0 |
| − | 32 33 1 1 0 0 0 | + | 32 33 1 1 0 0 0 |
| − | 34 33 1 1 0 0 0 | + | 34 33 1 1 0 0 0 |
| − | 34 35 1 0 0 0 0 | + | 34 35 1 0 0 0 0 |
| − | 35 30 1 0 0 0 0 | + | 35 30 1 0 0 0 0 |
| − | 34 29 1 0 0 0 0 | + | 34 29 1 0 0 0 0 |
| − | 32 36 1 0 0 0 0 | + | 32 36 1 0 0 0 0 |
| − | 31 37 1 0 0 0 0 | + | 31 37 1 0 0 0 0 |
| − | 30 38 1 0 0 0 0 | + | 30 38 1 0 0 0 0 |
| − | 33 39 1 0 0 0 0 | + | 33 39 1 0 0 0 0 |
| − | 18 24 1 0 0 0 0 | + | 18 24 1 0 0 0 0 |
| − | 40 41 1 0 0 0 0 | + | 40 41 1 0 0 0 0 |
| − | 26 40 1 0 0 0 0 | + | 26 40 1 0 0 0 0 |
| − | M STY 1 1 SUP | + | M STY 1 1 SUP |
| − | M SLB 1 1 1 | + | M SLB 1 1 1 |
| − | M SAL 1 2 40 41 | + | M SAL 1 2 40 41 |
| − | M SBL 1 1 45 | + | M SBL 1 1 45 |
| − | M SMT 1 ^CH2OH | + | M SMT 1 ^CH2OH |
| − | M SBV 1 45 0.6597 -0.6861 | + | M SBV 1 45 0.6597 -0.6861 |
| − | S SKP 5 | + | S SKP 5 |
| − | ID FL3FAAGS0021 | + | ID FL3FAAGS0021 |
| − | FORMULA C27H30O14 | + | FORMULA C27H30O14 |
| − | EXACTMASS 578.163555668 | + | EXACTMASS 578.163555668 |
| − | AVERAGEMASS 578.5187000000001 | + | AVERAGEMASS 578.5187000000001 |
| − | SMILES c(c13)c(OC(C4OC(O5)C(C(O)C(O)C(C)5)O)OC(CO)C(C4O)O)cc(c(C(C=C(O3)c(c2)ccc(c2)O)=O)1)O | + | SMILES c(c13)c(OC(C4OC(O5)C(C(O)C(O)C(C)5)O)OC(CO)C(C4O)O)cc(c(C(C=C(O3)c(c2)ccc(c2)O)=O)1)O |
M END | M END | ||
| − | |||
Latest revision as of 09:00, 14 March 2009
Copyright: ARM project http://www.metabolome.jp/
41 45 0 0 0 0 0 0 0 0999 V2000
-0.5439 0.6495 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.5439 -0.1600 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.1572 -0.5648 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.8582 -0.1600 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.8582 0.6495 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.1572 1.0541 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.5593 -0.5648 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.2603 -0.1600 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.2603 0.6495 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.5593 1.0541 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
1.5593 -1.1958 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
2.9610 1.0540 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.6755 0.6416 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
4.3900 1.0540 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
4.3900 1.8790 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.6755 2.2915 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.9610 1.8790 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.2446 1.0540 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
0.1572 -1.3739 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
5.1042 2.2915 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-4.4823 1.1209 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-3.8324 0.2631 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.8968 0.6270 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.9940 0.6367 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.6500 1.2929 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-3.6058 0.9498 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-5.1042 0.8650 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-4.6171 0.2537 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-2.1132 0.0983 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-3.9069 -0.6615 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-4.4176 -1.5461 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-3.4354 -1.2653 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.4472 -1.5461 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.9363 -0.6615 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.9186 -0.9421 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-3.9974 -1.9924 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-4.9853 -1.3563 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-4.7527 -0.7823 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.4472 -2.2915 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-4.2656 1.6358 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-3.3652 1.1160 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
1 2 1 0 0 0 0
2 3 2 0 0 0 0
3 4 1 0 0 0 0
4 5 2 0 0 0 0
5 6 1 0 0 0 0
6 1 2 0 0 0 0
4 7 1 0 0 0 0
7 8 1 0 0 0 0
8 9 2 0 0 0 0
9 10 1 0 0 0 0
10 5 1 0 0 0 0
7 11 2 0 0 0 0
9 12 1 0 0 0 0
12 13 2 0 0 0 0
13 14 1 0 0 0 0
14 15 2 0 0 0 0
15 16 1 0 0 0 0
16 17 2 0 0 0 0
17 12 1 0 0 0 0
1 18 1 0 0 0 0
3 19 1 0 0 0 0
15 20 1 0 0 0 0
21 22 1 1 0 0 0
22 23 1 1 0 0 0
24 23 1 1 0 0 0
24 25 1 0 0 0 0
25 26 1 0 0 0 0
26 21 1 0 0 0 0
21 27 1 0 0 0 0
22 28 1 0 0 0 0
23 29 1 0 0 0 0
30 31 1 0 0 0 0
31 32 1 1 0 0 0
32 33 1 1 0 0 0
34 33 1 1 0 0 0
34 35 1 0 0 0 0
35 30 1 0 0 0 0
34 29 1 0 0 0 0
32 36 1 0 0 0 0
31 37 1 0 0 0 0
30 38 1 0 0 0 0
33 39 1 0 0 0 0
18 24 1 0 0 0 0
40 41 1 0 0 0 0
26 40 1 0 0 0 0
M STY 1 1 SUP
M SLB 1 1 1
M SAL 1 2 40 41
M SBL 1 1 45
M SMT 1 ^CH2OH
M SBV 1 45 0.6597 -0.6861
S SKP 5
ID FL3FAAGS0021
FORMULA C27H30O14
EXACTMASS 578.163555668
AVERAGEMASS 578.5187000000001
SMILES c(c13)c(OC(C4OC(O5)C(C(O)C(O)C(C)5)O)OC(CO)C(C4O)O)cc(c(C(C=C(O3)c(c2)ccc(c2)O)=O)1)O
M END