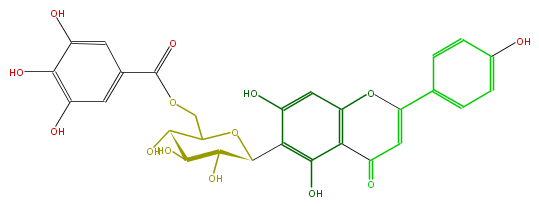
Mol:FL3FAACS0083
From Metabolomics.JP
(Difference between revisions)
| Line 1: | Line 1: | ||
| − | + | ||
| − | + | ||
| − | Copyright: ARM project http://www.metabolome.jp/ | + | Copyright: ARM project http://www.metabolome.jp/ |
| − | 42 46 0 0 0 0 0 0 0 0999 V2000 | + | 42 46 0 0 0 0 0 0 0 0999 V2000 |
| − | 3.1600 -0.1691 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.1600 -0.1691 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.4456 0.2434 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.4456 0.2434 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.7311 -0.1691 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.7311 -0.1691 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.7311 -0.9941 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.7311 -0.9941 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.4456 -1.4066 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.4456 -1.4066 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.1600 -0.9941 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.1600 -0.9941 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.0166 0.2434 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.0166 0.2434 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.3021 -0.1691 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.3021 -0.1691 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.3021 -0.9941 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.3021 -0.9941 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.0166 -1.4066 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.0166 -1.4066 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.4456 -1.9787 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.4456 -1.9787 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.9614 0.2936 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.9614 0.2936 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 4.6759 -0.1189 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 4.6759 -0.1189 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 5.3903 0.2936 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 5.3903 0.2936 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 5.3903 1.1186 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 5.3903 1.1186 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 4.6759 1.5311 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 4.6759 1.5311 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.9614 1.1186 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.9614 1.1186 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 6.0738 1.5131 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 6.0738 1.5131 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.0166 -2.1766 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.0166 -2.1766 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.3883 0.2295 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.3883 0.2295 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.4373 -0.6802 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.4373 -0.6802 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.0247 -1.3948 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.0247 -1.3948 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.2264 -1.1857 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.2264 -1.1857 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.4329 -1.4126 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.4329 -1.4126 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.8454 -0.6978 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.8454 -0.6978 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.6438 -0.9069 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.6438 -0.9069 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.8008 -0.3210 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.8008 -0.3210 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.9205 -1.1634 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.9205 -1.1634 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.4734 -1.1358 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.4734 -1.1358 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.3978 -1.8256 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.3978 -1.8256 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -4.0170 0.0263 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -4.0170 0.0263 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.6000 0.7381 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.6000 0.7381 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -4.0079 1.4552 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -4.0079 1.4552 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -4.8329 1.4605 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -4.8329 1.4605 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -5.2499 0.7487 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -5.2499 0.7487 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -4.8420 0.0316 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -4.8420 0.0316 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -5.2585 -0.6793 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -5.2585 -0.6793 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -6.0738 0.7539 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -6.0738 0.7539 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -5.2403 2.1766 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -5.2403 2.1766 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.7761 0.7329 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.7761 0.7329 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.3596 1.4438 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.3596 1.4438 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.3687 0.0168 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.3687 0.0168 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1 2 1 0 0 0 0 | + | 1 2 1 0 0 0 0 |
| − | 2 3 1 0 0 0 0 | + | 2 3 1 0 0 0 0 |
| − | 3 4 2 0 0 0 0 | + | 3 4 2 0 0 0 0 |
| − | 4 5 1 0 0 0 0 | + | 4 5 1 0 0 0 0 |
| − | 5 6 1 0 0 0 0 | + | 5 6 1 0 0 0 0 |
| − | 6 1 2 0 0 0 0 | + | 6 1 2 0 0 0 0 |
| − | 3 7 1 0 0 0 0 | + | 3 7 1 0 0 0 0 |
| − | 7 8 2 0 0 0 0 | + | 7 8 2 0 0 0 0 |
| − | 8 9 1 0 0 0 0 | + | 8 9 1 0 0 0 0 |
| − | 9 10 2 0 0 0 0 | + | 9 10 2 0 0 0 0 |
| − | 10 4 1 0 0 0 0 | + | 10 4 1 0 0 0 0 |
| − | 5 11 2 0 0 0 0 | + | 5 11 2 0 0 0 0 |
| − | 1 12 1 0 0 0 0 | + | 1 12 1 0 0 0 0 |
| − | 12 13 2 0 0 0 0 | + | 12 13 2 0 0 0 0 |
| − | 13 14 1 0 0 0 0 | + | 13 14 1 0 0 0 0 |
| − | 14 15 2 0 0 0 0 | + | 14 15 2 0 0 0 0 |
| − | 15 16 1 0 0 0 0 | + | 15 16 1 0 0 0 0 |
| − | 16 17 2 0 0 0 0 | + | 16 17 2 0 0 0 0 |
| − | 17 12 1 0 0 0 0 | + | 17 12 1 0 0 0 0 |
| − | 15 18 1 0 0 0 0 | + | 15 18 1 0 0 0 0 |
| − | 10 19 1 0 0 0 0 | + | 10 19 1 0 0 0 0 |
| − | 8 20 1 0 0 0 0 | + | 8 20 1 0 0 0 0 |
| − | 21 22 1 1 0 0 0 | + | 21 22 1 1 0 0 0 |
| − | 22 23 1 1 0 0 0 | + | 22 23 1 1 0 0 0 |
| − | 24 23 1 1 0 0 0 | + | 24 23 1 1 0 0 0 |
| − | 24 25 1 0 0 0 0 | + | 24 25 1 0 0 0 0 |
| − | 25 26 1 0 0 0 0 | + | 25 26 1 0 0 0 0 |
| − | 26 21 1 0 0 0 0 | + | 26 21 1 0 0 0 0 |
| − | 26 27 1 0 0 0 0 | + | 26 27 1 0 0 0 0 |
| − | 21 28 1 0 0 0 0 | + | 21 28 1 0 0 0 0 |
| − | 22 29 1 0 0 0 0 | + | 22 29 1 0 0 0 0 |
| − | 23 30 1 0 0 0 0 | + | 23 30 1 0 0 0 0 |
| − | 24 9 1 0 0 0 0 | + | 24 9 1 0 0 0 0 |
| − | 31 32 2 0 0 0 0 | + | 31 32 2 0 0 0 0 |
| − | 32 33 1 0 0 0 0 | + | 32 33 1 0 0 0 0 |
| − | 33 34 2 0 0 0 0 | + | 33 34 2 0 0 0 0 |
| − | 34 35 1 0 0 0 0 | + | 34 35 1 0 0 0 0 |
| − | 35 36 2 0 0 0 0 | + | 35 36 2 0 0 0 0 |
| − | 36 31 1 0 0 0 0 | + | 36 31 1 0 0 0 0 |
| − | 36 37 1 0 0 0 0 | + | 36 37 1 0 0 0 0 |
| − | 35 38 1 0 0 0 0 | + | 35 38 1 0 0 0 0 |
| − | 34 39 1 0 0 0 0 | + | 34 39 1 0 0 0 0 |
| − | 32 40 1 0 0 0 0 | + | 32 40 1 0 0 0 0 |
| − | 40 41 2 0 0 0 0 | + | 40 41 2 0 0 0 0 |
| − | 40 42 1 0 0 0 0 | + | 40 42 1 0 0 0 0 |
| − | 42 27 1 0 0 0 0 | + | 42 27 1 0 0 0 0 |
| − | S SKP 8 | + | S SKP 8 |
| − | ID FL3FAACS0083 | + | ID FL3FAACS0083 |
| − | KNApSAcK_ID C00014084 | + | KNApSAcK_ID C00014084 |
| − | NAME 2''-O-Galloylisovitexin | + | NAME 2''-O-Galloylisovitexin |
| − | CAS_RN 267883-58-9 | + | CAS_RN 267883-58-9 |
| − | FORMULA C28H24O14 | + | FORMULA C28H24O14 |
| − | EXACTMASS 584.116605476 | + | EXACTMASS 584.116605476 |
| − | AVERAGEMASS 584.48176 | + | AVERAGEMASS 584.48176 |
| − | SMILES O=C(C=4)c(c(OC(c(c5)ccc(c5)O)4)3)c(c(c(O)c3)C(O1)C(C(O)C(C1COC(c(c2)cc(O)c(c(O)2)O)=O)O)O)O | + | SMILES O=C(C=4)c(c(OC(c(c5)ccc(c5)O)4)3)c(c(c(O)c3)C(O1)C(C(O)C(C1COC(c(c2)cc(O)c(c(O)2)O)=O)O)O)O |
M END | M END | ||
| − | |||
Latest revision as of 09:00, 14 March 2009
Copyright: ARM project http://www.metabolome.jp/
42 46 0 0 0 0 0 0 0 0999 V2000
3.1600 -0.1691 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.4456 0.2434 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
1.7311 -0.1691 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.7311 -0.9941 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.4456 -1.4066 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.1600 -0.9941 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.0166 0.2434 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.3021 -0.1691 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.3021 -0.9941 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.0166 -1.4066 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.4456 -1.9787 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
3.9614 0.2936 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
4.6759 -0.1189 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
5.3903 0.2936 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
5.3903 1.1186 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
4.6759 1.5311 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.9614 1.1186 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
6.0738 1.5131 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
1.0166 -2.1766 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-0.3883 0.2295 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-2.4373 -0.6802 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.0247 -1.3948 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.2264 -1.1857 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.4329 -1.4126 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.8454 -0.6978 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-1.6438 -0.9069 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.8008 -0.3210 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.9205 -1.1634 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-2.4734 -1.1358 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-1.3978 -1.8256 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-4.0170 0.0263 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-3.6000 0.7381 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-4.0079 1.4552 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-4.8329 1.4605 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-5.2499 0.7487 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-4.8420 0.0316 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-5.2585 -0.6793 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-6.0738 0.7539 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-5.2403 2.1766 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-2.7761 0.7329 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.3596 1.4438 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-2.3687 0.0168 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
1 2 1 0 0 0 0
2 3 1 0 0 0 0
3 4 2 0 0 0 0
4 5 1 0 0 0 0
5 6 1 0 0 0 0
6 1 2 0 0 0 0
3 7 1 0 0 0 0
7 8 2 0 0 0 0
8 9 1 0 0 0 0
9 10 2 0 0 0 0
10 4 1 0 0 0 0
5 11 2 0 0 0 0
1 12 1 0 0 0 0
12 13 2 0 0 0 0
13 14 1 0 0 0 0
14 15 2 0 0 0 0
15 16 1 0 0 0 0
16 17 2 0 0 0 0
17 12 1 0 0 0 0
15 18 1 0 0 0 0
10 19 1 0 0 0 0
8 20 1 0 0 0 0
21 22 1 1 0 0 0
22 23 1 1 0 0 0
24 23 1 1 0 0 0
24 25 1 0 0 0 0
25 26 1 0 0 0 0
26 21 1 0 0 0 0
26 27 1 0 0 0 0
21 28 1 0 0 0 0
22 29 1 0 0 0 0
23 30 1 0 0 0 0
24 9 1 0 0 0 0
31 32 2 0 0 0 0
32 33 1 0 0 0 0
33 34 2 0 0 0 0
34 35 1 0 0 0 0
35 36 2 0 0 0 0
36 31 1 0 0 0 0
36 37 1 0 0 0 0
35 38 1 0 0 0 0
34 39 1 0 0 0 0
32 40 1 0 0 0 0
40 41 2 0 0 0 0
40 42 1 0 0 0 0
42 27 1 0 0 0 0
S SKP 8
ID FL3FAACS0083
KNApSAcK_ID C00014084
NAME 2''-O-Galloylisovitexin
CAS_RN 267883-58-9
FORMULA C28H24O14
EXACTMASS 584.116605476
AVERAGEMASS 584.48176
SMILES O=C(C=4)c(c(OC(c(c5)ccc(c5)O)4)3)c(c(c(O)c3)C(O1)C(C(O)C(C1COC(c(c2)cc(O)c(c(O)2)O)=O)O)O)O
M END