Mol:FL3FAACS0077
From Metabolomics.JP
(Difference between revisions)
| Line 1: | Line 1: | ||
| − | + | ||
| − | + | ||
| − | Copyright: ARM project http://www.metabolome.jp/ | + | Copyright: ARM project http://www.metabolome.jp/ |
| − | 42 46 0 0 0 0 0 0 0 0999 V2000 | + | 42 46 0 0 0 0 0 0 0 0999 V2000 |
| − | 3.0236 0.1631 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.0236 0.1631 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.3091 0.5756 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.3091 0.5756 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.5946 0.1631 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.5946 0.1631 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.5946 -0.6619 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.5946 -0.6619 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.3091 -1.0744 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.3091 -1.0744 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.0236 -0.6619 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.0236 -0.6619 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.8801 0.5756 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.8801 0.5756 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.1657 0.1631 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.1657 0.1631 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.1657 -0.6619 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.1657 -0.6619 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.8801 -1.0744 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.8801 -1.0744 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.4801 0.5359 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.4801 0.5359 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.8801 -1.8139 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.8801 -1.8139 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.3091 -1.6465 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.3091 -1.6465 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.8053 0.6144 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.8053 0.6144 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 4.5198 0.2019 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 4.5198 0.2019 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 5.2342 0.6144 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 5.2342 0.6144 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 5.2342 1.4394 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 5.2342 1.4394 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 4.5198 1.8519 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 4.5198 1.8519 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.8053 1.4394 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.8053 1.4394 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 5.8495 1.7946 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 5.8495 1.7946 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.3938 -1.0663 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.3938 -1.0663 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.8259 -1.6651 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.8259 -1.6651 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.0983 -1.2756 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.0983 -1.2756 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.2738 -1.3112 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.2738 -1.3112 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.8416 -0.7123 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.8416 -0.7123 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.5693 -1.1018 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.5693 -1.1018 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.8794 -0.5302 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.8794 -0.5302 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.4109 -0.3726 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.4109 -0.3726 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.8129 -0.8093 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.8129 -0.8093 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.7214 -1.6414 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.7214 -1.6414 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.7856 -1.8519 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.7856 -1.8519 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -5.3967 -0.0974 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -5.3967 -0.0974 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -4.9841 -0.8121 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -4.9841 -0.8121 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -4.1858 -0.6029 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -4.1858 -0.6029 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.3923 -0.8298 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.3923 -0.8298 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.8049 -0.1150 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.8049 -0.1150 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -4.6033 -0.3241 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -4.6033 -0.3241 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -4.7716 0.3041 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -4.7716 0.3041 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -5.2516 0.5812 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -5.2516 0.5812 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -5.7444 0.2503 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -5.7444 0.2503 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -5.8495 -0.5802 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -5.8495 -0.5802 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -4.0161 -1.2363 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -4.0161 -1.2363 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1 2 1 0 0 0 0 | + | 1 2 1 0 0 0 0 |
| − | 2 3 1 0 0 0 0 | + | 2 3 1 0 0 0 0 |
| − | 3 4 2 0 0 0 0 | + | 3 4 2 0 0 0 0 |
| − | 4 5 1 0 0 0 0 | + | 4 5 1 0 0 0 0 |
| − | 5 6 1 0 0 0 0 | + | 5 6 1 0 0 0 0 |
| − | 6 1 2 0 0 0 0 | + | 6 1 2 0 0 0 0 |
| − | 3 7 1 0 0 0 0 | + | 3 7 1 0 0 0 0 |
| − | 7 8 2 0 0 0 0 | + | 7 8 2 0 0 0 0 |
| − | 8 9 1 0 0 0 0 | + | 8 9 1 0 0 0 0 |
| − | 9 10 2 0 0 0 0 | + | 9 10 2 0 0 0 0 |
| − | 10 4 1 0 0 0 0 | + | 10 4 1 0 0 0 0 |
| − | 8 11 1 0 0 0 0 | + | 8 11 1 0 0 0 0 |
| − | 10 12 1 0 0 0 0 | + | 10 12 1 0 0 0 0 |
| − | 5 13 2 0 0 0 0 | + | 5 13 2 0 0 0 0 |
| − | 1 14 1 0 0 0 0 | + | 1 14 1 0 0 0 0 |
| − | 14 15 2 0 0 0 0 | + | 14 15 2 0 0 0 0 |
| − | 15 16 1 0 0 0 0 | + | 15 16 1 0 0 0 0 |
| − | 16 17 2 0 0 0 0 | + | 16 17 2 0 0 0 0 |
| − | 17 18 1 0 0 0 0 | + | 17 18 1 0 0 0 0 |
| − | 18 19 2 0 0 0 0 | + | 18 19 2 0 0 0 0 |
| − | 19 14 1 0 0 0 0 | + | 19 14 1 0 0 0 0 |
| − | 17 20 1 0 0 0 0 | + | 17 20 1 0 0 0 0 |
| − | 21 22 1 1 0 0 0 | + | 21 22 1 1 0 0 0 |
| − | 22 23 1 1 0 0 0 | + | 22 23 1 1 0 0 0 |
| − | 24 23 1 1 0 0 0 | + | 24 23 1 1 0 0 0 |
| − | 24 25 1 0 0 0 0 | + | 24 25 1 0 0 0 0 |
| − | 25 26 1 0 0 0 0 | + | 25 26 1 0 0 0 0 |
| − | 26 21 1 0 0 0 0 | + | 26 21 1 0 0 0 0 |
| − | 26 27 1 0 0 0 0 | + | 26 27 1 0 0 0 0 |
| − | 27 28 1 0 0 0 0 | + | 27 28 1 0 0 0 0 |
| − | 21 29 1 0 0 0 0 | + | 21 29 1 0 0 0 0 |
| − | 22 30 1 0 0 0 0 | + | 22 30 1 0 0 0 0 |
| − | 23 31 1 0 0 0 0 | + | 23 31 1 0 0 0 0 |
| − | 24 9 1 0 0 0 0 | + | 24 9 1 0 0 0 0 |
| − | 32 33 1 1 0 0 0 | + | 32 33 1 1 0 0 0 |
| − | 33 34 1 1 0 0 0 | + | 33 34 1 1 0 0 0 |
| − | 35 34 1 1 0 0 0 | + | 35 34 1 1 0 0 0 |
| − | 35 36 1 0 0 0 0 | + | 35 36 1 0 0 0 0 |
| − | 36 37 1 0 0 0 0 | + | 36 37 1 0 0 0 0 |
| − | 37 32 1 0 0 0 0 | + | 37 32 1 0 0 0 0 |
| − | 37 38 1 0 0 0 0 | + | 37 38 1 0 0 0 0 |
| − | 38 39 1 0 0 0 0 | + | 38 39 1 0 0 0 0 |
| − | 32 40 1 0 0 0 0 | + | 32 40 1 0 0 0 0 |
| − | 33 41 1 0 0 0 0 | + | 33 41 1 0 0 0 0 |
| − | 34 42 1 0 0 0 0 | + | 34 42 1 0 0 0 0 |
| − | 35 28 1 0 0 0 0 | + | 35 28 1 0 0 0 0 |
| − | S SKP 8 | + | S SKP 8 |
| − | ID FL3FAACS0077 | + | ID FL3FAACS0077 |
| − | KNApSAcK_ID C00014048 | + | KNApSAcK_ID C00014048 |
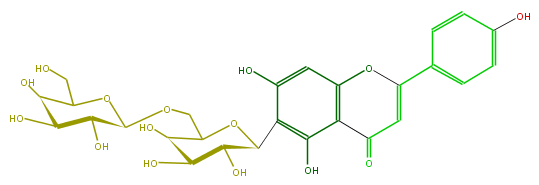
| − | NAME 6-C-Galactosylapigenin 6''-O-galactoside;6-(6-O-beta-D-Galactopyranosyl-beta-D-galactopyranosyl)-5,7-dihydroxy-2-(4-hydroxyphenyl)-4H-1-benzopyran-4-one | + | NAME 6-C-Galactosylapigenin 6''-O-galactoside;6-(6-O-beta-D-Galactopyranosyl-beta-D-galactopyranosyl)-5,7-dihydroxy-2-(4-hydroxyphenyl)-4H-1-benzopyran-4-one |
| − | CAS_RN 615558-90-2 | + | CAS_RN 615558-90-2 |
| − | FORMULA C27H30O15 | + | FORMULA C27H30O15 |
| − | EXACTMASS 594.15847029 | + | EXACTMASS 594.15847029 |
| − | AVERAGEMASS 594.5181 | + | AVERAGEMASS 594.5181 |
| − | SMILES c(c5)(ccc(c5)C(O1)=CC(c(c2O)c1cc(c(C(O3)C(C(O)C(O)C(COC(C4O)OC(C(C(O)4)O)CO)3)O)2)O)=O)O | + | SMILES c(c5)(ccc(c5)C(O1)=CC(c(c2O)c1cc(c(C(O3)C(C(O)C(O)C(COC(C4O)OC(C(C(O)4)O)CO)3)O)2)O)=O)O |
M END | M END | ||
| − | |||
Latest revision as of 09:00, 14 March 2009
Copyright: ARM project http://www.metabolome.jp/
42 46 0 0 0 0 0 0 0 0999 V2000
3.0236 0.1631 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.3091 0.5756 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
1.5946 0.1631 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.5946 -0.6619 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.3091 -1.0744 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.0236 -0.6619 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.8801 0.5756 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.1657 0.1631 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.1657 -0.6619 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.8801 -1.0744 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.4801 0.5359 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
0.8801 -1.8139 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
2.3091 -1.6465 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
3.8053 0.6144 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
4.5198 0.2019 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
5.2342 0.6144 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
5.2342 1.4394 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
4.5198 1.8519 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.8053 1.4394 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
5.8495 1.7946 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-2.3938 -1.0663 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.8259 -1.6651 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.0983 -1.2756 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.2738 -1.3112 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.8416 -0.7123 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-1.5693 -1.1018 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.8794 -0.5302 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.4109 -0.3726 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-2.8129 -0.8093 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-2.7214 -1.6414 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-0.7856 -1.8519 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-5.3967 -0.0974 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-4.9841 -0.8121 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-4.1858 -0.6029 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-3.3923 -0.8298 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-3.8049 -0.1150 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-4.6033 -0.3241 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-4.7716 0.3041 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-5.2516 0.5812 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-5.7444 0.2503 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-5.8495 -0.5802 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-4.0161 -1.2363 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
1 2 1 0 0 0 0
2 3 1 0 0 0 0
3 4 2 0 0 0 0
4 5 1 0 0 0 0
5 6 1 0 0 0 0
6 1 2 0 0 0 0
3 7 1 0 0 0 0
7 8 2 0 0 0 0
8 9 1 0 0 0 0
9 10 2 0 0 0 0
10 4 1 0 0 0 0
8 11 1 0 0 0 0
10 12 1 0 0 0 0
5 13 2 0 0 0 0
1 14 1 0 0 0 0
14 15 2 0 0 0 0
15 16 1 0 0 0 0
16 17 2 0 0 0 0
17 18 1 0 0 0 0
18 19 2 0 0 0 0
19 14 1 0 0 0 0
17 20 1 0 0 0 0
21 22 1 1 0 0 0
22 23 1 1 0 0 0
24 23 1 1 0 0 0
24 25 1 0 0 0 0
25 26 1 0 0 0 0
26 21 1 0 0 0 0
26 27 1 0 0 0 0
27 28 1 0 0 0 0
21 29 1 0 0 0 0
22 30 1 0 0 0 0
23 31 1 0 0 0 0
24 9 1 0 0 0 0
32 33 1 1 0 0 0
33 34 1 1 0 0 0
35 34 1 1 0 0 0
35 36 1 0 0 0 0
36 37 1 0 0 0 0
37 32 1 0 0 0 0
37 38 1 0 0 0 0
38 39 1 0 0 0 0
32 40 1 0 0 0 0
33 41 1 0 0 0 0
34 42 1 0 0 0 0
35 28 1 0 0 0 0
S SKP 8
ID FL3FAACS0077
KNApSAcK_ID C00014048
NAME 6-C-Galactosylapigenin 6''-O-galactoside;6-(6-O-beta-D-Galactopyranosyl-beta-D-galactopyranosyl)-5,7-dihydroxy-2-(4-hydroxyphenyl)-4H-1-benzopyran-4-one
CAS_RN 615558-90-2
FORMULA C27H30O15
EXACTMASS 594.15847029
AVERAGEMASS 594.5181
SMILES c(c5)(ccc(c5)C(O1)=CC(c(c2O)c1cc(c(C(O3)C(C(O)C(O)C(COC(C4O)OC(C(C(O)4)O)CO)3)O)2)O)=O)O
M END