Mol:FL3FAACS0074
From Metabolomics.JP
(Difference between revisions)
| Line 1: | Line 1: | ||
| − | + | ||
| − | + | ||
| − | Copyright: ARM project http://www.metabolome.jp/ | + | Copyright: ARM project http://www.metabolome.jp/ |
| − | 42 46 0 0 0 0 0 0 0 0999 V2000 | + | 42 46 0 0 0 0 0 0 0 0999 V2000 |
| − | 1.7915 -1.2818 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.7915 -1.2818 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.0770 -0.8693 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.0770 -0.8693 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.3625 -1.2818 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.3625 -1.2818 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.3625 -2.1068 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.3625 -2.1068 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.0770 -2.5193 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.0770 -2.5193 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.7915 -2.1068 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.7915 -2.1068 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.3520 -0.8693 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.3520 -0.8693 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.0664 -1.2818 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.0664 -1.2818 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.0664 -2.1068 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.0664 -2.1068 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.3520 -2.5193 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.3520 -2.5193 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.7375 -0.8944 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.7375 -0.8944 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.3520 -3.2588 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.3520 -3.2588 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.0770 -3.0914 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.0770 -3.0914 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.5732 -0.8305 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.5732 -0.8305 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.2877 -1.2430 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.2877 -1.2430 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 4.0022 -0.8305 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 4.0022 -0.8305 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 4.0022 -0.0055 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 4.0022 -0.0055 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.2877 0.4070 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.2877 0.4070 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.5732 -0.0055 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.5732 -0.0055 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 4.6174 0.3497 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 4.6174 0.3497 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.5168 1.5446 0.0000 C 0 0 2 0 0 0 0 0 0 0 0 0 | + | -1.5168 1.5446 0.0000 C 0 0 2 0 0 0 0 0 0 0 0 0 |
| − | -1.8134 0.7744 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0 | + | -1.8134 0.7744 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0 |
| − | -1.1489 0.2849 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0 | + | -1.1489 0.2849 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0 |
| − | -0.8267 -0.4748 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0 | + | -0.8267 -0.4748 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0 |
| − | -0.5300 0.2953 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.5300 0.2953 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.1945 0.7849 0.0000 C 0 0 3 0 0 0 0 0 0 0 0 0 | + | -1.1945 0.7849 0.0000 C 0 0 3 0 0 0 0 0 0 0 0 0 |
| − | -0.8375 1.2752 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.8375 1.2752 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.1115 2.2980 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.1115 2.2980 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.8402 2.4909 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.8402 2.4909 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.9765 1.3523 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.9765 1.3523 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.7545 0.0161 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.7545 0.0161 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -4.1342 2.6227 0.0000 C 0 0 2 0 0 0 0 0 0 0 0 0 | + | -4.1342 2.6227 0.0000 C 0 0 2 0 0 0 0 0 0 0 0 0 |
| − | -3.7215 1.9080 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0 | + | -3.7215 1.9080 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0 |
| − | -2.9232 2.1171 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0 | + | -2.9232 2.1171 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0 |
| − | -2.1297 1.8903 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0 | + | -2.1297 1.8903 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0 |
| − | -2.5423 2.6050 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.5423 2.6050 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.3407 2.3960 0.0000 C 0 0 3 0 0 0 0 0 0 0 0 0 | + | -3.3407 2.3960 0.0000 C 0 0 3 0 0 0 0 0 0 0 0 0 |
| − | -3.4977 2.9818 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.4977 2.9818 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.9775 3.2588 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.9775 3.2588 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -4.6174 2.1395 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -4.6174 2.1395 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -4.1702 2.1670 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -4.1702 2.1670 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.0947 1.4772 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.0947 1.4772 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1 2 1 0 0 0 0 | + | 1 2 1 0 0 0 0 |
| − | 2 3 1 0 0 0 0 | + | 2 3 1 0 0 0 0 |
| − | 3 4 2 0 0 0 0 | + | 3 4 2 0 0 0 0 |
| − | 4 5 1 0 0 0 0 | + | 4 5 1 0 0 0 0 |
| − | 5 6 1 0 0 0 0 | + | 5 6 1 0 0 0 0 |
| − | 6 1 2 0 0 0 0 | + | 6 1 2 0 0 0 0 |
| − | 3 7 1 0 0 0 0 | + | 3 7 1 0 0 0 0 |
| − | 7 8 2 0 0 0 0 | + | 7 8 2 0 0 0 0 |
| − | 8 9 1 0 0 0 0 | + | 8 9 1 0 0 0 0 |
| − | 9 10 2 0 0 0 0 | + | 9 10 2 0 0 0 0 |
| − | 10 4 1 0 0 0 0 | + | 10 4 1 0 0 0 0 |
| − | 8 11 1 0 0 0 0 | + | 8 11 1 0 0 0 0 |
| − | 10 12 1 0 0 0 0 | + | 10 12 1 0 0 0 0 |
| − | 5 13 2 0 0 0 0 | + | 5 13 2 0 0 0 0 |
| − | 1 14 1 0 0 0 0 | + | 1 14 1 0 0 0 0 |
| − | 14 15 2 0 0 0 0 | + | 14 15 2 0 0 0 0 |
| − | 15 16 1 0 0 0 0 | + | 15 16 1 0 0 0 0 |
| − | 16 17 2 0 0 0 0 | + | 16 17 2 0 0 0 0 |
| − | 17 18 1 0 0 0 0 | + | 17 18 1 0 0 0 0 |
| − | 18 19 2 0 0 0 0 | + | 18 19 2 0 0 0 0 |
| − | 19 14 1 0 0 0 0 | + | 19 14 1 0 0 0 0 |
| − | 17 20 1 0 0 0 0 | + | 17 20 1 0 0 0 0 |
| − | 21 22 1 1 0 0 0 | + | 21 22 1 1 0 0 0 |
| − | 22 23 1 1 0 0 0 | + | 22 23 1 1 0 0 0 |
| − | 24 23 1 1 0 0 0 | + | 24 23 1 1 0 0 0 |
| − | 24 25 1 0 0 0 0 | + | 24 25 1 0 0 0 0 |
| − | 25 26 1 0 0 0 0 | + | 25 26 1 0 0 0 0 |
| − | 26 21 1 0 0 0 0 | + | 26 21 1 0 0 0 0 |
| − | 26 27 1 0 0 0 0 | + | 26 27 1 0 0 0 0 |
| − | 27 28 1 0 0 0 0 | + | 27 28 1 0 0 0 0 |
| − | 21 29 1 0 0 0 0 | + | 21 29 1 0 0 0 0 |
| − | 22 30 1 0 0 0 0 | + | 22 30 1 0 0 0 0 |
| − | 23 31 1 0 0 0 0 | + | 23 31 1 0 0 0 0 |
| − | 7 24 1 0 0 0 0 | + | 7 24 1 0 0 0 0 |
| − | 32 33 1 1 0 0 0 | + | 32 33 1 1 0 0 0 |
| − | 33 34 1 1 0 0 0 | + | 33 34 1 1 0 0 0 |
| − | 35 34 1 1 0 0 0 | + | 35 34 1 1 0 0 0 |
| − | 35 36 1 0 0 0 0 | + | 35 36 1 0 0 0 0 |
| − | 36 37 1 0 0 0 0 | + | 36 37 1 0 0 0 0 |
| − | 37 32 1 0 0 0 0 | + | 37 32 1 0 0 0 0 |
| − | 37 38 1 0 0 0 0 | + | 37 38 1 0 0 0 0 |
| − | 38 39 1 0 0 0 0 | + | 38 39 1 0 0 0 0 |
| − | 32 40 1 0 0 0 0 | + | 32 40 1 0 0 0 0 |
| − | 33 41 1 0 0 0 0 | + | 33 41 1 0 0 0 0 |
| − | 34 42 1 0 0 0 0 | + | 34 42 1 0 0 0 0 |
| − | 35 28 1 0 0 0 0 | + | 35 28 1 0 0 0 0 |
| − | M STY 1 1 SUP | + | M STY 1 1 SUP |
| − | M SLB 1 1 1 | + | M SLB 1 1 1 |
| − | M SAL 1 2 38 39 | + | M SAL 1 2 38 39 |
| − | M SBL 1 1 41 | + | M SBL 1 1 41 |
| − | M SMT 1 CH2OH | + | M SMT 1 CH2OH |
| − | M SVB 1 41 -3.4977 2.9818 | + | M SVB 1 41 -3.4977 2.9818 |
| − | S SKP 8 | + | S SKP 8 |
| − | ID FL3FAACS0074 | + | ID FL3FAACS0074 |
| − | KNApSAcK_ID C00014042 | + | KNApSAcK_ID C00014042 |
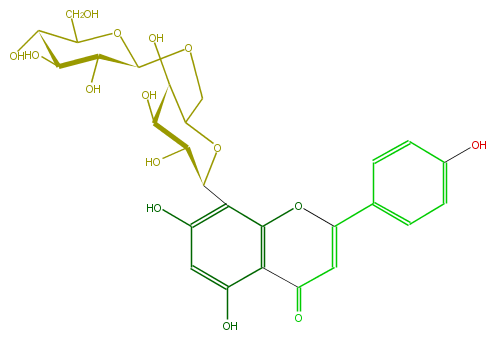
| − | NAME Vitexin 6''-O-glucoside;8-(6-O-beta-D-Glucopyranosyl-beta-D-glucopyranosyl)-5,7-dihydroxy-2-(4-hydroxyphenyl)-4H-1-benzopyran-4-one | + | NAME Vitexin 6''-O-glucoside;8-(6-O-beta-D-Glucopyranosyl-beta-D-glucopyranosyl)-5,7-dihydroxy-2-(4-hydroxyphenyl)-4H-1-benzopyran-4-one |
| − | CAS_RN 639850-16-1 | + | CAS_RN 639850-16-1 |
| − | FORMULA C27H30O15 | + | FORMULA C27H30O15 |
| − | EXACTMASS 594.15847029 | + | EXACTMASS 594.15847029 |
| − | AVERAGEMASS 594.5181 | + | AVERAGEMASS 594.5181 |
| − | SMILES [C@@H]([C@@H](O)1)([C@H](OCC(O2)[C@@H]([C@H](O)[C@H](O)[C@H](c(c35)c(cc(c3C(=O)C=C(O5)c(c4)ccc(O)c4)O)O)2)O)OC([C@@H]1O)CO)O | + | SMILES [C@@H]([C@@H](O)1)([C@H](OCC(O2)[C@@H]([C@H](O)[C@H](O)[C@H](c(c35)c(cc(c3C(=O)C=C(O5)c(c4)ccc(O)c4)O)O)2)O)OC([C@@H]1O)CO)O |
M END | M END | ||
| − | |||
Latest revision as of 09:00, 14 March 2009
Copyright: ARM project http://www.metabolome.jp/
42 46 0 0 0 0 0 0 0 0999 V2000
1.7915 -1.2818 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.0770 -0.8693 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
0.3625 -1.2818 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.3625 -2.1068 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.0770 -2.5193 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.7915 -2.1068 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.3520 -0.8693 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.0664 -1.2818 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.0664 -2.1068 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.3520 -2.5193 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.7375 -0.8944 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-0.3520 -3.2588 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
1.0770 -3.0914 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
2.5732 -0.8305 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.2877 -1.2430 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
4.0022 -0.8305 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
4.0022 -0.0055 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.2877 0.4070 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.5732 -0.0055 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
4.6174 0.3497 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-1.5168 1.5446 0.0000 C 0 0 2 0 0 0 0 0 0 0 0 0
-1.8134 0.7744 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0
-1.1489 0.2849 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0
-0.8267 -0.4748 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0
-0.5300 0.2953 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-1.1945 0.7849 0.0000 C 0 0 3 0 0 0 0 0 0 0 0 0
-0.8375 1.2752 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.1115 2.2980 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-1.8402 2.4909 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-1.9765 1.3523 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-1.7545 0.0161 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-4.1342 2.6227 0.0000 C 0 0 2 0 0 0 0 0 0 0 0 0
-3.7215 1.9080 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0
-2.9232 2.1171 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0
-2.1297 1.8903 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0
-2.5423 2.6050 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-3.3407 2.3960 0.0000 C 0 0 3 0 0 0 0 0 0 0 0 0
-3.4977 2.9818 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-3.9775 3.2588 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-4.6174 2.1395 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-4.1702 2.1670 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-3.0947 1.4772 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
1 2 1 0 0 0 0
2 3 1 0 0 0 0
3 4 2 0 0 0 0
4 5 1 0 0 0 0
5 6 1 0 0 0 0
6 1 2 0 0 0 0
3 7 1 0 0 0 0
7 8 2 0 0 0 0
8 9 1 0 0 0 0
9 10 2 0 0 0 0
10 4 1 0 0 0 0
8 11 1 0 0 0 0
10 12 1 0 0 0 0
5 13 2 0 0 0 0
1 14 1 0 0 0 0
14 15 2 0 0 0 0
15 16 1 0 0 0 0
16 17 2 0 0 0 0
17 18 1 0 0 0 0
18 19 2 0 0 0 0
19 14 1 0 0 0 0
17 20 1 0 0 0 0
21 22 1 1 0 0 0
22 23 1 1 0 0 0
24 23 1 1 0 0 0
24 25 1 0 0 0 0
25 26 1 0 0 0 0
26 21 1 0 0 0 0
26 27 1 0 0 0 0
27 28 1 0 0 0 0
21 29 1 0 0 0 0
22 30 1 0 0 0 0
23 31 1 0 0 0 0
7 24 1 0 0 0 0
32 33 1 1 0 0 0
33 34 1 1 0 0 0
35 34 1 1 0 0 0
35 36 1 0 0 0 0
36 37 1 0 0 0 0
37 32 1 0 0 0 0
37 38 1 0 0 0 0
38 39 1 0 0 0 0
32 40 1 0 0 0 0
33 41 1 0 0 0 0
34 42 1 0 0 0 0
35 28 1 0 0 0 0
M STY 1 1 SUP
M SLB 1 1 1
M SAL 1 2 38 39
M SBL 1 1 41
M SMT 1 CH2OH
M SVB 1 41 -3.4977 2.9818
S SKP 8
ID FL3FAACS0074
KNApSAcK_ID C00014042
NAME Vitexin 6''-O-glucoside;8-(6-O-beta-D-Glucopyranosyl-beta-D-glucopyranosyl)-5,7-dihydroxy-2-(4-hydroxyphenyl)-4H-1-benzopyran-4-one
CAS_RN 639850-16-1
FORMULA C27H30O15
EXACTMASS 594.15847029
AVERAGEMASS 594.5181
SMILES [C@@H]([C@@H](O)1)([C@H](OCC(O2)[C@@H]([C@H](O)[C@H](O)[C@H](c(c35)c(cc(c3C(=O)C=C(O5)c(c4)ccc(O)c4)O)O)2)O)OC([C@@H]1O)CO)O
M END