Mol:FL3FAACS0064
From Metabolomics.JP
(Difference between revisions)
| Line 1: | Line 1: | ||
| − | + | ||
| − | + | ||
| − | Copyright: ARM project http://www.metabolome.jp/ | + | Copyright: ARM project http://www.metabolome.jp/ |
| − | 42 46 0 0 0 0 0 0 0 0999 V2000 | + | 42 46 0 0 0 0 0 0 0 0999 V2000 |
| − | -0.5802 1.1718 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.5802 1.1718 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.0864 1.5566 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.0864 1.5566 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.0864 2.3263 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.0864 2.3263 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.5802 2.7112 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.5802 2.7112 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.2468 2.3263 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.2468 2.3263 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.2468 1.5566 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.2468 1.5566 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.7529 1.1718 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.7529 1.1718 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.4195 1.5566 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.4195 1.5566 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.4195 2.3263 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.4195 2.3263 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.7529 2.7112 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.7529 2.7112 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.0579 1.1880 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.0579 1.1880 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.0579 0.4574 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.0579 0.4574 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.6907 0.0921 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.6907 0.0921 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.3234 0.4574 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.3234 0.4574 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.3234 1.1880 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.3234 1.1880 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.6907 1.5534 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.6907 1.5534 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.7580 0.2065 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.7580 0.2065 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.7529 3.1687 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.7529 3.1687 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.5802 3.3170 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.5802 3.3170 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.7745 1.2519 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.7745 1.2519 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.2561 0.4788 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.2561 0.4788 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.7163 -0.1451 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.7163 -0.1451 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.9720 0.2107 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.9720 0.2107 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.1502 0.1375 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.1502 0.1375 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.6900 0.7615 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.6900 0.7615 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.4344 0.4057 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.4344 0.4057 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.7580 -0.0231 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.7580 -0.0231 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.3309 0.0195 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.3309 0.0195 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.6563 -0.3361 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.6563 -0.3361 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.7573 0.9651 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.7573 0.9651 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.1576 1.1961 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.1576 1.1961 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.1540 -0.8383 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.1540 -0.8383 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.5865 -0.5107 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.5865 -0.5107 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.1540 -1.4187 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.1540 -1.4187 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.7397 -1.7568 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.7397 -1.7568 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.7397 -2.4331 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.7397 -2.4331 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.1540 -2.7712 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.1540 -2.7712 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.5684 -2.4331 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.5684 -2.4331 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.5684 -1.7568 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.5684 -1.7568 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.1540 -3.3170 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.1540 -3.3170 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.2326 -2.7177 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.2326 -2.7177 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.9730 -2.7177 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.9730 -2.7177 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1 2 2 0 0 0 0 | + | 1 2 2 0 0 0 0 |
| − | 2 3 1 0 0 0 0 | + | 2 3 1 0 0 0 0 |
| − | 3 4 2 0 0 0 0 | + | 3 4 2 0 0 0 0 |
| − | 4 5 1 0 0 0 0 | + | 4 5 1 0 0 0 0 |
| − | 5 6 2 0 0 0 0 | + | 5 6 2 0 0 0 0 |
| − | 6 1 1 0 0 0 0 | + | 6 1 1 0 0 0 0 |
| − | 2 7 1 0 0 0 0 | + | 2 7 1 0 0 0 0 |
| − | 7 8 1 0 0 0 0 | + | 7 8 1 0 0 0 0 |
| − | 8 9 2 0 0 0 0 | + | 8 9 2 0 0 0 0 |
| − | 9 10 1 0 0 0 0 | + | 9 10 1 0 0 0 0 |
| − | 10 3 1 0 0 0 0 | + | 10 3 1 0 0 0 0 |
| − | 8 11 1 0 0 0 0 | + | 8 11 1 0 0 0 0 |
| − | 11 12 2 0 0 0 0 | + | 11 12 2 0 0 0 0 |
| − | 12 13 1 0 0 0 0 | + | 12 13 1 0 0 0 0 |
| − | 13 14 2 0 0 0 0 | + | 13 14 2 0 0 0 0 |
| − | 14 15 1 0 0 0 0 | + | 14 15 1 0 0 0 0 |
| − | 15 16 2 0 0 0 0 | + | 15 16 2 0 0 0 0 |
| − | 16 11 1 0 0 0 0 | + | 16 11 1 0 0 0 0 |
| − | 14 17 1 0 0 0 0 | + | 14 17 1 0 0 0 0 |
| − | 10 18 2 0 0 0 0 | + | 10 18 2 0 0 0 0 |
| − | 4 19 1 0 0 0 0 | + | 4 19 1 0 0 0 0 |
| − | 6 20 1 0 0 0 0 | + | 6 20 1 0 0 0 0 |
| − | 21 22 1 1 0 0 0 | + | 21 22 1 1 0 0 0 |
| − | 22 23 1 1 0 0 0 | + | 22 23 1 1 0 0 0 |
| − | 24 23 1 1 0 0 0 | + | 24 23 1 1 0 0 0 |
| − | 24 25 1 0 0 0 0 | + | 24 25 1 0 0 0 0 |
| − | 25 26 1 0 0 0 0 | + | 25 26 1 0 0 0 0 |
| − | 26 21 1 0 0 0 0 | + | 26 21 1 0 0 0 0 |
| − | 1 24 1 0 0 0 0 | + | 1 24 1 0 0 0 0 |
| − | 21 27 1 0 0 0 0 | + | 21 27 1 0 0 0 0 |
| − | 22 28 1 0 0 0 0 | + | 22 28 1 0 0 0 0 |
| − | 23 29 1 0 0 0 0 | + | 23 29 1 0 0 0 0 |
| − | 26 30 1 0 0 0 0 | + | 26 30 1 0 0 0 0 |
| − | 30 31 1 0 0 0 0 | + | 30 31 1 0 0 0 0 |
| − | 29 32 1 0 0 0 0 | + | 29 32 1 0 0 0 0 |
| − | 32 33 2 0 0 0 0 | + | 32 33 2 0 0 0 0 |
| − | 32 34 1 0 0 0 0 | + | 32 34 1 0 0 0 0 |
| − | 34 35 2 0 0 0 0 | + | 34 35 2 0 0 0 0 |
| − | 35 36 1 0 0 0 0 | + | 35 36 1 0 0 0 0 |
| − | 36 37 2 0 0 0 0 | + | 36 37 2 0 0 0 0 |
| − | 37 38 1 0 0 0 0 | + | 37 38 1 0 0 0 0 |
| − | 38 39 2 0 0 0 0 | + | 38 39 2 0 0 0 0 |
| − | 39 34 1 0 0 0 0 | + | 39 34 1 0 0 0 0 |
| − | 37 40 1 0 0 0 0 | + | 37 40 1 0 0 0 0 |
| − | 36 41 1 0 0 0 0 | + | 36 41 1 0 0 0 0 |
| − | 41 42 1 0 0 0 0 | + | 41 42 1 0 0 0 0 |
| − | S SKP 8 | + | S SKP 8 |
| − | ID FL3FAACS0064 | + | ID FL3FAACS0064 |
| − | KNApSAcK_ID C00011121 | + | KNApSAcK_ID C00011121 |
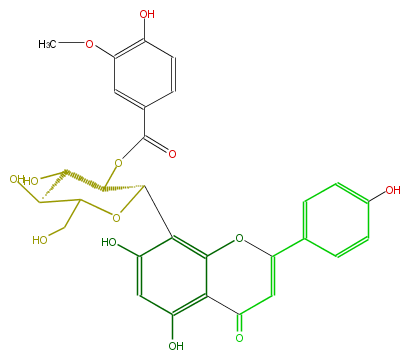
| − | NAME 2''-O-Vanilloylvitexin | + | NAME 2''-O-Vanilloylvitexin |
| − | CAS_RN 861691-31-8 | + | CAS_RN 861691-31-8 |
| − | FORMULA C29H26O13 | + | FORMULA C29H26O13 |
| − | EXACTMASS 582.137340918 | + | EXACTMASS 582.137340918 |
| − | AVERAGEMASS 582.5089399999999 | + | AVERAGEMASS 582.5089399999999 |
| − | SMILES c(c1)(c(O)ccc(C(=O)OC(C2c(c(O)5)c(O3)c(c(c5)O)C(=O)C=C3c(c4)ccc(c4)O)C(C(O)C(CO)O2)O)1)OC | + | SMILES c(c1)(c(O)ccc(C(=O)OC(C2c(c(O)5)c(O3)c(c(c5)O)C(=O)C=C3c(c4)ccc(c4)O)C(C(O)C(CO)O2)O)1)OC |
M END | M END | ||
| − | |||
Latest revision as of 09:00, 14 March 2009
Copyright: ARM project http://www.metabolome.jp/
42 46 0 0 0 0 0 0 0 0999 V2000
-0.5802 1.1718 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.0864 1.5566 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.0864 2.3263 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.5802 2.7112 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.2468 2.3263 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.2468 1.5566 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.7529 1.1718 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
1.4195 1.5566 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.4195 2.3263 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.7529 2.7112 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.0579 1.1880 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.0579 0.4574 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.6907 0.0921 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.3234 0.4574 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.3234 1.1880 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.6907 1.5534 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.7580 0.2065 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
0.7529 3.1687 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-0.5802 3.3170 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-1.7745 1.2519 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-3.2561 0.4788 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.7163 -0.1451 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.9720 0.2107 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.1502 0.1375 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.6900 0.7615 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-2.4344 0.4057 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-3.7580 -0.0231 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-3.3309 0.0195 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-1.6563 -0.3361 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-2.7573 0.9651 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-3.1576 1.1961 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-1.1540 -0.8383 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.5865 -0.5107 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-1.1540 -1.4187 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.7397 -1.7568 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.7397 -2.4331 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.1540 -2.7712 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.5684 -2.4331 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.5684 -1.7568 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.1540 -3.3170 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-2.2326 -2.7177 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-2.9730 -2.7177 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1 2 2 0 0 0 0
2 3 1 0 0 0 0
3 4 2 0 0 0 0
4 5 1 0 0 0 0
5 6 2 0 0 0 0
6 1 1 0 0 0 0
2 7 1 0 0 0 0
7 8 1 0 0 0 0
8 9 2 0 0 0 0
9 10 1 0 0 0 0
10 3 1 0 0 0 0
8 11 1 0 0 0 0
11 12 2 0 0 0 0
12 13 1 0 0 0 0
13 14 2 0 0 0 0
14 15 1 0 0 0 0
15 16 2 0 0 0 0
16 11 1 0 0 0 0
14 17 1 0 0 0 0
10 18 2 0 0 0 0
4 19 1 0 0 0 0
6 20 1 0 0 0 0
21 22 1 1 0 0 0
22 23 1 1 0 0 0
24 23 1 1 0 0 0
24 25 1 0 0 0 0
25 26 1 0 0 0 0
26 21 1 0 0 0 0
1 24 1 0 0 0 0
21 27 1 0 0 0 0
22 28 1 0 0 0 0
23 29 1 0 0 0 0
26 30 1 0 0 0 0
30 31 1 0 0 0 0
29 32 1 0 0 0 0
32 33 2 0 0 0 0
32 34 1 0 0 0 0
34 35 2 0 0 0 0
35 36 1 0 0 0 0
36 37 2 0 0 0 0
37 38 1 0 0 0 0
38 39 2 0 0 0 0
39 34 1 0 0 0 0
37 40 1 0 0 0 0
36 41 1 0 0 0 0
41 42 1 0 0 0 0
S SKP 8
ID FL3FAACS0064
KNApSAcK_ID C00011121
NAME 2''-O-Vanilloylvitexin
CAS_RN 861691-31-8
FORMULA C29H26O13
EXACTMASS 582.137340918
AVERAGEMASS 582.5089399999999
SMILES c(c1)(c(O)ccc(C(=O)OC(C2c(c(O)5)c(O3)c(c(c5)O)C(=O)C=C3c(c4)ccc(c4)O)C(C(O)C(CO)O2)O)1)OC
M END