Mol:FL3FAACS0044
From Metabolomics.JP
(Difference between revisions)
| Line 1: | Line 1: | ||
| − | + | ||
| − | + | ||
| − | Copyright: ARM project http://www.metabolome.jp/ | + | Copyright: ARM project http://www.metabolome.jp/ |
| − | 34 37 0 0 0 0 0 0 0 0999 V2000 | + | 34 37 0 0 0 0 0 0 0 0999 V2000 |
| − | -1.6712 -0.8357 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.6712 -0.8357 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.6712 -1.4781 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.6712 -1.4781 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.1149 -1.7993 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.1149 -1.7993 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.5586 -1.4781 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.5586 -1.4781 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.5586 -0.8357 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.5586 -0.8357 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.1149 -0.5145 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.1149 -0.5145 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.0023 -1.7993 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.0023 -1.7993 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.5540 -1.4781 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.5540 -1.4781 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.5540 -0.8357 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.5540 -0.8357 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.0023 -0.5145 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.0023 -0.5145 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.0023 -2.3001 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.0023 -2.3001 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.2273 -0.5147 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.2273 -0.5147 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.1149 -2.4414 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.1149 -2.4414 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.1719 -0.4940 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.1719 -0.4940 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.7581 -0.8325 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.7581 -0.8325 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.3444 -0.4940 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.3444 -0.4940 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.3444 0.1829 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.3444 0.1829 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.7581 0.5213 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.7581 0.5213 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.1719 0.1829 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.1719 0.1829 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.9302 0.5211 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.9302 0.5211 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.7664 1.8193 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.7664 1.8193 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.3720 1.3605 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.3720 1.3605 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.1151 0.6999 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.1151 0.6999 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.1082 0.0624 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.1082 0.0624 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.6449 0.5256 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.6449 0.5256 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.9499 1.1036 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.9499 1.1036 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.9331 2.4414 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.9331 2.4414 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.4068 2.0757 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.4068 2.0757 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.7505 0.3214 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.7505 0.3214 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.5088 0.5245 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.5088 0.5245 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.5088 1.1238 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.5088 1.1238 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.9302 0.2813 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.9302 0.2813 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.4443 1.3956 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.4443 1.3956 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.2702 0.9831 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.2702 0.9831 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1 2 1 0 0 0 0 | + | 1 2 1 0 0 0 0 |
| − | 2 3 2 0 0 0 0 | + | 2 3 2 0 0 0 0 |
| − | 3 4 1 0 0 0 0 | + | 3 4 1 0 0 0 0 |
| − | 4 5 2 0 0 0 0 | + | 4 5 2 0 0 0 0 |
| − | 5 6 1 0 0 0 0 | + | 5 6 1 0 0 0 0 |
| − | 6 1 2 0 0 0 0 | + | 6 1 2 0 0 0 0 |
| − | 4 7 1 0 0 0 0 | + | 4 7 1 0 0 0 0 |
| − | 7 8 1 0 0 0 0 | + | 7 8 1 0 0 0 0 |
| − | 8 9 2 0 0 0 0 | + | 8 9 2 0 0 0 0 |
| − | 9 10 1 0 0 0 0 | + | 9 10 1 0 0 0 0 |
| − | 10 5 1 0 0 0 0 | + | 10 5 1 0 0 0 0 |
| − | 7 11 2 0 0 0 0 | + | 7 11 2 0 0 0 0 |
| − | 1 12 1 0 0 0 0 | + | 1 12 1 0 0 0 0 |
| − | 3 13 1 0 0 0 0 | + | 3 13 1 0 0 0 0 |
| − | 14 15 2 0 0 0 0 | + | 14 15 2 0 0 0 0 |
| − | 15 16 1 0 0 0 0 | + | 15 16 1 0 0 0 0 |
| − | 16 17 2 0 0 0 0 | + | 16 17 2 0 0 0 0 |
| − | 17 18 1 0 0 0 0 | + | 17 18 1 0 0 0 0 |
| − | 18 19 2 0 0 0 0 | + | 18 19 2 0 0 0 0 |
| − | 19 14 1 0 0 0 0 | + | 19 14 1 0 0 0 0 |
| − | 14 9 1 0 0 0 0 | + | 14 9 1 0 0 0 0 |
| − | 17 20 1 0 0 0 0 | + | 17 20 1 0 0 0 0 |
| − | 21 22 1 1 0 0 0 | + | 21 22 1 1 0 0 0 |
| − | 22 23 1 1 0 0 0 | + | 22 23 1 1 0 0 0 |
| − | 24 23 1 1 0 0 0 | + | 24 23 1 1 0 0 0 |
| − | 24 25 1 0 0 0 0 | + | 24 25 1 0 0 0 0 |
| − | 25 26 1 0 0 0 0 | + | 25 26 1 0 0 0 0 |
| − | 26 21 1 0 0 0 0 | + | 26 21 1 0 0 0 0 |
| − | 21 27 1 0 0 0 0 | + | 21 27 1 0 0 0 0 |
| − | 22 28 1 0 0 0 0 | + | 22 28 1 0 0 0 0 |
| − | 23 29 1 0 0 0 0 | + | 23 29 1 0 0 0 0 |
| − | 6 24 1 0 0 0 0 | + | 6 24 1 0 0 0 0 |
| − | 29 30 1 0 0 0 0 | + | 29 30 1 0 0 0 0 |
| − | 30 31 1 0 0 0 0 | + | 30 31 1 0 0 0 0 |
| − | 30 32 2 0 0 0 0 | + | 30 32 2 0 0 0 0 |
| − | 26 33 1 0 0 0 0 | + | 26 33 1 0 0 0 0 |
| − | 33 34 1 0 0 0 0 | + | 33 34 1 0 0 0 0 |
| − | M STY 1 1 SUP | + | M STY 1 1 SUP |
| − | M SLB 1 1 1 | + | M SLB 1 1 1 |
| − | M SAL 1 2 33 34 | + | M SAL 1 2 33 34 |
| − | M SBL 1 1 36 | + | M SBL 1 1 36 |
| − | M SMT 1 CH2OH | + | M SMT 1 CH2OH |
| − | M SBV 1 36 -6.8127 5.7675 | + | M SBV 1 36 -6.8127 5.7675 |
| − | S SKP 8 | + | S SKP 8 |
| − | ID FL3FAACS0044 | + | ID FL3FAACS0044 |
| − | KNApSAcK_ID C00006369 | + | KNApSAcK_ID C00006369 |
| − | NAME Vitexin 2''-acetate | + | NAME Vitexin 2''-acetate |
| − | CAS_RN 264142-91-8 | + | CAS_RN 264142-91-8 |
| − | FORMULA C23H22O11 | + | FORMULA C23H22O11 |
| − | EXACTMASS 474.116211546 | + | EXACTMASS 474.116211546 |
| − | AVERAGEMASS 474.41418000000004 | + | AVERAGEMASS 474.41418000000004 |
| − | SMILES C(=C4)(Oc(c3C4=O)c(c(O)cc(O)3)C(C(OC(C)=O)2)OC(C(C(O)2)O)CO)c(c1)ccc(c1)O | + | SMILES C(=C4)(Oc(c3C4=O)c(c(O)cc(O)3)C(C(OC(C)=O)2)OC(C(C(O)2)O)CO)c(c1)ccc(c1)O |
M END | M END | ||
| − | |||
Latest revision as of 09:00, 14 March 2009
Copyright: ARM project http://www.metabolome.jp/
34 37 0 0 0 0 0 0 0 0999 V2000
-1.6712 -0.8357 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.6712 -1.4781 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.1149 -1.7993 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.5586 -1.4781 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.5586 -0.8357 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.1149 -0.5145 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.0023 -1.7993 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.5540 -1.4781 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.5540 -0.8357 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.0023 -0.5145 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-0.0023 -2.3001 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-2.2273 -0.5147 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-1.1149 -2.4414 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
1.1719 -0.4940 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.7581 -0.8325 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.3444 -0.4940 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.3444 0.1829 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.7581 0.5213 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.1719 0.1829 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.9302 0.5211 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-0.7664 1.8193 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.3720 1.3605 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.1151 0.6999 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.1082 0.0624 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.6449 0.5256 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-0.9499 1.1036 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.9331 2.4414 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-1.4068 2.0757 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-1.7505 0.3214 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-2.5088 0.5245 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.5088 1.1238 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.9302 0.2813 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-0.4443 1.3956 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.2702 0.9831 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
1 2 1 0 0 0 0
2 3 2 0 0 0 0
3 4 1 0 0 0 0
4 5 2 0 0 0 0
5 6 1 0 0 0 0
6 1 2 0 0 0 0
4 7 1 0 0 0 0
7 8 1 0 0 0 0
8 9 2 0 0 0 0
9 10 1 0 0 0 0
10 5 1 0 0 0 0
7 11 2 0 0 0 0
1 12 1 0 0 0 0
3 13 1 0 0 0 0
14 15 2 0 0 0 0
15 16 1 0 0 0 0
16 17 2 0 0 0 0
17 18 1 0 0 0 0
18 19 2 0 0 0 0
19 14 1 0 0 0 0
14 9 1 0 0 0 0
17 20 1 0 0 0 0
21 22 1 1 0 0 0
22 23 1 1 0 0 0
24 23 1 1 0 0 0
24 25 1 0 0 0 0
25 26 1 0 0 0 0
26 21 1 0 0 0 0
21 27 1 0 0 0 0
22 28 1 0 0 0 0
23 29 1 0 0 0 0
6 24 1 0 0 0 0
29 30 1 0 0 0 0
30 31 1 0 0 0 0
30 32 2 0 0 0 0
26 33 1 0 0 0 0
33 34 1 0 0 0 0
M STY 1 1 SUP
M SLB 1 1 1
M SAL 1 2 33 34
M SBL 1 1 36
M SMT 1 CH2OH
M SBV 1 36 -6.8127 5.7675
S SKP 8
ID FL3FAACS0044
KNApSAcK_ID C00006369
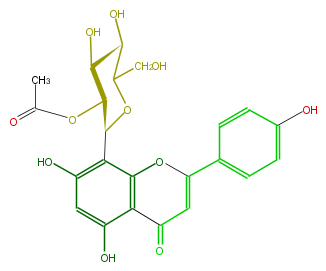
NAME Vitexin 2''-acetate
CAS_RN 264142-91-8
FORMULA C23H22O11
EXACTMASS 474.116211546
AVERAGEMASS 474.41418000000004
SMILES C(=C4)(Oc(c3C4=O)c(c(O)cc(O)3)C(C(OC(C)=O)2)OC(C(C(O)2)O)CO)c(c1)ccc(c1)O
M END