Mol:FL2FEGGS0001
From Metabolomics.JP
(Difference between revisions)
| Line 1: | Line 1: | ||
| − | + | ||
| − | + | ||
| − | Copyright: ARM project http://www.metabolome.jp/ | + | Copyright: ARM project http://www.metabolome.jp/ |
| − | 38 41 0 0 0 0 0 0 0 0999 V2000 | + | 38 41 0 0 0 0 0 0 0 0999 V2000 |
| − | -2.2798 0.3443 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.2798 0.3443 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.2798 -0.4778 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.2798 -0.4778 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.5677 -0.8890 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.5677 -0.8890 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.8556 -0.4778 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.8556 -0.4778 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.8556 0.3443 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.8556 0.3443 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.5677 0.7555 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.5677 0.7555 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.1435 -0.8890 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.1435 -0.8890 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.5686 -0.4778 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.5686 -0.4778 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.5686 0.3443 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.5686 0.3443 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.1435 0.7555 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.1435 0.7555 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.2804 0.7553 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.2804 0.7553 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.0060 0.3363 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.0060 0.3363 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.7318 0.7553 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.7318 0.7553 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.7318 1.5934 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.7318 1.5934 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.0060 2.0123 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.0060 2.0123 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.2804 1.5934 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.2804 1.5934 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.1435 -1.5851 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.1435 -1.5851 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.5677 -1.7110 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.5677 -1.7110 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.1674 -2.5724 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.1674 -2.5724 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.5957 -3.3141 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.5957 -3.3141 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.7721 -3.0787 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.7721 -3.0787 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.9436 -3.2958 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.9436 -3.2958 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.5153 -2.5542 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.5153 -2.5542 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.3389 -2.7895 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.3389 -2.7895 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.8743 -2.5978 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.8743 -2.5978 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -4.2714 -3.1120 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -4.2714 -3.1120 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.1514 -3.6363 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.1514 -3.6363 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.9423 -3.8930 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.9423 -3.8930 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.2174 0.2836 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.2174 0.2836 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 4.1320 -0.2444 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 4.1320 -0.2444 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.9626 0.7432 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.9626 0.7432 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.6026 1.8517 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.6026 1.8517 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.0017 2.7359 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.0017 2.7359 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.5491 3.8930 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.5491 3.8930 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.2436 2.0258 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.2436 2.0258 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 4.3521 2.6659 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 4.3521 2.6659 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.0898 -0.8407 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.0898 -0.8407 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -4.3521 -0.6280 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -4.3521 -0.6280 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1 2 1 0 0 0 0 | + | 1 2 1 0 0 0 0 |
| − | 2 3 2 0 0 0 0 | + | 2 3 2 0 0 0 0 |
| − | 3 4 1 0 0 0 0 | + | 3 4 1 0 0 0 0 |
| − | 4 5 2 0 0 0 0 | + | 4 5 2 0 0 0 0 |
| − | 5 6 1 0 0 0 0 | + | 5 6 1 0 0 0 0 |
| − | 6 1 2 0 0 0 0 | + | 6 1 2 0 0 0 0 |
| − | 4 7 1 0 0 0 0 | + | 4 7 1 0 0 0 0 |
| − | 7 8 1 0 0 0 0 | + | 7 8 1 0 0 0 0 |
| − | 8 9 1 0 0 0 0 | + | 8 9 1 0 0 0 0 |
| − | 9 10 1 0 0 0 0 | + | 9 10 1 0 0 0 0 |
| − | 10 5 1 0 0 0 0 | + | 10 5 1 0 0 0 0 |
| − | 9 11 1 0 0 0 0 | + | 9 11 1 0 0 0 0 |
| − | 11 12 2 0 0 0 0 | + | 11 12 2 0 0 0 0 |
| − | 12 13 1 0 0 0 0 | + | 12 13 1 0 0 0 0 |
| − | 13 14 2 0 0 0 0 | + | 13 14 2 0 0 0 0 |
| − | 14 15 1 0 0 0 0 | + | 14 15 1 0 0 0 0 |
| − | 15 16 2 0 0 0 0 | + | 15 16 2 0 0 0 0 |
| − | 16 11 1 0 0 0 0 | + | 16 11 1 0 0 0 0 |
| − | 7 17 2 0 0 0 0 | + | 7 17 2 0 0 0 0 |
| − | 3 18 1 0 0 0 0 | + | 3 18 1 0 0 0 0 |
| − | 19 20 1 0 0 0 0 | + | 19 20 1 0 0 0 0 |
| − | 20 21 1 1 0 0 0 | + | 20 21 1 1 0 0 0 |
| − | 22 21 1 1 0 0 0 | + | 22 21 1 1 0 0 0 |
| − | 23 22 1 1 0 0 0 | + | 23 22 1 1 0 0 0 |
| − | 23 24 1 0 0 0 0 | + | 23 24 1 0 0 0 0 |
| − | 24 19 1 0 0 0 0 | + | 24 19 1 0 0 0 0 |
| − | 18 23 1 0 0 0 0 | + | 18 23 1 0 0 0 0 |
| − | 19 25 1 0 0 0 0 | + | 19 25 1 0 0 0 0 |
| − | 20 26 1 0 0 0 0 | + | 20 26 1 0 0 0 0 |
| − | 21 27 1 0 0 0 0 | + | 21 27 1 0 0 0 0 |
| − | 22 28 1 0 0 0 0 | + | 22 28 1 0 0 0 0 |
| − | 29 30 1 0 0 0 0 | + | 29 30 1 0 0 0 0 |
| − | 13 29 1 0 0 0 0 | + | 13 29 1 0 0 0 0 |
| − | 31 32 1 0 0 0 0 | + | 31 32 1 0 0 0 0 |
| − | 1 31 1 0 0 0 0 | + | 1 31 1 0 0 0 0 |
| − | 33 34 1 0 0 0 0 | + | 33 34 1 0 0 0 0 |
| − | 15 33 1 0 0 0 0 | + | 15 33 1 0 0 0 0 |
| − | 35 36 1 0 0 0 0 | + | 35 36 1 0 0 0 0 |
| − | 14 35 1 0 0 0 0 | + | 14 35 1 0 0 0 0 |
| − | 37 38 1 0 0 0 0 | + | 37 38 1 0 0 0 0 |
| − | 2 37 1 0 0 0 0 | + | 2 37 1 0 0 0 0 |
| − | M STY 1 1 SUP | + | M STY 1 1 SUP |
| − | M SLB 1 1 1 | + | M SLB 1 1 1 |
| − | M SAL 1 2 29 30 | + | M SAL 1 2 29 30 |
| − | M SBL 1 1 33 | + | M SBL 1 1 33 |
| − | M SMT 1 OCH3 | + | M SMT 1 OCH3 |
| − | M SBV 1 33 -0.4856 0.4717 | + | M SBV 1 33 -0.4856 0.4717 |
| − | M STY 1 2 SUP | + | M STY 1 2 SUP |
| − | M SLB 1 2 2 | + | M SLB 1 2 2 |
| − | M SAL 2 2 31 32 | + | M SAL 2 2 31 32 |
| − | M SBL 2 1 35 | + | M SBL 2 1 35 |
| − | M SMT 2 ^ OCH3 | + | M SMT 2 ^ OCH3 |
| − | M SBV 2 35 0.6829 -0.3988 | + | M SBV 2 35 0.6829 -0.3988 |
| − | M STY 1 3 SUP | + | M STY 1 3 SUP |
| − | M SLB 1 3 3 | + | M SLB 1 3 3 |
| − | M SAL 3 2 33 34 | + | M SAL 3 2 33 34 |
| − | M SBL 3 1 37 | + | M SBL 3 1 37 |
| − | M SMT 3 OCH3 | + | M SMT 3 OCH3 |
| − | M SBV 3 37 0.0043 -0.7235 | + | M SBV 3 37 0.0043 -0.7235 |
| − | M STY 1 4 SUP | + | M STY 1 4 SUP |
| − | M SLB 1 4 4 | + | M SLB 1 4 4 |
| − | M SAL 4 2 35 36 | + | M SAL 4 2 35 36 |
| − | M SBL 4 1 39 | + | M SBL 4 1 39 |
| − | M SMT 4 OCH3 | + | M SMT 4 OCH3 |
| − | M SBV 4 39 -0.5118 -0.4324 | + | M SBV 4 39 -0.5118 -0.4324 |
| − | M STY 1 5 SUP | + | M STY 1 5 SUP |
| − | M SLB 1 5 5 | + | M SLB 1 5 5 |
| − | M SAL 5 2 37 38 | + | M SAL 5 2 37 38 |
| − | M SBL 5 1 41 | + | M SBL 5 1 41 |
| − | M SMT 5 ^ OCH3 | + | M SMT 5 ^ OCH3 |
| − | M SBV 5 41 0.8101 0.3629 | + | M SBV 5 41 0.8101 0.3629 |
| − | S SKP 5 | + | S SKP 5 |
| − | ID FL2FEGGS0001 | + | ID FL2FEGGS0001 |
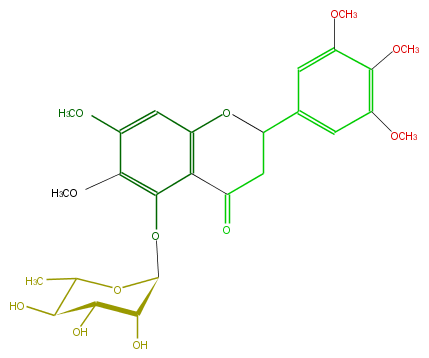
| − | FORMULA C26H32O12 | + | FORMULA C26H32O12 |
| − | EXACTMASS 536.189376488 | + | EXACTMASS 536.189376488 |
| − | AVERAGEMASS 536.5250799999999 | + | AVERAGEMASS 536.5250799999999 |
| − | SMILES c(c4)(cc(OC)c(OC)c4OC)C(C3)Oc(c1)c(C3=O)c(OC(C(O)2)OC(C)C(O)C2O)c(OC)c1OC | + | SMILES c(c4)(cc(OC)c(OC)c4OC)C(C3)Oc(c1)c(C3=O)c(OC(C(O)2)OC(C)C(O)C2O)c(OC)c1OC |
M END | M END | ||
| − | |||
Latest revision as of 09:00, 14 March 2009
Copyright: ARM project http://www.metabolome.jp/
38 41 0 0 0 0 0 0 0 0999 V2000
-2.2798 0.3443 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.2798 -0.4778 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.5677 -0.8890 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.8556 -0.4778 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.8556 0.3443 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.5677 0.7555 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.1435 -0.8890 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.5686 -0.4778 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.5686 0.3443 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.1435 0.7555 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
1.2804 0.7553 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.0060 0.3363 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.7318 0.7553 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.7318 1.5934 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.0060 2.0123 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.2804 1.5934 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.1435 -1.5851 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-1.5677 -1.7110 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-3.1674 -2.5724 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-3.5957 -3.3141 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.7721 -3.0787 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.9436 -3.2958 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.5153 -2.5542 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.3389 -2.7895 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-3.8743 -2.5978 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-4.2714 -3.1120 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-3.1514 -3.6363 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-1.9423 -3.8930 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
3.2174 0.2836 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
4.1320 -0.2444 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.9626 0.7432 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-3.6026 1.8517 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.0017 2.7359 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
2.5491 3.8930 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.2436 2.0258 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
4.3521 2.6659 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-3.0898 -0.8407 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-4.3521 -0.6280 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1 2 1 0 0 0 0
2 3 2 0 0 0 0
3 4 1 0 0 0 0
4 5 2 0 0 0 0
5 6 1 0 0 0 0
6 1 2 0 0 0 0
4 7 1 0 0 0 0
7 8 1 0 0 0 0
8 9 1 0 0 0 0
9 10 1 0 0 0 0
10 5 1 0 0 0 0
9 11 1 0 0 0 0
11 12 2 0 0 0 0
12 13 1 0 0 0 0
13 14 2 0 0 0 0
14 15 1 0 0 0 0
15 16 2 0 0 0 0
16 11 1 0 0 0 0
7 17 2 0 0 0 0
3 18 1 0 0 0 0
19 20 1 0 0 0 0
20 21 1 1 0 0 0
22 21 1 1 0 0 0
23 22 1 1 0 0 0
23 24 1 0 0 0 0
24 19 1 0 0 0 0
18 23 1 0 0 0 0
19 25 1 0 0 0 0
20 26 1 0 0 0 0
21 27 1 0 0 0 0
22 28 1 0 0 0 0
29 30 1 0 0 0 0
13 29 1 0 0 0 0
31 32 1 0 0 0 0
1 31 1 0 0 0 0
33 34 1 0 0 0 0
15 33 1 0 0 0 0
35 36 1 0 0 0 0
14 35 1 0 0 0 0
37 38 1 0 0 0 0
2 37 1 0 0 0 0
M STY 1 1 SUP
M SLB 1 1 1
M SAL 1 2 29 30
M SBL 1 1 33
M SMT 1 OCH3
M SBV 1 33 -0.4856 0.4717
M STY 1 2 SUP
M SLB 1 2 2
M SAL 2 2 31 32
M SBL 2 1 35
M SMT 2 ^ OCH3
M SBV 2 35 0.6829 -0.3988
M STY 1 3 SUP
M SLB 1 3 3
M SAL 3 2 33 34
M SBL 3 1 37
M SMT 3 OCH3
M SBV 3 37 0.0043 -0.7235
M STY 1 4 SUP
M SLB 1 4 4
M SAL 4 2 35 36
M SBL 4 1 39
M SMT 4 OCH3
M SBV 4 39 -0.5118 -0.4324
M STY 1 5 SUP
M SLB 1 5 5
M SAL 5 2 37 38
M SBL 5 1 41
M SMT 5 ^ OCH3
M SBV 5 41 0.8101 0.3629
S SKP 5
ID FL2FEGGS0001
FORMULA C26H32O12
EXACTMASS 536.189376488
AVERAGEMASS 536.5250799999999
SMILES c(c4)(cc(OC)c(OC)c4OC)C(C3)Oc(c1)c(C3=O)c(OC(C(O)2)OC(C)C(O)C2O)c(OC)c1OC
M END