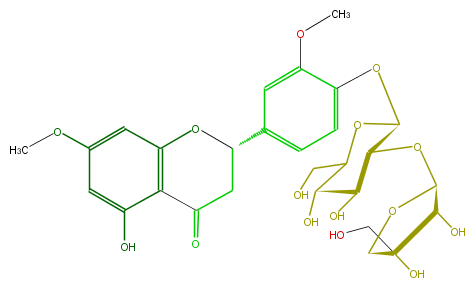
Mol:FL2FCDGS0002
From Metabolomics.JP
(Difference between revisions)
| Line 1: | Line 1: | ||
| − | + | ||
| − | + | ||
| − | Copyright: ARM project http://www.metabolome.jp/ | + | Copyright: ARM project http://www.metabolome.jp/ |
| − | 43 47 0 0 0 0 0 0 0 0999 V2000 | + | 43 47 0 0 0 0 0 0 0 0999 V2000 |
| − | -3.0217 -0.1395 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.0217 -0.1395 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.3090 0.2762 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.3090 0.2762 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.0185 -0.9645 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.0185 -0.9645 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.3017 -1.3738 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.3017 -1.3738 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.5891 -0.9582 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.5891 -0.9582 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.5927 -0.1332 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.5927 -0.1332 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.8728 -1.3675 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.8728 -1.3675 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.1602 -0.9518 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.1602 -0.9518 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.1638 -0.1268 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.1638 -0.1268 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.8801 0.2825 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.8801 0.2825 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.4900 0.2540 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.4900 0.2540 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.2060 -0.1558 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.2060 -0.1558 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.9189 0.2595 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.9189 0.2595 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.9157 1.0846 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.9157 1.0846 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.1996 1.4943 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.1996 1.4943 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.4868 1.0791 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.4868 1.0791 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.8717 -2.0107 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.8717 -2.0107 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.6471 0.2216 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.6471 0.2216 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.3017 -2.0820 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.3017 -2.0820 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.7244 1.5166 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.7244 1.5166 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -4.3048 -0.1581 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -4.3048 -0.1581 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.5186 -0.9657 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.5186 -0.9657 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.3436 -0.9835 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.3436 -0.9835 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.5788 -0.1925 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.5788 -0.1925 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.1843 0.3683 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.1843 0.3683 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.3592 0.3862 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.3592 0.3862 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.1239 -0.4048 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.1239 -0.4048 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.4663 -0.5047 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.4663 -0.5047 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.1602 -1.0349 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.1602 -1.0349 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.3590 -1.5612 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.3590 -1.5612 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.8854 -1.4418 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.8854 -1.4418 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.5494 -0.0509 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.5494 -0.0509 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.0842 -2.2041 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.0842 -2.2041 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.4779 -2.5978 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.4779 -2.5978 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 4.3048 -1.7501 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 4.3048 -1.7501 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.5731 -1.6701 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.5731 -1.6701 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.9270 -0.8521 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.9270 -0.8521 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.5714 -2.2077 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.5714 -2.2077 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.9215 -1.3668 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.9215 -1.3668 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.0668 -1.3466 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.0668 -1.3466 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.0220 -1.8178 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.0220 -1.8178 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.1996 2.1956 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.1996 2.1956 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.8963 2.5978 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.8963 2.5978 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1 2 2 0 0 0 0 | + | 1 2 2 0 0 0 0 |
| − | 1 3 1 0 0 0 0 | + | 1 3 1 0 0 0 0 |
| − | 3 4 2 0 0 0 0 | + | 3 4 2 0 0 0 0 |
| − | 4 5 1 0 0 0 0 | + | 4 5 1 0 0 0 0 |
| − | 5 6 2 0 0 0 0 | + | 5 6 2 0 0 0 0 |
| − | 6 2 1 0 0 0 0 | + | 6 2 1 0 0 0 0 |
| − | 5 7 1 0 0 0 0 | + | 5 7 1 0 0 0 0 |
| − | 7 8 1 0 0 0 0 | + | 7 8 1 0 0 0 0 |
| − | 8 9 1 0 0 0 0 | + | 8 9 1 0 0 0 0 |
| − | 9 10 1 0 0 0 0 | + | 9 10 1 0 0 0 0 |
| − | 10 6 1 0 0 0 0 | + | 10 6 1 0 0 0 0 |
| − | 9 11 1 6 0 0 0 | + | 9 11 1 6 0 0 0 |
| − | 11 12 2 0 0 0 0 | + | 11 12 2 0 0 0 0 |
| − | 12 13 1 0 0 0 0 | + | 12 13 1 0 0 0 0 |
| − | 13 14 2 0 0 0 0 | + | 13 14 2 0 0 0 0 |
| − | 14 15 1 0 0 0 0 | + | 14 15 1 0 0 0 0 |
| − | 15 16 2 0 0 0 0 | + | 15 16 2 0 0 0 0 |
| − | 16 11 1 0 0 0 0 | + | 16 11 1 0 0 0 0 |
| − | 7 17 2 0 0 0 0 | + | 7 17 2 0 0 0 0 |
| − | 1 18 1 0 0 0 0 | + | 1 18 1 0 0 0 0 |
| − | 4 19 1 0 0 0 0 | + | 4 19 1 0 0 0 0 |
| − | 14 20 1 0 0 0 0 | + | 14 20 1 0 0 0 0 |
| − | 18 21 1 0 0 0 0 | + | 18 21 1 0 0 0 0 |
| − | 22 23 1 1 0 0 0 | + | 22 23 1 1 0 0 0 |
| − | 23 24 1 1 0 0 0 | + | 23 24 1 1 0 0 0 |
| − | 25 24 1 1 0 0 0 | + | 25 24 1 1 0 0 0 |
| − | 25 26 1 0 0 0 0 | + | 25 26 1 0 0 0 0 |
| − | 26 27 1 0 0 0 0 | + | 26 27 1 0 0 0 0 |
| − | 27 22 1 0 0 0 0 | + | 27 22 1 0 0 0 0 |
| − | 27 28 1 0 0 0 0 | + | 27 28 1 0 0 0 0 |
| − | 28 29 1 0 0 0 0 | + | 28 29 1 0 0 0 0 |
| − | 22 30 1 0 0 0 0 | + | 22 30 1 0 0 0 0 |
| − | 23 31 1 0 0 0 0 | + | 23 31 1 0 0 0 0 |
| − | 24 32 1 0 0 0 0 | + | 24 32 1 0 0 0 0 |
| − | 39 33 1 1 0 0 0 | + | 39 33 1 1 0 0 0 |
| − | 38 33 1 1 0 0 0 | + | 38 33 1 1 0 0 0 |
| − | 37 39 1 1 0 0 0 | + | 37 39 1 1 0 0 0 |
| − | 33 34 1 0 0 0 0 | + | 33 34 1 0 0 0 0 |
| − | 39 35 1 0 0 0 0 | + | 39 35 1 0 0 0 0 |
| − | 40 37 1 0 0 0 0 | + | 40 37 1 0 0 0 0 |
| − | 36 41 1 0 0 0 0 | + | 36 41 1 0 0 0 0 |
| − | 38 40 1 0 0 0 0 | + | 38 40 1 0 0 0 0 |
| − | 33 36 1 0 0 0 0 | + | 33 36 1 0 0 0 0 |
| − | 25 20 1 0 0 0 0 | + | 25 20 1 0 0 0 0 |
| − | 15 42 1 0 0 0 0 | + | 15 42 1 0 0 0 0 |
| − | 42 43 1 0 0 0 0 | + | 42 43 1 0 0 0 0 |
| − | 32 37 1 0 0 0 0 | + | 32 37 1 0 0 0 0 |
| − | S SKP 5 | + | S SKP 5 |
| − | ID FL2FCDGS0002 | + | ID FL2FCDGS0002 |
| − | FORMULA C28H34O15 | + | FORMULA C28H34O15 |
| − | EXACTMASS 610.189770418 | + | EXACTMASS 610.189770418 |
| − | AVERAGEMASS 610.56056 | + | AVERAGEMASS 610.56056 |
| − | SMILES Oc(c5)c(C(=O)4)c(cc5OC)OC(C4)c(c1)cc(c(OC(C2OC(C(O)3)OCC3(CO)O)OC(CO)C(C(O)2)O)c1)OC | + | SMILES Oc(c5)c(C(=O)4)c(cc5OC)OC(C4)c(c1)cc(c(OC(C2OC(C(O)3)OCC3(CO)O)OC(CO)C(C(O)2)O)c1)OC |
M END | M END | ||
| − | |||
Latest revision as of 09:00, 14 March 2009
Copyright: ARM project http://www.metabolome.jp/
43 47 0 0 0 0 0 0 0 0999 V2000
-3.0217 -0.1395 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.3090 0.2762 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-3.0185 -0.9645 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.3017 -1.3738 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.5891 -0.9582 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.5927 -0.1332 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.8728 -1.3675 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.1602 -0.9518 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.1638 -0.1268 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.8801 0.2825 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
0.4900 0.2540 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.2060 -0.1558 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.9189 0.2595 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.9157 1.0846 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.1996 1.4943 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.4868 1.0791 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.8717 -2.0107 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-3.6471 0.2216 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-2.3017 -2.0820 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
2.7244 1.5166 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-4.3048 -0.1581 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.5186 -0.9657 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.3436 -0.9835 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.5788 -0.1925 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.1843 0.3683 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.3592 0.3862 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
2.1239 -0.4048 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.4663 -0.5047 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.1602 -1.0349 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
1.3590 -1.5612 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
1.8854 -1.4418 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
3.5494 -0.0509 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
3.0842 -2.2041 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.4779 -2.5978 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
4.3048 -1.7501 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
2.5731 -1.6701 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.9270 -0.8521 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.5714 -2.2077 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.9215 -1.3668 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.0668 -1.3466 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
2.0220 -1.8178 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
1.1996 2.1956 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
1.8963 2.5978 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1 2 2 0 0 0 0
1 3 1 0 0 0 0
3 4 2 0 0 0 0
4 5 1 0 0 0 0
5 6 2 0 0 0 0
6 2 1 0 0 0 0
5 7 1 0 0 0 0
7 8 1 0 0 0 0
8 9 1 0 0 0 0
9 10 1 0 0 0 0
10 6 1 0 0 0 0
9 11 1 6 0 0 0
11 12 2 0 0 0 0
12 13 1 0 0 0 0
13 14 2 0 0 0 0
14 15 1 0 0 0 0
15 16 2 0 0 0 0
16 11 1 0 0 0 0
7 17 2 0 0 0 0
1 18 1 0 0 0 0
4 19 1 0 0 0 0
14 20 1 0 0 0 0
18 21 1 0 0 0 0
22 23 1 1 0 0 0
23 24 1 1 0 0 0
25 24 1 1 0 0 0
25 26 1 0 0 0 0
26 27 1 0 0 0 0
27 22 1 0 0 0 0
27 28 1 0 0 0 0
28 29 1 0 0 0 0
22 30 1 0 0 0 0
23 31 1 0 0 0 0
24 32 1 0 0 0 0
39 33 1 1 0 0 0
38 33 1 1 0 0 0
37 39 1 1 0 0 0
33 34 1 0 0 0 0
39 35 1 0 0 0 0
40 37 1 0 0 0 0
36 41 1 0 0 0 0
38 40 1 0 0 0 0
33 36 1 0 0 0 0
25 20 1 0 0 0 0
15 42 1 0 0 0 0
42 43 1 0 0 0 0
32 37 1 0 0 0 0
S SKP 5
ID FL2FCDGS0002
FORMULA C28H34O15
EXACTMASS 610.189770418
AVERAGEMASS 610.56056
SMILES Oc(c5)c(C(=O)4)c(cc5OC)OC(C4)c(c1)cc(c(OC(C2OC(C(O)3)OCC3(CO)O)OC(CO)C(C(O)2)O)c1)OC
M END