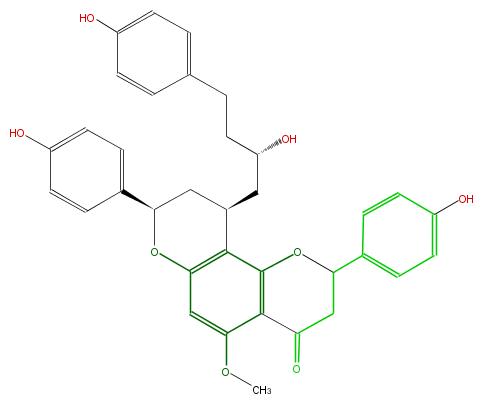
Mol:FL2FBANC0012
From Metabolomics.JP
(Difference between revisions)
| Line 1: | Line 1: | ||
| − | + | ||
| − | + | ||
| − | Copyright: ARM project http://www.metabolome.jp/ | + | Copyright: ARM project http://www.metabolome.jp/ |
| − | 43 48 0 0 0 0 0 0 0 0999 V2000 | + | 43 48 0 0 0 0 0 0 0 0999 V2000 |
| − | -0.3729 -2.6732 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.3729 -2.6732 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.0805 -2.2489 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.0805 -2.2489 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.3485 -2.2730 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.3485 -2.2730 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.3619 -1.4477 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.3619 -1.4477 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.3457 -1.0234 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.3457 -1.0234 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.0669 -1.4241 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.0669 -1.4241 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.3321 -0.1985 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.3321 -0.1985 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.0396 0.2257 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.0396 0.2257 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.7608 -0.1749 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.7608 -0.1749 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.7744 -0.9998 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.7744 -0.9998 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.4095 0.2145 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.4095 0.2145 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.3954 1.0394 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.3954 1.0394 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.1027 1.4640 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.1027 1.4640 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.8241 1.0638 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.8241 1.0638 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.8382 0.2389 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.8382 0.2389 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.1309 -0.1857 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.1309 -0.1857 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -4.4114 1.4164 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -4.4114 1.4164 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.3857 -3.4191 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.3857 -3.4191 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.2155 -3.7827 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.2155 -3.7827 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.0514 -2.7000 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.0514 -2.7000 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.7745 -2.3019 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.7745 -2.3019 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.7895 -1.4795 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.7895 -1.4795 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.0849 -1.0495 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.0849 -1.0495 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.0375 -3.3858 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.0375 -3.3858 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.2969 0.1478 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.2969 0.1478 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.3122 0.9035 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.3122 0.9035 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.8764 1.2142 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.8764 1.2142 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.3068 1.2778 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.3068 1.2778 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.2904 2.0838 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.2904 2.0838 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.0299 2.5309 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.0299 2.5309 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.0132 3.3558 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.0132 3.3558 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.7191 3.7827 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.7191 3.7827 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.4418 3.3847 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.4418 3.3847 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.4585 2.5599 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.4585 2.5599 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.7526 2.1330 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.7526 2.1330 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.9656 3.7015 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.9656 3.7015 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.3869 -1.1505 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.3869 -1.1505 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.0928 -1.5774 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.0928 -1.5774 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.8155 -1.1795 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.8155 -1.1795 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.8322 -0.3546 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.8322 -0.3546 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.1263 0.0722 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.1263 0.0722 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.4036 -0.3257 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.4036 -0.3257 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 4.4114 -0.0357 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 4.4114 -0.0357 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1 2 2 0 0 0 0 | + | 1 2 2 0 0 0 0 |
| − | 1 3 1 0 0 0 0 | + | 1 3 1 0 0 0 0 |
| − | 3 4 2 0 0 0 0 | + | 3 4 2 0 0 0 0 |
| − | 4 5 1 0 0 0 0 | + | 4 5 1 0 0 0 0 |
| − | 5 6 2 0 0 0 0 | + | 5 6 2 0 0 0 0 |
| − | 6 2 1 0 0 0 0 | + | 6 2 1 0 0 0 0 |
| − | 5 7 1 0 0 0 0 | + | 5 7 1 0 0 0 0 |
| − | 7 8 1 0 0 0 0 | + | 7 8 1 0 0 0 0 |
| − | 8 9 1 0 0 0 0 | + | 8 9 1 0 0 0 0 |
| − | 9 10 1 0 0 0 0 | + | 9 10 1 0 0 0 0 |
| − | 10 6 1 0 0 0 0 | + | 10 6 1 0 0 0 0 |
| − | 9 11 1 1 0 0 0 | + | 9 11 1 1 0 0 0 |
| − | 11 12 2 0 0 0 0 | + | 11 12 2 0 0 0 0 |
| − | 12 13 1 0 0 0 0 | + | 12 13 1 0 0 0 0 |
| − | 13 14 2 0 0 0 0 | + | 13 14 2 0 0 0 0 |
| − | 14 15 1 0 0 0 0 | + | 14 15 1 0 0 0 0 |
| − | 15 16 2 0 0 0 0 | + | 15 16 2 0 0 0 0 |
| − | 16 11 1 0 0 0 0 | + | 16 11 1 0 0 0 0 |
| − | 14 17 1 0 0 0 0 | + | 14 17 1 0 0 0 0 |
| − | 1 18 1 0 0 0 0 | + | 1 18 1 0 0 0 0 |
| − | 18 19 1 0 0 0 0 | + | 18 19 1 0 0 0 0 |
| − | 3 20 1 0 0 0 0 | + | 3 20 1 0 0 0 0 |
| − | 20 21 1 0 0 0 0 | + | 20 21 1 0 0 0 0 |
| − | 21 22 1 0 0 0 0 | + | 21 22 1 0 0 0 0 |
| − | 22 23 1 0 0 0 0 | + | 22 23 1 0 0 0 0 |
| − | 23 4 1 0 0 0 0 | + | 23 4 1 0 0 0 0 |
| − | 20 24 2 0 0 0 0 | + | 20 24 2 0 0 0 0 |
| − | 7 25 1 1 0 0 0 | + | 7 25 1 1 0 0 0 |
| − | 25 26 1 0 0 0 0 | + | 25 26 1 0 0 0 0 |
| − | 26 27 1 6 0 0 0 | + | 26 27 1 6 0 0 0 |
| − | 26 28 1 0 0 0 0 | + | 26 28 1 0 0 0 0 |
| − | 28 29 1 0 0 0 0 | + | 28 29 1 0 0 0 0 |
| − | 29 30 1 0 0 0 0 | + | 29 30 1 0 0 0 0 |
| − | 30 31 2 0 0 0 0 | + | 30 31 2 0 0 0 0 |
| − | 31 32 1 0 0 0 0 | + | 31 32 1 0 0 0 0 |
| − | 32 33 2 0 0 0 0 | + | 32 33 2 0 0 0 0 |
| − | 33 34 1 0 0 0 0 | + | 33 34 1 0 0 0 0 |
| − | 34 35 2 0 0 0 0 | + | 34 35 2 0 0 0 0 |
| − | 35 30 1 0 0 0 0 | + | 35 30 1 0 0 0 0 |
| − | 33 36 1 0 0 0 0 | + | 33 36 1 0 0 0 0 |
| − | 22 37 1 0 0 0 0 | + | 22 37 1 0 0 0 0 |
| − | 37 38 2 0 0 0 0 | + | 37 38 2 0 0 0 0 |
| − | 38 39 1 0 0 0 0 | + | 38 39 1 0 0 0 0 |
| − | 39 40 2 0 0 0 0 | + | 39 40 2 0 0 0 0 |
| − | 40 41 1 0 0 0 0 | + | 40 41 1 0 0 0 0 |
| − | 41 42 2 0 0 0 0 | + | 41 42 2 0 0 0 0 |
| − | 42 37 1 0 0 0 0 | + | 42 37 1 0 0 0 0 |
| − | 40 43 1 0 0 0 0 | + | 40 43 1 0 0 0 0 |
| − | S SKP 8 | + | S SKP 8 |
| − | ID FL2FBANC0012 | + | ID FL2FBANC0012 |
| − | KNApSAcK_ID C00014296 | + | KNApSAcK_ID C00014296 |
| − | NAME Epicalyxin M | + | NAME Epicalyxin M |
| − | CAS_RN 332396-26-6 | + | CAS_RN 332396-26-6 |
| − | FORMULA C35H34O8 | + | FORMULA C35H34O8 |
| − | EXACTMASS 582.225368064 | + | EXACTMASS 582.225368064 |
| − | AVERAGEMASS 582.63966 | + | AVERAGEMASS 582.63966 |
| − | SMILES O(c52)C(CC(CC(O)CCc(c6)ccc(c6)O)c2c(c(c(c5)OC)4)OC(CC(=O)4)c(c3)ccc(c3)O)c(c1)ccc(O)c1 | + | SMILES O(c52)C(CC(CC(O)CCc(c6)ccc(c6)O)c2c(c(c(c5)OC)4)OC(CC(=O)4)c(c3)ccc(c3)O)c(c1)ccc(O)c1 |
M END | M END | ||
| − | |||
Latest revision as of 09:00, 14 March 2009
Copyright: ARM project http://www.metabolome.jp/
43 48 0 0 0 0 0 0 0 0999 V2000
-0.3729 -2.6732 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.0805 -2.2489 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.3485 -2.2730 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.3619 -1.4477 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.3457 -1.0234 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.0669 -1.4241 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.3321 -0.1985 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.0396 0.2257 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.7608 -0.1749 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.7744 -0.9998 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-2.4095 0.2145 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.3954 1.0394 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-3.1027 1.4640 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-3.8241 1.0638 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-3.8382 0.2389 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-3.1309 -0.1857 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-4.4114 1.4164 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-0.3857 -3.4191 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
0.2155 -3.7827 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.0514 -2.7000 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.7745 -2.3019 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.7895 -1.4795 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.0849 -1.0495 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
1.0375 -3.3858 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
0.2969 0.1478 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.3122 0.9035 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.8764 1.2142 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-0.3068 1.2778 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.2904 2.0838 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.0299 2.5309 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.0132 3.3558 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.7191 3.7827 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.4418 3.3847 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.4585 2.5599 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.7526 2.1330 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.9656 3.7015 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
2.3869 -1.1505 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.0928 -1.5774 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.8155 -1.1795 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.8322 -0.3546 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.1263 0.0722 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.4036 -0.3257 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
4.4114 -0.0357 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
1 2 2 0 0 0 0
1 3 1 0 0 0 0
3 4 2 0 0 0 0
4 5 1 0 0 0 0
5 6 2 0 0 0 0
6 2 1 0 0 0 0
5 7 1 0 0 0 0
7 8 1 0 0 0 0
8 9 1 0 0 0 0
9 10 1 0 0 0 0
10 6 1 0 0 0 0
9 11 1 1 0 0 0
11 12 2 0 0 0 0
12 13 1 0 0 0 0
13 14 2 0 0 0 0
14 15 1 0 0 0 0
15 16 2 0 0 0 0
16 11 1 0 0 0 0
14 17 1 0 0 0 0
1 18 1 0 0 0 0
18 19 1 0 0 0 0
3 20 1 0 0 0 0
20 21 1 0 0 0 0
21 22 1 0 0 0 0
22 23 1 0 0 0 0
23 4 1 0 0 0 0
20 24 2 0 0 0 0
7 25 1 1 0 0 0
25 26 1 0 0 0 0
26 27 1 6 0 0 0
26 28 1 0 0 0 0
28 29 1 0 0 0 0
29 30 1 0 0 0 0
30 31 2 0 0 0 0
31 32 1 0 0 0 0
32 33 2 0 0 0 0
33 34 1 0 0 0 0
34 35 2 0 0 0 0
35 30 1 0 0 0 0
33 36 1 0 0 0 0
22 37 1 0 0 0 0
37 38 2 0 0 0 0
38 39 1 0 0 0 0
39 40 2 0 0 0 0
40 41 1 0 0 0 0
41 42 2 0 0 0 0
42 37 1 0 0 0 0
40 43 1 0 0 0 0
S SKP 8
ID FL2FBANC0012
KNApSAcK_ID C00014296
NAME Epicalyxin M
CAS_RN 332396-26-6
FORMULA C35H34O8
EXACTMASS 582.225368064
AVERAGEMASS 582.63966
SMILES O(c52)C(CC(CC(O)CCc(c6)ccc(c6)O)c2c(c(c(c5)OC)4)OC(CC(=O)4)c(c3)ccc(c3)O)c(c1)ccc(O)c1
M END