Mol:FL2FAIGS0002
From Metabolomics.JP
(Difference between revisions)
| Line 1: | Line 1: | ||
| − | + | ||
| − | + | ||
| − | Copyright: ARM project http://www.metabolome.jp/ | + | Copyright: ARM project http://www.metabolome.jp/ |
| − | 35 38 0 0 0 0 0 0 0 0999 V2000 | + | 35 38 0 0 0 0 0 0 0 0999 V2000 |
| − | -2.0500 0.1772 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.0500 0.1772 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.3373 0.5929 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.3373 0.5929 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.0468 -0.6478 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.0468 -0.6478 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.3300 -1.0571 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.3300 -1.0571 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.6174 -0.6414 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.6174 -0.6414 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.6210 0.1836 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.6210 0.1836 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.0989 -1.0508 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.0989 -1.0508 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.8115 -0.6351 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.8115 -0.6351 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.8079 0.1899 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.8079 0.1899 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.0916 0.5992 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.0916 0.5992 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.4617 0.5707 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.4617 0.5707 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.1777 0.1610 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.1777 0.1610 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.8906 0.5762 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.8906 0.5762 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.8874 1.4012 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.8874 1.4012 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.1714 1.8110 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.1714 1.8110 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.4585 1.3957 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.4585 1.3957 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.1000 -1.6940 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.1000 -1.6940 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.6754 0.5383 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.6754 0.5383 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.3300 -1.7652 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.3300 -1.7652 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.5185 1.7656 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.5185 1.7656 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.1714 2.5275 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.1714 2.5275 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.9260 2.9632 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.9260 2.9632 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.5670 0.1857 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.5670 0.1857 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 4.3784 0.6541 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 4.3784 0.6541 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.8952 -1.8177 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.8952 -1.8177 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.4826 -2.5324 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.4826 -2.5324 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.6842 -2.3233 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.6842 -2.3233 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.8907 -2.5501 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.8907 -2.5501 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.3033 -1.8354 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.3033 -1.8354 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.1017 -2.0444 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.1017 -2.0444 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.2587 -1.4586 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.2587 -1.4586 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.7385 -1.1816 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.7385 -1.1816 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -4.3784 -2.3009 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -4.3784 -2.3009 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.9312 -2.2734 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.9312 -2.2734 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.8557 -2.9632 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.8557 -2.9632 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1 2 2 0 0 0 0 | + | 1 2 2 0 0 0 0 |
| − | 1 3 1 0 0 0 0 | + | 1 3 1 0 0 0 0 |
| − | 3 4 2 0 0 0 0 | + | 3 4 2 0 0 0 0 |
| − | 4 5 1 0 0 0 0 | + | 4 5 1 0 0 0 0 |
| − | 5 6 2 0 0 0 0 | + | 5 6 2 0 0 0 0 |
| − | 6 2 1 0 0 0 0 | + | 6 2 1 0 0 0 0 |
| − | 5 7 1 0 0 0 0 | + | 5 7 1 0 0 0 0 |
| − | 7 8 1 0 0 0 0 | + | 7 8 1 0 0 0 0 |
| − | 8 9 1 0 0 0 0 | + | 8 9 1 0 0 0 0 |
| − | 9 10 1 0 0 0 0 | + | 9 10 1 0 0 0 0 |
| − | 10 6 1 0 0 0 0 | + | 10 6 1 0 0 0 0 |
| − | 9 11 1 6 0 0 0 | + | 9 11 1 6 0 0 0 |
| − | 11 12 2 0 0 0 0 | + | 11 12 2 0 0 0 0 |
| − | 12 13 1 0 0 0 0 | + | 12 13 1 0 0 0 0 |
| − | 13 14 2 0 0 0 0 | + | 13 14 2 0 0 0 0 |
| − | 14 15 1 0 0 0 0 | + | 14 15 1 0 0 0 0 |
| − | 15 16 2 0 0 0 0 | + | 15 16 2 0 0 0 0 |
| − | 16 11 1 0 0 0 0 | + | 16 11 1 0 0 0 0 |
| − | 7 17 2 0 0 0 0 | + | 7 17 2 0 0 0 0 |
| − | 1 18 1 0 0 0 0 | + | 1 18 1 0 0 0 0 |
| − | 4 19 1 0 0 0 0 | + | 4 19 1 0 0 0 0 |
| − | 14 20 1 0 0 0 0 | + | 14 20 1 0 0 0 0 |
| − | 15 21 1 0 0 0 0 | + | 15 21 1 0 0 0 0 |
| − | 21 22 1 0 0 0 0 | + | 21 22 1 0 0 0 0 |
| − | 13 23 1 0 0 0 0 | + | 13 23 1 0 0 0 0 |
| − | 23 24 1 0 0 0 0 | + | 23 24 1 0 0 0 0 |
| − | 25 26 1 1 0 0 0 | + | 25 26 1 1 0 0 0 |
| − | 26 27 1 1 0 0 0 | + | 26 27 1 1 0 0 0 |
| − | 28 27 1 1 0 0 0 | + | 28 27 1 1 0 0 0 |
| − | 28 29 1 0 0 0 0 | + | 28 29 1 0 0 0 0 |
| − | 29 30 1 0 0 0 0 | + | 29 30 1 0 0 0 0 |
| − | 30 25 1 0 0 0 0 | + | 30 25 1 0 0 0 0 |
| − | 30 31 1 0 0 0 0 | + | 30 31 1 0 0 0 0 |
| − | 31 32 1 0 0 0 0 | + | 31 32 1 0 0 0 0 |
| − | 25 33 1 0 0 0 0 | + | 25 33 1 0 0 0 0 |
| − | 26 34 1 0 0 0 0 | + | 26 34 1 0 0 0 0 |
| − | 27 35 1 0 0 0 0 | + | 27 35 1 0 0 0 0 |
| − | 28 19 1 0 0 0 0 | + | 28 19 1 0 0 0 0 |
| − | S SKP 8 | + | S SKP 8 |
| − | ID FL2FAIGS0002 | + | ID FL2FAIGS0002 |
| − | KNApSAcK_ID C00014351 | + | KNApSAcK_ID C00014351 |
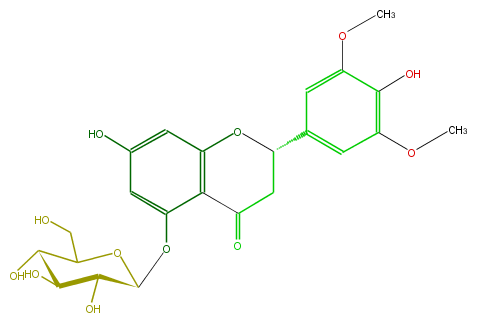
| − | NAME Peruvianoside II;(2S)-2,7,4'-Trihydroxy-3',5'-methoxyflavanone 5-glucoside | + | NAME Peruvianoside II;(2S)-2,7,4'-Trihydroxy-3',5'-methoxyflavanone 5-glucoside |
| − | CAS_RN 450407-31-5 | + | CAS_RN 450407-31-5 |
| − | FORMULA C23H26O12 | + | FORMULA C23H26O12 |
| − | EXACTMASS 494.142426296 | + | EXACTMASS 494.142426296 |
| − | AVERAGEMASS 494.44534 | + | AVERAGEMASS 494.44534 |
| − | SMILES OC(C4O)C(OC(CO)C4O)Oc(c3)c(c(cc3O)1)C(=O)CC(c(c2)cc(c(c(OC)2)O)OC)O1 | + | SMILES OC(C4O)C(OC(CO)C4O)Oc(c3)c(c(cc3O)1)C(=O)CC(c(c2)cc(c(c(OC)2)O)OC)O1 |
M END | M END | ||
| − | |||
Latest revision as of 09:00, 14 March 2009
Copyright: ARM project http://www.metabolome.jp/
35 38 0 0 0 0 0 0 0 0999 V2000
-2.0500 0.1772 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.3373 0.5929 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.0468 -0.6478 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.3300 -1.0571 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.6174 -0.6414 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.6210 0.1836 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.0989 -1.0508 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.8115 -0.6351 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.8079 0.1899 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.0916 0.5992 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
1.4617 0.5707 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.1777 0.1610 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.8906 0.5762 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.8874 1.4012 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.1714 1.8110 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.4585 1.3957 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.1000 -1.6940 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-2.6754 0.5383 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-1.3300 -1.7652 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
3.5185 1.7656 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
2.1714 2.5275 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
2.9260 2.9632 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.5670 0.1857 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
4.3784 0.6541 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-3.8952 -1.8177 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-3.4826 -2.5324 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.6842 -2.3233 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.8907 -2.5501 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.3033 -1.8354 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-3.1017 -2.0444 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-3.2587 -1.4586 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-3.7385 -1.1816 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-4.3784 -2.3009 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-3.9312 -2.2734 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-2.8557 -2.9632 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
1 2 2 0 0 0 0
1 3 1 0 0 0 0
3 4 2 0 0 0 0
4 5 1 0 0 0 0
5 6 2 0 0 0 0
6 2 1 0 0 0 0
5 7 1 0 0 0 0
7 8 1 0 0 0 0
8 9 1 0 0 0 0
9 10 1 0 0 0 0
10 6 1 0 0 0 0
9 11 1 6 0 0 0
11 12 2 0 0 0 0
12 13 1 0 0 0 0
13 14 2 0 0 0 0
14 15 1 0 0 0 0
15 16 2 0 0 0 0
16 11 1 0 0 0 0
7 17 2 0 0 0 0
1 18 1 0 0 0 0
4 19 1 0 0 0 0
14 20 1 0 0 0 0
15 21 1 0 0 0 0
21 22 1 0 0 0 0
13 23 1 0 0 0 0
23 24 1 0 0 0 0
25 26 1 1 0 0 0
26 27 1 1 0 0 0
28 27 1 1 0 0 0
28 29 1 0 0 0 0
29 30 1 0 0 0 0
30 25 1 0 0 0 0
30 31 1 0 0 0 0
31 32 1 0 0 0 0
25 33 1 0 0 0 0
26 34 1 0 0 0 0
27 35 1 0 0 0 0
28 19 1 0 0 0 0
S SKP 8
ID FL2FAIGS0002
KNApSAcK_ID C00014351
NAME Peruvianoside II;(2S)-2,7,4'-Trihydroxy-3',5'-methoxyflavanone 5-glucoside
CAS_RN 450407-31-5
FORMULA C23H26O12
EXACTMASS 494.142426296
AVERAGEMASS 494.44534
SMILES OC(C4O)C(OC(CO)C4O)Oc(c3)c(c(cc3O)1)C(=O)CC(c(c2)cc(c(c(OC)2)O)OC)O1
M END