Mol:FL2FACNP0006
From Metabolomics.JP
(Difference between revisions)
| Line 1: | Line 1: | ||
| − | + | ||
| − | + | ||
| − | Copyright: ARM project http://www.metabolome.jp/ | + | Copyright: ARM project http://www.metabolome.jp/ |
| − | 36 39 0 0 0 0 0 0 0 0999 V2000 | + | 36 39 0 0 0 0 0 0 0 0999 V2000 |
| − | -2.0876 -0.9016 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.0876 -0.9016 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.5667 -1.2023 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.5667 -1.2023 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.0458 -0.9016 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.0458 -0.9016 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.0458 -0.3001 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.0458 -0.3001 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.5667 0.0006 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.5667 0.0006 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.0876 -0.3001 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.0876 -0.3001 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.5249 -1.2023 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.5249 -1.2023 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.0040 -0.9016 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.0040 -0.9016 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.0040 -0.3001 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.0040 -0.3001 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.5249 0.0006 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.5249 0.0006 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.5164 0.0003 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.5164 0.0003 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.5249 -1.7262 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.5249 -1.7262 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.0507 -0.3082 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.0507 -0.3082 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.5851 0.0003 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.5851 0.0003 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.5851 0.6173 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.5851 0.6173 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.0507 0.9258 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.0507 0.9258 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.5164 0.6173 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.5164 0.6173 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.6084 0.0006 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.6084 0.0006 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.5667 -1.8032 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.5667 -1.8032 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.1194 0.9258 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.1194 0.9258 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.5667 0.6015 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.5667 0.6015 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.0870 0.9019 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.0870 0.9019 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.6079 -1.2020 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.6079 -1.2020 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.1283 -0.9016 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.1283 -0.9016 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.1194 -0.3082 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.1194 -0.3082 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.6538 0.0003 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.6538 0.0003 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.0870 1.5028 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.0870 1.5028 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.6074 1.8032 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.6074 1.8032 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.5667 1.8032 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.5667 1.8032 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.0507 1.5418 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.0507 1.5418 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.1283 -0.3007 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.1283 -0.3007 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.5796 0.1506 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.5796 0.1506 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.7195 -0.4592 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.7195 -0.4592 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.1873 -0.3077 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.1873 -0.3077 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.7195 -0.0004 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.7195 -0.0004 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.1873 -0.9223 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.1873 -0.9223 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1 2 2 0 0 0 0 | + | 1 2 2 0 0 0 0 |
| − | 2 3 1 0 0 0 0 | + | 2 3 1 0 0 0 0 |
| − | 3 4 2 0 0 0 0 | + | 3 4 2 0 0 0 0 |
| − | 4 5 1 0 0 0 0 | + | 4 5 1 0 0 0 0 |
| − | 5 6 2 0 0 0 0 | + | 5 6 2 0 0 0 0 |
| − | 6 1 1 0 0 0 0 | + | 6 1 1 0 0 0 0 |
| − | 3 7 1 0 0 0 0 | + | 3 7 1 0 0 0 0 |
| − | 7 8 1 0 0 0 0 | + | 7 8 1 0 0 0 0 |
| − | 8 9 1 0 0 0 0 | + | 8 9 1 0 0 0 0 |
| − | 9 10 1 0 0 0 0 | + | 9 10 1 0 0 0 0 |
| − | 10 4 1 0 0 0 0 | + | 10 4 1 0 0 0 0 |
| − | 9 11 1 0 0 0 0 | + | 9 11 1 0 0 0 0 |
| − | 7 12 2 0 0 0 0 | + | 7 12 2 0 0 0 0 |
| − | 11 13 2 0 0 0 0 | + | 11 13 2 0 0 0 0 |
| − | 13 14 1 0 0 0 0 | + | 13 14 1 0 0 0 0 |
| − | 14 15 2 0 0 0 0 | + | 14 15 2 0 0 0 0 |
| − | 15 16 1 0 0 0 0 | + | 15 16 1 0 0 0 0 |
| − | 16 17 2 0 0 0 0 | + | 16 17 2 0 0 0 0 |
| − | 17 11 1 0 0 0 0 | + | 17 11 1 0 0 0 0 |
| − | 6 18 1 0 0 0 0 | + | 6 18 1 0 0 0 0 |
| − | 2 19 1 0 0 0 0 | + | 2 19 1 0 0 0 0 |
| − | 20 15 1 0 0 0 0 | + | 20 15 1 0 0 0 0 |
| − | 5 21 1 0 0 0 0 | + | 5 21 1 0 0 0 0 |
| − | 21 22 1 0 0 0 0 | + | 21 22 1 0 0 0 0 |
| − | 1 23 1 0 0 0 0 | + | 1 23 1 0 0 0 0 |
| − | 23 24 2 0 0 0 0 | + | 23 24 2 0 0 0 0 |
| − | 14 25 1 0 0 0 0 | + | 14 25 1 0 0 0 0 |
| − | 25 26 1 0 0 0 0 | + | 25 26 1 0 0 0 0 |
| − | 22 27 2 0 0 0 0 | + | 22 27 2 0 0 0 0 |
| − | 27 28 1 0 0 0 0 | + | 27 28 1 0 0 0 0 |
| − | 27 29 1 0 0 0 0 | + | 27 29 1 0 0 0 0 |
| − | 16 30 1 0 0 0 0 | + | 16 30 1 0 0 0 0 |
| − | 18 31 1 0 0 0 0 | + | 18 31 1 0 0 0 0 |
| − | 31 24 1 0 0 0 0 | + | 31 24 1 0 0 0 0 |
| − | 31 32 1 0 0 0 0 | + | 31 32 1 0 0 0 0 |
| − | 31 33 1 0 0 0 0 | + | 31 33 1 0 0 0 0 |
| − | 26 34 2 0 0 0 0 | + | 26 34 2 0 0 0 0 |
| − | 34 35 1 0 0 0 0 | + | 34 35 1 0 0 0 0 |
| − | 34 36 1 0 0 0 0 | + | 34 36 1 0 0 0 0 |
| − | S SKP 8 | + | S SKP 8 |
| − | ID FL2FACNP0006 | + | ID FL2FACNP0006 |
| − | KNApSAcK_ID C00008475 | + | KNApSAcK_ID C00008475 |
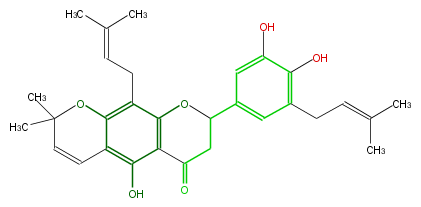
| − | NAME Amoridin | + | NAME Amoridin |
| − | CAS_RN 119347-05-6 | + | CAS_RN 119347-05-6 |
| − | FORMULA C30H34O6 | + | FORMULA C30H34O6 |
| − | EXACTMASS 490.23553882 | + | EXACTMASS 490.23553882 |
| − | AVERAGEMASS 490.58736000000005 | + | AVERAGEMASS 490.58736000000005 |
| − | SMILES c(c34)(C=CC(C)(C)O4)c(O)c(c(c3CC=C(C)C)1)C(CC(c(c2)cc(c(c(O)2)O)CC=C(C)C)O1)=O | + | SMILES c(c34)(C=CC(C)(C)O4)c(O)c(c(c3CC=C(C)C)1)C(CC(c(c2)cc(c(c(O)2)O)CC=C(C)C)O1)=O |
M END | M END | ||
| − | |||
Latest revision as of 09:00, 14 March 2009
Copyright: ARM project http://www.metabolome.jp/
36 39 0 0 0 0 0 0 0 0999 V2000
-2.0876 -0.9016 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.5667 -1.2023 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.0458 -0.9016 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.0458 -0.3001 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.5667 0.0006 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.0876 -0.3001 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.5249 -1.2023 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.0040 -0.9016 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.0040 -0.3001 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.5249 0.0006 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
0.5164 0.0003 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.5249 -1.7262 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
1.0507 -0.3082 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.5851 0.0003 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.5851 0.6173 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.0507 0.9258 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.5164 0.6173 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.6084 0.0006 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-1.5667 -1.8032 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
2.1194 0.9258 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-1.5667 0.6015 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.0870 0.9019 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.6079 -1.2020 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-3.1283 -0.9016 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.1194 -0.3082 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.6538 0.0003 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.0870 1.5028 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.6074 1.8032 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.5667 1.8032 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.0507 1.5418 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-3.1283 -0.3007 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-3.5796 0.1506 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-3.7195 -0.4592 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.1873 -0.3077 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.7195 -0.0004 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.1873 -0.9223 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1 2 2 0 0 0 0
2 3 1 0 0 0 0
3 4 2 0 0 0 0
4 5 1 0 0 0 0
5 6 2 0 0 0 0
6 1 1 0 0 0 0
3 7 1 0 0 0 0
7 8 1 0 0 0 0
8 9 1 0 0 0 0
9 10 1 0 0 0 0
10 4 1 0 0 0 0
9 11 1 0 0 0 0
7 12 2 0 0 0 0
11 13 2 0 0 0 0
13 14 1 0 0 0 0
14 15 2 0 0 0 0
15 16 1 0 0 0 0
16 17 2 0 0 0 0
17 11 1 0 0 0 0
6 18 1 0 0 0 0
2 19 1 0 0 0 0
20 15 1 0 0 0 0
5 21 1 0 0 0 0
21 22 1 0 0 0 0
1 23 1 0 0 0 0
23 24 2 0 0 0 0
14 25 1 0 0 0 0
25 26 1 0 0 0 0
22 27 2 0 0 0 0
27 28 1 0 0 0 0
27 29 1 0 0 0 0
16 30 1 0 0 0 0
18 31 1 0 0 0 0
31 24 1 0 0 0 0
31 32 1 0 0 0 0
31 33 1 0 0 0 0
26 34 2 0 0 0 0
34 35 1 0 0 0 0
34 36 1 0 0 0 0
S SKP 8
ID FL2FACNP0006
KNApSAcK_ID C00008475
NAME Amoridin
CAS_RN 119347-05-6
FORMULA C30H34O6
EXACTMASS 490.23553882
AVERAGEMASS 490.58736000000005
SMILES c(c34)(C=CC(C)(C)O4)c(O)c(c(c3CC=C(C)C)1)C(CC(c(c2)cc(c(c(O)2)O)CC=C(C)C)O1)=O
M END