Mol:FL2FACGS0007
From Metabolomics.JP
(Difference between revisions)
| Line 1: | Line 1: | ||
| − | + | ||
| − | + | ||
| − | Copyright: ARM project http://www.metabolome.jp/ | + | Copyright: ARM project http://www.metabolome.jp/ |
| − | 43 47 0 0 0 0 0 0 0 0999 V2000 | + | 43 47 0 0 0 0 0 0 0 0999 V2000 |
| − | -1.3163 -0.9653 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.3163 -0.9653 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.3163 -1.6077 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.3163 -1.6077 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.7600 -1.9288 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.7600 -1.9288 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.2037 -1.6077 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.2037 -1.6077 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.2037 -0.9653 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.2037 -0.9653 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.7600 -0.6441 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.7600 -0.6441 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.3526 -1.9288 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.3526 -1.9288 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.9089 -1.6077 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.9089 -1.6077 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.9089 -0.9653 0.0000 C 0 0 3 0 0 0 0 0 0 0 0 0 | + | 0.9089 -0.9653 0.0000 C 0 0 3 0 0 0 0 0 0 0 0 0 |
| − | 0.3526 -0.6441 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.3526 -0.6441 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.4650 -0.6442 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.4650 -0.6442 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.0320 -0.9716 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.0320 -0.9716 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.5990 -0.6442 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.5990 -0.6442 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.5990 0.0105 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.5990 0.0105 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.0320 0.3378 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.0320 0.3378 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.4650 0.0105 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.4650 0.0105 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.3526 -2.4726 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.3526 -2.4726 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.1658 0.3377 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.1658 0.3377 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.8724 -0.6442 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.8724 -0.6442 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.0320 1.0562 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.0320 1.0562 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.7600 -2.5710 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.7600 -2.5710 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.3296 2.1975 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.3296 2.1975 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.8975 1.8696 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0 | + | 1.8975 1.8696 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0 |
| − | 1.7173 2.5001 0.0000 C 0 0 3 0 0 0 0 0 0 0 0 0 | + | 1.7173 2.5001 0.0000 C 0 0 3 0 0 0 0 0 0 0 0 0 |
| − | 1.8975 3.1345 0.0000 C 0 0 3 0 0 0 0 0 0 0 0 0 | + | 1.8975 3.1345 0.0000 C 0 0 3 0 0 0 0 0 0 0 0 0 |
| − | 1.3296 3.4624 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0 | + | 1.3296 3.4624 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0 |
| − | 1.5097 2.8319 0.0000 C 0 0 2 0 0 0 0 0 0 0 0 0 | + | 1.5097 2.8319 0.0000 C 0 0 2 0 0 0 0 0 0 0 0 0 |
| − | 1.2849 3.9728 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.2849 3.9728 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.2519 3.3391 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.2519 3.3391 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.1349 2.2590 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.1349 2.2590 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.7653 -3.0564 0.0000 C 0 0 2 0 0 0 0 0 0 0 0 0 | + | -2.7653 -3.0564 0.0000 C 0 0 2 0 0 0 0 0 0 0 0 0 |
| − | -2.4071 -3.5292 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0 | + | -2.4071 -3.5292 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0 |
| − | -1.8913 -3.3286 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0 | + | -1.8913 -3.3286 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0 |
| − | -1.3936 -3.3232 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0 | + | -1.3936 -3.3232 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0 |
| − | -1.7552 -2.9615 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.7552 -2.9615 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.2821 -3.1507 0.0000 C 0 0 3 0 0 0 0 0 0 0 0 0 | + | -2.2821 -3.1507 0.0000 C 0 0 3 0 0 0 0 0 0 0 0 0 |
| − | -3.2258 -3.0160 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.2258 -3.0160 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.7452 -3.9743 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.7452 -3.9743 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.5957 -3.8247 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.5957 -3.8247 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.8069 3.0277 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.8069 3.0277 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.0928 2.3277 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.0928 2.3277 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.5544 -2.4187 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.5544 -2.4187 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.5404 -2.2520 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.5404 -2.2520 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1 2 1 0 0 0 0 | + | 1 2 1 0 0 0 0 |
| − | 2 3 2 0 0 0 0 | + | 2 3 2 0 0 0 0 |
| − | 3 4 1 0 0 0 0 | + | 3 4 1 0 0 0 0 |
| − | 4 5 2 0 0 0 0 | + | 4 5 2 0 0 0 0 |
| − | 5 6 1 0 0 0 0 | + | 5 6 1 0 0 0 0 |
| − | 6 1 2 0 0 0 0 | + | 6 1 2 0 0 0 0 |
| − | 4 7 1 0 0 0 0 | + | 4 7 1 0 0 0 0 |
| − | 7 8 1 0 0 0 0 | + | 7 8 1 0 0 0 0 |
| − | 8 9 1 0 0 0 0 | + | 8 9 1 0 0 0 0 |
| − | 9 10 1 0 0 0 0 | + | 9 10 1 0 0 0 0 |
| − | 10 5 1 0 0 0 0 | + | 10 5 1 0 0 0 0 |
| − | 9 11 1 0 0 0 0 | + | 9 11 1 0 0 0 0 |
| − | 11 12 2 0 0 0 0 | + | 11 12 2 0 0 0 0 |
| − | 12 13 1 0 0 0 0 | + | 12 13 1 0 0 0 0 |
| − | 13 14 2 0 0 0 0 | + | 13 14 2 0 0 0 0 |
| − | 14 15 1 0 0 0 0 | + | 14 15 1 0 0 0 0 |
| − | 15 16 2 0 0 0 0 | + | 15 16 2 0 0 0 0 |
| − | 16 11 1 0 0 0 0 | + | 16 11 1 0 0 0 0 |
| − | 7 17 2 0 0 0 0 | + | 7 17 2 0 0 0 0 |
| − | 14 18 1 0 0 0 0 | + | 14 18 1 0 0 0 0 |
| − | 1 19 1 0 0 0 0 | + | 1 19 1 0 0 0 0 |
| − | 20 15 1 0 0 0 0 | + | 20 15 1 0 0 0 0 |
| − | 3 21 1 0 0 0 0 | + | 3 21 1 0 0 0 0 |
| − | 23 22 1 1 0 0 0 | + | 23 22 1 1 0 0 0 |
| − | 23 24 1 0 0 0 0 | + | 23 24 1 0 0 0 0 |
| − | 24 25 1 0 0 0 0 | + | 24 25 1 0 0 0 0 |
| − | 25 26 1 0 0 0 0 | + | 25 26 1 0 0 0 0 |
| − | 26 27 1 1 0 0 0 | + | 26 27 1 1 0 0 0 |
| − | 27 22 1 1 0 0 0 | + | 27 22 1 1 0 0 0 |
| − | 26 28 1 0 0 0 0 | + | 26 28 1 0 0 0 0 |
| − | 25 29 1 0 0 0 0 | + | 25 29 1 0 0 0 0 |
| − | 24 30 1 0 0 0 0 | + | 24 30 1 0 0 0 0 |
| − | 23 20 1 0 0 0 0 | + | 23 20 1 0 0 0 0 |
| − | 31 32 1 1 0 0 0 | + | 31 32 1 1 0 0 0 |
| − | 32 33 1 1 0 0 0 | + | 32 33 1 1 0 0 0 |
| − | 34 33 1 1 0 0 0 | + | 34 33 1 1 0 0 0 |
| − | 34 35 1 0 0 0 0 | + | 34 35 1 0 0 0 0 |
| − | 35 36 1 0 0 0 0 | + | 35 36 1 0 0 0 0 |
| − | 36 31 1 0 0 0 0 | + | 36 31 1 0 0 0 0 |
| − | 31 37 1 0 0 0 0 | + | 31 37 1 0 0 0 0 |
| − | 32 38 1 0 0 0 0 | + | 32 38 1 0 0 0 0 |
| − | 33 39 1 0 0 0 0 | + | 33 39 1 0 0 0 0 |
| − | 21 34 1 0 0 0 0 | + | 21 34 1 0 0 0 0 |
| − | 27 40 1 0 0 0 0 | + | 27 40 1 0 0 0 0 |
| − | 40 41 1 0 0 0 0 | + | 40 41 1 0 0 0 0 |
| − | 36 42 1 0 0 0 0 | + | 36 42 1 0 0 0 0 |
| − | 42 43 1 0 0 0 0 | + | 42 43 1 0 0 0 0 |
| − | M STY 1 2 SUP | + | M STY 1 2 SUP |
| − | M SLB 1 2 2 | + | M SLB 1 2 2 |
| − | M SAL 2 2 42 43 | + | M SAL 2 2 42 43 |
| − | M SBL 2 1 46 | + | M SBL 2 1 46 |
| − | M SMT 2 CH2OH | + | M SMT 2 CH2OH |
| − | M SVB 2 46 -2.5544 -2.4187 | + | M SVB 2 46 -2.5544 -2.4187 |
| − | M STY 1 1 SUP | + | M STY 1 1 SUP |
| − | M SLB 1 1 1 | + | M SLB 1 1 1 |
| − | M SAL 1 2 40 41 | + | M SAL 1 2 40 41 |
| − | M SBL 1 1 44 | + | M SBL 1 1 44 |
| − | M SMT 1 CH2OH | + | M SMT 1 CH2OH |
| − | M SVB 1 44 0.9555 3.3064 | + | M SVB 1 44 0.9555 3.3064 |
| − | S SKP 8 | + | S SKP 8 |
| − | ID FL2FACGS0007 | + | ID FL2FACGS0007 |
| − | KNApSAcK_ID C00008294 | + | KNApSAcK_ID C00008294 |
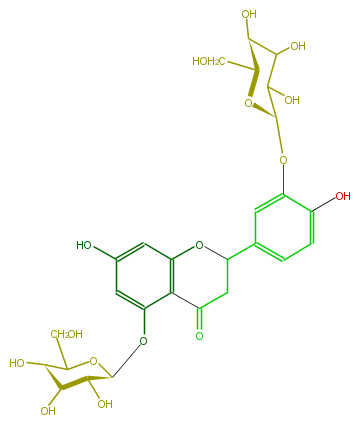
| − | NAME Eriodictyol 5,3'-di-O-glucoside | + | NAME Eriodictyol 5,3'-di-O-glucoside |
| − | CAS_RN 33983-44-7 | + | CAS_RN 33983-44-7 |
| − | FORMULA C27H32O16 | + | FORMULA C27H32O16 |
| − | EXACTMASS 612.1690349759999 | + | EXACTMASS 612.1690349759999 |
| − | AVERAGEMASS 612.53338 | + | AVERAGEMASS 612.53338 |
| − | SMILES O[C@H]([C@H](Oc(c32)cc(cc(OC(c(c4)ccc(O)c4O[C@@H](O5)C(C([C@H](O)[C@H](CO)5)O)O)CC3=O)2)O)1)[C@H]([C@@H](O)C(CO)O1)O | + | SMILES O[C@H]([C@H](Oc(c32)cc(cc(OC(c(c4)ccc(O)c4O[C@@H](O5)C(C([C@H](O)[C@H](CO)5)O)O)CC3=O)2)O)1)[C@H]([C@@H](O)C(CO)O1)O |
M END | M END | ||
| − | |||
Latest revision as of 09:00, 14 March 2009
Copyright: ARM project http://www.metabolome.jp/
43 47 0 0 0 0 0 0 0 0999 V2000
-1.3163 -0.9653 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.3163 -1.6077 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.7600 -1.9288 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.2037 -1.6077 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.2037 -0.9653 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.7600 -0.6441 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.3526 -1.9288 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.9089 -1.6077 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.9089 -0.9653 0.0000 C 0 0 3 0 0 0 0 0 0 0 0 0
0.3526 -0.6441 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
1.4650 -0.6442 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.0320 -0.9716 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.5990 -0.6442 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.5990 0.0105 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.0320 0.3378 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.4650 0.0105 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.3526 -2.4726 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
3.1658 0.3377 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-1.8724 -0.6442 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
2.0320 1.0562 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-0.7600 -2.5710 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
1.3296 2.1975 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
1.8975 1.8696 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0
1.7173 2.5001 0.0000 C 0 0 3 0 0 0 0 0 0 0 0 0
1.8975 3.1345 0.0000 C 0 0 3 0 0 0 0 0 0 0 0 0
1.3296 3.4624 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0
1.5097 2.8319 0.0000 C 0 0 2 0 0 0 0 0 0 0 0 0
1.2849 3.9728 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
2.2519 3.3391 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
2.1349 2.2590 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-2.7653 -3.0564 0.0000 C 0 0 2 0 0 0 0 0 0 0 0 0
-2.4071 -3.5292 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0
-1.8913 -3.3286 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0
-1.3936 -3.3232 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0
-1.7552 -2.9615 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-2.2821 -3.1507 0.0000 C 0 0 3 0 0 0 0 0 0 0 0 0
-3.2258 -3.0160 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-2.7452 -3.9743 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-1.5957 -3.8247 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
0.8069 3.0277 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.0928 2.3277 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-2.5544 -2.4187 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-3.5404 -2.2520 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
1 2 1 0 0 0 0
2 3 2 0 0 0 0
3 4 1 0 0 0 0
4 5 2 0 0 0 0
5 6 1 0 0 0 0
6 1 2 0 0 0 0
4 7 1 0 0 0 0
7 8 1 0 0 0 0
8 9 1 0 0 0 0
9 10 1 0 0 0 0
10 5 1 0 0 0 0
9 11 1 0 0 0 0
11 12 2 0 0 0 0
12 13 1 0 0 0 0
13 14 2 0 0 0 0
14 15 1 0 0 0 0
15 16 2 0 0 0 0
16 11 1 0 0 0 0
7 17 2 0 0 0 0
14 18 1 0 0 0 0
1 19 1 0 0 0 0
20 15 1 0 0 0 0
3 21 1 0 0 0 0
23 22 1 1 0 0 0
23 24 1 0 0 0 0
24 25 1 0 0 0 0
25 26 1 0 0 0 0
26 27 1 1 0 0 0
27 22 1 1 0 0 0
26 28 1 0 0 0 0
25 29 1 0 0 0 0
24 30 1 0 0 0 0
23 20 1 0 0 0 0
31 32 1 1 0 0 0
32 33 1 1 0 0 0
34 33 1 1 0 0 0
34 35 1 0 0 0 0
35 36 1 0 0 0 0
36 31 1 0 0 0 0
31 37 1 0 0 0 0
32 38 1 0 0 0 0
33 39 1 0 0 0 0
21 34 1 0 0 0 0
27 40 1 0 0 0 0
40 41 1 0 0 0 0
36 42 1 0 0 0 0
42 43 1 0 0 0 0
M STY 1 2 SUP
M SLB 1 2 2
M SAL 2 2 42 43
M SBL 2 1 46
M SMT 2 CH2OH
M SVB 2 46 -2.5544 -2.4187
M STY 1 1 SUP
M SLB 1 1 1
M SAL 1 2 40 41
M SBL 1 1 44
M SMT 1 CH2OH
M SVB 1 44 0.9555 3.3064
S SKP 8
ID FL2FACGS0007
KNApSAcK_ID C00008294
NAME Eriodictyol 5,3'-di-O-glucoside
CAS_RN 33983-44-7
FORMULA C27H32O16
EXACTMASS 612.1690349759999
AVERAGEMASS 612.53338
SMILES O[C@H]([C@H](Oc(c32)cc(cc(OC(c(c4)ccc(O)c4O[C@@H](O5)C(C([C@H](O)[C@H](CO)5)O)O)CC3=O)2)O)1)[C@H]([C@@H](O)C(CO)O1)O
M END