Mol:FL2FAAGS0011
From Metabolomics.JP
(Difference between revisions)
| Line 1: | Line 1: | ||
| − | + | ||
| − | + | ||
| − | Copyright: ARM project http://www.metabolome.jp/ | + | Copyright: ARM project http://www.metabolome.jp/ |
| − | 42 46 0 0 0 0 0 0 0 0999 V2000 | + | 42 46 0 0 0 0 0 0 0 0999 V2000 |
| − | -0.6761 -0.5867 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.6761 -0.5867 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.6761 -1.2290 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.6761 -1.2290 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.1198 -1.5502 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.1198 -1.5502 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.4365 -1.2290 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.4365 -1.2290 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.4365 -0.5867 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.4365 -0.5867 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.1198 -0.2655 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.1198 -0.2655 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.9928 -1.5502 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.9928 -1.5502 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.5491 -1.2290 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.5491 -1.2290 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.5491 -0.5867 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.5491 -0.5867 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.9928 -0.2655 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.9928 -0.2655 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.1052 -0.2656 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.1052 -0.2656 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.6721 -0.5930 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.6721 -0.5930 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.2391 -0.2656 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.2391 -0.2656 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.2391 0.3891 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.2391 0.3891 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.6721 0.7164 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.6721 0.7164 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.1052 0.3891 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.1052 0.3891 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.9928 -2.0940 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.9928 -2.0940 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.2324 -0.2655 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.2324 -0.2655 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.8061 0.7164 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.8061 0.7164 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.1198 -2.1923 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.1198 -2.1923 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.2964 -0.3989 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.2964 -0.3989 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.9283 -0.8848 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.9283 -0.8848 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.3981 -0.6787 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.3981 -0.6787 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.8866 -0.6731 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.8866 -0.6731 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.2583 -0.3013 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.2583 -0.3013 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.7221 -0.5461 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.7221 -0.5461 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.8061 -0.6931 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.8061 -0.6931 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.5022 -0.9128 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.5022 -0.9128 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.0944 -1.1886 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.0944 -1.1886 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.1481 0.1917 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.1481 0.1917 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.1481 0.7160 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.1481 0.7160 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.7367 1.1274 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.7367 1.1274 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.0407 1.6540 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.0407 1.6540 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.1804 1.1274 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.1804 1.1274 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.2110 1.7256 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.2110 1.7256 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.9415 1.2588 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.9415 1.2588 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.4025 1.2588 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.4025 1.2588 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.1330 1.7256 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.1330 1.7256 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.4025 2.1923 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.4025 2.1923 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.9415 2.1923 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.9415 2.1923 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.4046 1.7256 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.4046 1.7256 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.8738 1.7256 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.8738 1.7256 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1 2 1 0 0 0 0 | + | 1 2 1 0 0 0 0 |
| − | 2 3 2 0 0 0 0 | + | 2 3 2 0 0 0 0 |
| − | 3 4 1 0 0 0 0 | + | 3 4 1 0 0 0 0 |
| − | 4 5 2 0 0 0 0 | + | 4 5 2 0 0 0 0 |
| − | 5 6 1 0 0 0 0 | + | 5 6 1 0 0 0 0 |
| − | 6 1 2 0 0 0 0 | + | 6 1 2 0 0 0 0 |
| − | 4 7 1 0 0 0 0 | + | 4 7 1 0 0 0 0 |
| − | 7 8 1 0 0 0 0 | + | 7 8 1 0 0 0 0 |
| − | 8 9 1 0 0 0 0 | + | 8 9 1 0 0 0 0 |
| − | 9 10 1 0 0 0 0 | + | 9 10 1 0 0 0 0 |
| − | 10 5 1 0 0 0 0 | + | 10 5 1 0 0 0 0 |
| − | 9 11 1 0 0 0 0 | + | 9 11 1 0 0 0 0 |
| − | 11 12 2 0 0 0 0 | + | 11 12 2 0 0 0 0 |
| − | 12 13 1 0 0 0 0 | + | 12 13 1 0 0 0 0 |
| − | 13 14 2 0 0 0 0 | + | 13 14 2 0 0 0 0 |
| − | 14 15 1 0 0 0 0 | + | 14 15 1 0 0 0 0 |
| − | 15 16 2 0 0 0 0 | + | 15 16 2 0 0 0 0 |
| − | 16 11 1 0 0 0 0 | + | 16 11 1 0 0 0 0 |
| − | 7 17 2 0 0 0 0 | + | 7 17 2 0 0 0 0 |
| − | 18 1 1 0 0 0 0 | + | 18 1 1 0 0 0 0 |
| − | 14 19 1 0 0 0 0 | + | 14 19 1 0 0 0 0 |
| − | 3 20 1 0 0 0 0 | + | 3 20 1 0 0 0 0 |
| − | 21 22 1 1 0 0 0 | + | 21 22 1 1 0 0 0 |
| − | 22 23 1 1 0 0 0 | + | 22 23 1 1 0 0 0 |
| − | 24 23 1 1 0 0 0 | + | 24 23 1 1 0 0 0 |
| − | 24 25 1 0 0 0 0 | + | 24 25 1 0 0 0 0 |
| − | 25 26 1 0 0 0 0 | + | 25 26 1 0 0 0 0 |
| − | 26 21 1 0 0 0 0 | + | 26 21 1 0 0 0 0 |
| − | 21 27 1 0 0 0 0 | + | 21 27 1 0 0 0 0 |
| − | 22 28 1 0 0 0 0 | + | 22 28 1 0 0 0 0 |
| − | 23 29 1 0 0 0 0 | + | 23 29 1 0 0 0 0 |
| − | 24 18 1 0 0 0 0 | + | 24 18 1 0 0 0 0 |
| − | 26 30 1 0 0 0 0 | + | 26 30 1 0 0 0 0 |
| − | 30 31 1 0 0 0 0 | + | 30 31 1 0 0 0 0 |
| − | 31 32 1 0 0 0 0 | + | 31 32 1 0 0 0 0 |
| − | 32 33 2 0 0 0 0 | + | 32 33 2 0 0 0 0 |
| − | 32 34 1 0 0 0 0 | + | 32 34 1 0 0 0 0 |
| − | 35 36 2 0 0 0 0 | + | 35 36 2 0 0 0 0 |
| − | 36 37 1 0 0 0 0 | + | 36 37 1 0 0 0 0 |
| − | 37 38 2 0 0 0 0 | + | 37 38 2 0 0 0 0 |
| − | 38 39 1 0 0 0 0 | + | 38 39 1 0 0 0 0 |
| − | 39 40 2 0 0 0 0 | + | 39 40 2 0 0 0 0 |
| − | 40 35 1 0 0 0 0 | + | 40 35 1 0 0 0 0 |
| − | 38 41 1 0 0 0 0 | + | 38 41 1 0 0 0 0 |
| − | 35 42 1 0 0 0 0 | + | 35 42 1 0 0 0 0 |
| − | 42 34 2 0 0 0 0 | + | 42 34 2 0 0 0 0 |
| − | S SKP 8 | + | S SKP 8 |
| − | ID FL2FAAGS0011 | + | ID FL2FAAGS0011 |
| − | KNApSAcK_ID C00008211 | + | KNApSAcK_ID C00008211 |
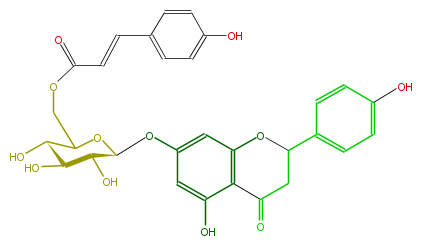
| − | NAME Prunin 6''-p-coumarate | + | NAME Prunin 6''-p-coumarate |
| − | CAS_RN 96686-70-3 | + | CAS_RN 96686-70-3 |
| − | FORMULA C30H28O12 | + | FORMULA C30H28O12 |
| − | EXACTMASS 580.15807636 | + | EXACTMASS 580.15807636 |
| − | AVERAGEMASS 580.53612 | + | AVERAGEMASS 580.53612 |
| − | SMILES c(c(O)1)c(OC(C4O)OC(COC(C=Cc(c5)ccc(O)c5)=O)C(C4O)O)cc(O2)c(C(CC2c(c3)ccc(O)c3)=O)1 | + | SMILES c(c(O)1)c(OC(C4O)OC(COC(C=Cc(c5)ccc(O)c5)=O)C(C4O)O)cc(O2)c(C(CC2c(c3)ccc(O)c3)=O)1 |
M END | M END | ||
| − | |||
Latest revision as of 09:00, 14 March 2009
Copyright: ARM project http://www.metabolome.jp/
42 46 0 0 0 0 0 0 0 0999 V2000
-0.6761 -0.5867 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.6761 -1.2290 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.1198 -1.5502 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.4365 -1.2290 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.4365 -0.5867 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.1198 -0.2655 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.9928 -1.5502 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.5491 -1.2290 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.5491 -0.5867 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.9928 -0.2655 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
2.1052 -0.2656 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.6721 -0.5930 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.2391 -0.2656 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.2391 0.3891 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.6721 0.7164 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.1052 0.3891 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.9928 -2.0940 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-1.2324 -0.2655 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
3.8061 0.7164 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-0.1198 -2.1923 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-3.2964 -0.3989 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.9283 -0.8848 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.3981 -0.6787 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.8866 -0.6731 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.2583 -0.3013 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-2.7221 -0.5461 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-3.8061 -0.6931 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-3.5022 -0.9128 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-2.0944 -1.1886 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-3.1481 0.1917 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-3.1481 0.7160 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-2.7367 1.1274 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-3.0407 1.6540 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-2.1804 1.1274 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.2110 1.7256 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.9415 1.2588 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.4025 1.2588 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.1330 1.7256 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.4025 2.1923 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.9415 2.1923 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.4046 1.7256 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-1.8738 1.7256 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1 2 1 0 0 0 0
2 3 2 0 0 0 0
3 4 1 0 0 0 0
4 5 2 0 0 0 0
5 6 1 0 0 0 0
6 1 2 0 0 0 0
4 7 1 0 0 0 0
7 8 1 0 0 0 0
8 9 1 0 0 0 0
9 10 1 0 0 0 0
10 5 1 0 0 0 0
9 11 1 0 0 0 0
11 12 2 0 0 0 0
12 13 1 0 0 0 0
13 14 2 0 0 0 0
14 15 1 0 0 0 0
15 16 2 0 0 0 0
16 11 1 0 0 0 0
7 17 2 0 0 0 0
18 1 1 0 0 0 0
14 19 1 0 0 0 0
3 20 1 0 0 0 0
21 22 1 1 0 0 0
22 23 1 1 0 0 0
24 23 1 1 0 0 0
24 25 1 0 0 0 0
25 26 1 0 0 0 0
26 21 1 0 0 0 0
21 27 1 0 0 0 0
22 28 1 0 0 0 0
23 29 1 0 0 0 0
24 18 1 0 0 0 0
26 30 1 0 0 0 0
30 31 1 0 0 0 0
31 32 1 0 0 0 0
32 33 2 0 0 0 0
32 34 1 0 0 0 0
35 36 2 0 0 0 0
36 37 1 0 0 0 0
37 38 2 0 0 0 0
38 39 1 0 0 0 0
39 40 2 0 0 0 0
40 35 1 0 0 0 0
38 41 1 0 0 0 0
35 42 1 0 0 0 0
42 34 2 0 0 0 0
S SKP 8
ID FL2FAAGS0011
KNApSAcK_ID C00008211
NAME Prunin 6''-p-coumarate
CAS_RN 96686-70-3
FORMULA C30H28O12
EXACTMASS 580.15807636
AVERAGEMASS 580.53612
SMILES c(c(O)1)c(OC(C4O)OC(COC(C=Cc(c5)ccc(O)c5)=O)C(C4O)O)cc(O2)c(C(CC2c(c3)ccc(O)c3)=O)1
M END