Mol:FL1DA9NR0002
From Metabolomics.JP
(Difference between revisions)
| Line 1: | Line 1: | ||
| − | + | ||
| − | + | ||
| − | Copyright: ARM project http://www.metabolome.jp/ | + | Copyright: ARM project http://www.metabolome.jp/ |
| − | 36 40 0 0 0 0 0 0 0 0999 V2000 | + | 36 40 0 0 0 0 0 0 0 0999 V2000 |
| − | 2.2247 -0.1203 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.2247 -0.1203 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.2247 -0.7627 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.2247 -0.7627 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.7810 -1.0839 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.7810 -1.0839 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.3373 -0.7627 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.3373 -0.7627 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.3373 -0.1203 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.3373 -0.1203 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.7810 0.2009 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.7810 0.2009 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.0915 -0.7420 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.0915 -0.7420 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.6478 -1.0632 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.6478 -1.0632 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.5354 -1.0631 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.5354 -1.0631 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.0255 -0.7295 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.0255 -0.7295 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.5354 -1.7039 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.5354 -1.7039 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.0255 -0.0914 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.0255 -0.0914 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.5782 0.2277 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.5782 0.2277 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.1308 -0.0914 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.1308 -0.0914 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.1308 -0.7295 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.1308 -0.7295 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.5782 -1.0486 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.5782 -1.0486 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.5782 -1.6866 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.5782 -1.6866 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.5782 0.8658 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.5782 0.8658 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.1308 1.1849 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.1308 1.1849 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.6835 0.8658 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.6835 0.8658 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.6835 0.2277 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.6835 0.2277 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.1371 1.5503 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.1371 1.5503 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.5596 1.2409 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.5596 1.2409 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.1435 1.2010 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.1435 1.2010 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.5954 0.8697 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.5954 0.8697 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.6160 0.2509 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.6160 0.2509 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.1420 1.4163 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.1420 1.4163 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.2377 0.8697 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.2377 0.8697 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.2782 1.7039 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.2782 1.7039 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.2992 1.0308 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.2992 1.0308 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.1280 0.5484 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.1280 0.5484 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.6833 -1.0485 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.6833 -1.0485 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.2347 -0.7302 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.2347 -0.7302 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.7860 -1.0485 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.7860 -1.0485 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.3373 -0.7302 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.3373 -0.7302 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.7860 -1.6851 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.7860 -1.6851 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1 2 1 0 0 0 0 | + | 1 2 1 0 0 0 0 |
| − | 2 3 2 0 0 0 0 | + | 2 3 2 0 0 0 0 |
| − | 3 4 1 0 0 0 0 | + | 3 4 1 0 0 0 0 |
| − | 4 5 2 0 0 0 0 | + | 4 5 2 0 0 0 0 |
| − | 5 6 1 0 0 0 0 | + | 5 6 1 0 0 0 0 |
| − | 6 1 2 0 0 0 0 | + | 6 1 2 0 0 0 0 |
| − | 7 8 2 0 0 0 0 | + | 7 8 2 0 0 0 0 |
| − | 7 9 1 0 0 0 0 | + | 7 9 1 0 0 0 0 |
| − | 9 10 1 0 0 0 0 | + | 9 10 1 0 0 0 0 |
| − | 9 11 2 0 0 0 0 | + | 9 11 2 0 0 0 0 |
| − | 2 8 1 0 0 0 0 | + | 2 8 1 0 0 0 0 |
| − | 10 12 2 0 0 0 0 | + | 10 12 2 0 0 0 0 |
| − | 12 13 1 0 0 0 0 | + | 12 13 1 0 0 0 0 |
| − | 13 14 2 0 0 0 0 | + | 13 14 2 0 0 0 0 |
| − | 14 15 1 0 0 0 0 | + | 14 15 1 0 0 0 0 |
| − | 15 16 2 0 0 0 0 | + | 15 16 2 0 0 0 0 |
| − | 16 10 1 0 0 0 0 | + | 16 10 1 0 0 0 0 |
| − | 16 17 1 0 0 0 0 | + | 16 17 1 0 0 0 0 |
| − | 13 18 1 0 0 0 0 | + | 13 18 1 0 0 0 0 |
| − | 18 19 1 0 0 0 0 | + | 18 19 1 0 0 0 0 |
| − | 19 20 1 0 0 0 0 | + | 19 20 1 0 0 0 0 |
| − | 20 21 1 0 0 0 0 | + | 20 21 1 0 0 0 0 |
| − | 21 14 1 0 0 0 0 | + | 21 14 1 0 0 0 0 |
| − | 20 22 1 0 0 0 0 | + | 20 22 1 0 0 0 0 |
| − | 22 23 1 0 0 0 0 | + | 22 23 1 0 0 0 0 |
| − | 23 24 1 0 0 0 0 | + | 23 24 1 0 0 0 0 |
| − | 24 18 1 0 0 0 0 | + | 24 18 1 0 0 0 0 |
| − | 24 25 1 0 0 0 0 | + | 24 25 1 0 0 0 0 |
| − | 25 26 1 0 0 0 0 | + | 25 26 1 0 0 0 0 |
| − | 26 12 1 0 0 0 0 | + | 26 12 1 0 0 0 0 |
| − | 25 27 1 0 0 0 0 | + | 25 27 1 0 0 0 0 |
| − | 25 28 1 0 0 0 0 | + | 25 28 1 0 0 0 0 |
| − | 24 29 1 1 0 0 0 | + | 24 29 1 1 0 0 0 |
| − | 20 30 1 1 0 0 0 | + | 20 30 1 1 0 0 0 |
| − | 18 31 1 6 0 0 0 | + | 18 31 1 6 0 0 0 |
| − | 15 32 1 0 0 0 0 | + | 15 32 1 0 0 0 0 |
| − | 32 33 1 0 0 0 0 | + | 32 33 1 0 0 0 0 |
| − | 33 34 2 0 0 0 0 | + | 33 34 2 0 0 0 0 |
| − | 34 35 1 0 0 0 0 | + | 34 35 1 0 0 0 0 |
| − | 34 36 1 0 0 0 0 | + | 34 36 1 0 0 0 0 |
| − | S SKP 8 | + | S SKP 8 |
| − | ID FL1DA9NR0002 | + | ID FL1DA9NR0002 |
| − | KNApSAcK_ID C00007158 | + | KNApSAcK_ID C00007158 |
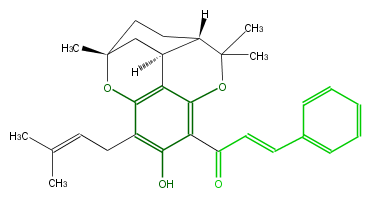
| − | NAME 3'-Prenylrubranine | + | NAME 3'-Prenylrubranine |
| − | CAS_RN 116133-67-6 | + | CAS_RN 116133-67-6 |
| − | FORMULA C30H34O4 | + | FORMULA C30H34O4 |
| − | EXACTMASS 458.24570957599997 | + | EXACTMASS 458.24570957599997 |
| − | AVERAGEMASS 458.58856000000003 | + | AVERAGEMASS 458.58856000000003 |
| − | SMILES C(c(c5)cccc5)=CC(=O)c(c42)c(O)c(c(c41)OC(C3)(C)CC1(C([H])(C3)C(O2)(C)C)[H])CC=C(C)C | + | SMILES C(c(c5)cccc5)=CC(=O)c(c42)c(O)c(c(c41)OC(C3)(C)CC1(C([H])(C3)C(O2)(C)C)[H])CC=C(C)C |
M END | M END | ||
| − | |||
Latest revision as of 09:00, 14 March 2009
Copyright: ARM project http://www.metabolome.jp/
36 40 0 0 0 0 0 0 0 0999 V2000
2.2247 -0.1203 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.2247 -0.7627 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.7810 -1.0839 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.3373 -0.7627 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.3373 -0.1203 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.7810 0.2009 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.0915 -0.7420 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.6478 -1.0632 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.5354 -1.0631 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.0255 -0.7295 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.5354 -1.7039 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-0.0255 -0.0914 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.5782 0.2277 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.1308 -0.0914 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.1308 -0.7295 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.5782 -1.0486 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.5782 -1.6866 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-0.5782 0.8658 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.1308 1.1849 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.6835 0.8658 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.6835 0.2277 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-1.1371 1.5503 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.5596 1.2409 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.1435 1.2010 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.5954 0.8697 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.6160 0.2509 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
1.1420 1.4163 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.2377 0.8697 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.2782 1.7039 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0
-2.2992 1.0308 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.1280 0.5484 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0
-1.6833 -1.0485 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.2347 -0.7302 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.7860 -1.0485 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-3.3373 -0.7302 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.7860 -1.6851 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1 2 1 0 0 0 0
2 3 2 0 0 0 0
3 4 1 0 0 0 0
4 5 2 0 0 0 0
5 6 1 0 0 0 0
6 1 2 0 0 0 0
7 8 2 0 0 0 0
7 9 1 0 0 0 0
9 10 1 0 0 0 0
9 11 2 0 0 0 0
2 8 1 0 0 0 0
10 12 2 0 0 0 0
12 13 1 0 0 0 0
13 14 2 0 0 0 0
14 15 1 0 0 0 0
15 16 2 0 0 0 0
16 10 1 0 0 0 0
16 17 1 0 0 0 0
13 18 1 0 0 0 0
18 19 1 0 0 0 0
19 20 1 0 0 0 0
20 21 1 0 0 0 0
21 14 1 0 0 0 0
20 22 1 0 0 0 0
22 23 1 0 0 0 0
23 24 1 0 0 0 0
24 18 1 0 0 0 0
24 25 1 0 0 0 0
25 26 1 0 0 0 0
26 12 1 0 0 0 0
25 27 1 0 0 0 0
25 28 1 0 0 0 0
24 29 1 1 0 0 0
20 30 1 1 0 0 0
18 31 1 6 0 0 0
15 32 1 0 0 0 0
32 33 1 0 0 0 0
33 34 2 0 0 0 0
34 35 1 0 0 0 0
34 36 1 0 0 0 0
S SKP 8
ID FL1DA9NR0002
KNApSAcK_ID C00007158
NAME 3'-Prenylrubranine
CAS_RN 116133-67-6
FORMULA C30H34O4
EXACTMASS 458.24570957599997
AVERAGEMASS 458.58856000000003
SMILES C(c(c5)cccc5)=CC(=O)c(c42)c(O)c(c(c41)OC(C3)(C)CC1(C([H])(C3)C(O2)(C)C)[H])CC=C(C)C
M END