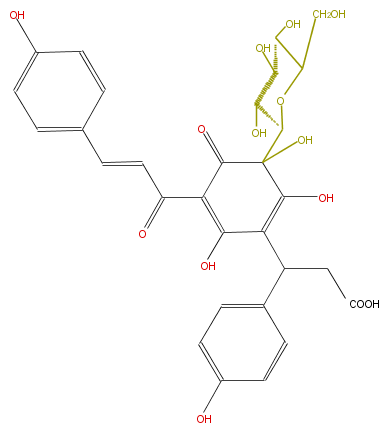
Mol:FL1CQUCN0001
From Metabolomics.JP
(Difference between revisions)
| Line 1: | Line 1: | ||
| − | + | ||
| − | + | ||
| − | Copyright: ARM project http://www.metabolome.jp/ | + | Copyright: ARM project http://www.metabolome.jp/ |
| − | 44 47 0 0 0 0 0 0 0 0999 V2000 | + | 44 47 0 0 0 0 0 0 0 0999 V2000 |
| − | -3.3470 -2.0107 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.3470 -2.0107 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.3470 -2.8669 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.3470 -2.8669 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.6055 -3.2950 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.6055 -3.2950 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.8641 -2.8669 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.8641 -2.8669 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.8641 -2.0107 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.8641 -2.0107 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.6055 -1.5826 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.6055 -1.5826 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.6055 -0.7268 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.6055 -0.7268 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.8644 -0.2990 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.8644 -0.2990 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.3466 -0.2990 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.3466 -0.2990 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.1690 -0.7005 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.1690 -0.7005 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.4735 -0.2990 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.4735 -0.2990 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.4735 0.5040 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.4735 0.5040 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.1690 0.9056 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.1690 0.9056 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.8644 0.5040 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.8644 0.5040 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.2212 -0.7001 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.2212 -0.7001 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.2212 -1.5006 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.2212 -1.5006 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.9145 -0.2999 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.9145 -0.2999 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.6078 -0.7001 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.6078 -0.7001 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.3009 -0.2999 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.3009 -0.2999 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.0751 -0.7468 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.0751 -0.7468 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.8492 -0.2999 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.8492 -0.2999 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.8492 0.5940 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.8492 0.5940 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.0751 1.0409 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.0751 1.0409 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.3009 0.5940 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.3009 0.5940 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 4.6227 1.0405 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 4.6227 1.0405 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.2212 0.9052 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.2212 0.9052 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.5592 0.9052 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.5592 0.9052 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.6258 1.6969 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.6258 1.6969 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.1906 1.6749 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.1906 1.6749 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.4813 1.8580 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.4813 1.8580 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.5151 2.5826 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.5151 2.5826 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.1610 3.2080 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.1610 3.2080 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.9389 2.9334 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.9389 2.9334 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.8703 2.1782 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.8703 2.1782 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.7675 1.3623 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.7675 1.3623 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.0161 2.8893 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.0161 2.8893 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.2642 3.5931 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.2642 3.5931 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.6055 -4.1507 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.6055 -4.1507 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.1690 -1.4493 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.1690 -1.4493 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -4.0421 -0.7005 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -4.0421 -0.7005 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -4.0421 -1.3154 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -4.0421 -1.3154 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -4.6227 -0.3653 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -4.6227 -0.3653 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.6430 4.0379 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.6430 4.0379 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.9346 4.1507 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.9346 4.1507 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1 2 2 0 0 0 0 | + | 1 2 2 0 0 0 0 |
| − | 2 3 1 0 0 0 0 | + | 2 3 1 0 0 0 0 |
| − | 3 4 2 0 0 0 0 | + | 3 4 2 0 0 0 0 |
| − | 4 5 1 0 0 0 0 | + | 4 5 1 0 0 0 0 |
| − | 5 6 2 0 0 0 0 | + | 5 6 2 0 0 0 0 |
| − | 6 1 1 0 0 0 0 | + | 6 1 1 0 0 0 0 |
| − | 6 7 1 0 0 0 0 | + | 6 7 1 0 0 0 0 |
| − | 7 8 1 0 0 0 0 | + | 7 8 1 0 0 0 0 |
| − | 7 9 1 0 0 0 0 | + | 7 9 1 0 0 0 0 |
| − | 8 10 1 0 0 0 0 | + | 8 10 1 0 0 0 0 |
| − | 10 11 2 0 0 0 0 | + | 10 11 2 0 0 0 0 |
| − | 11 12 1 0 0 0 0 | + | 11 12 1 0 0 0 0 |
| − | 12 13 1 0 0 0 0 | + | 12 13 1 0 0 0 0 |
| − | 13 14 1 0 0 0 0 | + | 13 14 1 0 0 0 0 |
| − | 14 8 2 0 0 0 0 | + | 14 8 2 0 0 0 0 |
| − | 11 15 1 0 0 0 0 | + | 11 15 1 0 0 0 0 |
| − | 15 16 2 0 0 0 0 | + | 15 16 2 0 0 0 0 |
| − | 15 17 1 0 0 0 0 | + | 15 17 1 0 0 0 0 |
| − | 17 18 2 0 0 0 0 | + | 17 18 2 0 0 0 0 |
| − | 18 19 1 0 0 0 0 | + | 18 19 1 0 0 0 0 |
| − | 19 20 2 0 0 0 0 | + | 19 20 2 0 0 0 0 |
| − | 20 21 1 0 0 0 0 | + | 20 21 1 0 0 0 0 |
| − | 21 22 2 0 0 0 0 | + | 21 22 2 0 0 0 0 |
| − | 22 23 1 0 0 0 0 | + | 22 23 1 0 0 0 0 |
| − | 23 24 2 0 0 0 0 | + | 23 24 2 0 0 0 0 |
| − | 24 19 1 0 0 0 0 | + | 24 19 1 0 0 0 0 |
| − | 22 25 1 0 0 0 0 | + | 22 25 1 0 0 0 0 |
| − | 12 26 2 0 0 0 0 | + | 12 26 2 0 0 0 0 |
| − | 14 27 1 0 0 0 0 | + | 14 27 1 0 0 0 0 |
| − | 13 28 1 0 0 0 0 | + | 13 28 1 0 0 0 0 |
| − | 29 30 1 1 0 0 0 | + | 29 30 1 1 0 0 0 |
| − | 30 31 1 1 0 0 0 | + | 30 31 1 1 0 0 0 |
| − | 32 31 1 1 0 0 0 | + | 32 31 1 1 0 0 0 |
| − | 32 33 1 0 0 0 0 | + | 32 33 1 0 0 0 0 |
| − | 33 34 1 0 0 0 0 | + | 33 34 1 0 0 0 0 |
| − | 34 29 1 0 0 0 0 | + | 34 29 1 0 0 0 0 |
| − | 30 35 1 0 0 0 0 | + | 30 35 1 0 0 0 0 |
| − | 31 36 1 0 0 0 0 | + | 31 36 1 0 0 0 0 |
| − | 29 13 1 0 0 0 0 | + | 29 13 1 0 0 0 0 |
| − | 32 37 1 0 0 0 0 | + | 32 37 1 0 0 0 0 |
| − | 3 38 1 0 0 0 0 | + | 3 38 1 0 0 0 0 |
| − | 10 39 1 0 0 0 0 | + | 10 39 1 0 0 0 0 |
| − | 40 41 2 0 0 0 0 | + | 40 41 2 0 0 0 0 |
| − | 40 42 1 0 0 0 0 | + | 40 42 1 0 0 0 0 |
| − | 9 40 1 0 0 0 0 | + | 9 40 1 0 0 0 0 |
| − | 43 44 1 0 0 0 0 | + | 43 44 1 0 0 0 0 |
| − | 33 43 1 0 0 0 0 | + | 33 43 1 0 0 0 0 |
| − | M STY 1 1 SUP | + | M STY 1 1 SUP |
| − | M SLB 1 1 1 | + | M SLB 1 1 1 |
| − | M SAL 1 3 40 41 42 | + | M SAL 1 3 40 41 42 |
| − | M SBL 1 1 45 | + | M SBL 1 1 45 |
| − | M SMT 1 ^ COOH | + | M SMT 1 ^ COOH |
| − | M SBV 1 45 0.6955 0.4016 | + | M SBV 1 45 0.6955 0.4016 |
| − | M STY 1 2 SUP | + | M STY 1 2 SUP |
| − | M SLB 1 2 2 | + | M SLB 1 2 2 |
| − | M SAL 2 2 43 44 | + | M SAL 2 2 43 44 |
| − | M SBL 2 1 47 | + | M SBL 2 1 47 |
| − | M SMT 2 CH2OH | + | M SMT 2 CH2OH |
| − | M SBV 2 47 -0.2960 -1.1045 | + | M SBV 2 47 -0.2960 -1.1045 |
| − | S SKP 5 | + | S SKP 5 |
| − | ID FL1CQUCN0001 | + | ID FL1CQUCN0001 |
| − | FORMULA C30H30O14 | + | FORMULA C30H30O14 |
| − | EXACTMASS 614.163555668 | + | EXACTMASS 614.163555668 |
| − | AVERAGEMASS 614.5508 | + | AVERAGEMASS 614.5508 |
| − | SMILES C(C(C(=O)C=Cc(c4)ccc(c4)O)=1)(C(O)(C(O3)C(C(C(C3CO)O)O)O)C(=C(C(CC(O)=O)c(c2)ccc(c2)O)C1O)O)=O | + | SMILES C(C(C(=O)C=Cc(c4)ccc(c4)O)=1)(C(O)(C(O3)C(C(C(C3CO)O)O)O)C(=C(C(CC(O)=O)c(c2)ccc(c2)O)C1O)O)=O |
M END | M END | ||
| − | |||
Latest revision as of 09:00, 14 March 2009
Copyright: ARM project http://www.metabolome.jp/
44 47 0 0 0 0 0 0 0 0999 V2000
-3.3470 -2.0107 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-3.3470 -2.8669 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.6055 -3.2950 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.8641 -2.8669 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.8641 -2.0107 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.6055 -1.5826 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.6055 -0.7268 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.8644 -0.2990 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-3.3466 -0.2990 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.1690 -0.7005 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.4735 -0.2990 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.4735 0.5040 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.1690 0.9056 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.8644 0.5040 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.2212 -0.7001 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.2212 -1.5006 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
0.9145 -0.2999 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.6078 -0.7001 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.3009 -0.2999 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.0751 -0.7468 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.8492 -0.2999 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.8492 0.5940 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.0751 1.0409 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.3009 0.5940 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
4.6227 1.0405 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
0.2212 0.9052 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-2.5592 0.9052 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-1.6258 1.6969 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-1.1906 1.6749 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.4813 1.8580 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.5151 2.5826 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.1610 3.2080 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.9389 2.9334 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.8703 2.1782 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-0.7675 1.3623 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
0.0161 2.8893 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-0.2642 3.5931 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-2.6055 -4.1507 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-1.1690 -1.4493 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-4.0421 -0.7005 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-4.0421 -1.3154 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-4.6227 -0.3653 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-0.6430 4.0379 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.9346 4.1507 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
1 2 2 0 0 0 0
2 3 1 0 0 0 0
3 4 2 0 0 0 0
4 5 1 0 0 0 0
5 6 2 0 0 0 0
6 1 1 0 0 0 0
6 7 1 0 0 0 0
7 8 1 0 0 0 0
7 9 1 0 0 0 0
8 10 1 0 0 0 0
10 11 2 0 0 0 0
11 12 1 0 0 0 0
12 13 1 0 0 0 0
13 14 1 0 0 0 0
14 8 2 0 0 0 0
11 15 1 0 0 0 0
15 16 2 0 0 0 0
15 17 1 0 0 0 0
17 18 2 0 0 0 0
18 19 1 0 0 0 0
19 20 2 0 0 0 0
20 21 1 0 0 0 0
21 22 2 0 0 0 0
22 23 1 0 0 0 0
23 24 2 0 0 0 0
24 19 1 0 0 0 0
22 25 1 0 0 0 0
12 26 2 0 0 0 0
14 27 1 0 0 0 0
13 28 1 0 0 0 0
29 30 1 1 0 0 0
30 31 1 1 0 0 0
32 31 1 1 0 0 0
32 33 1 0 0 0 0
33 34 1 0 0 0 0
34 29 1 0 0 0 0
30 35 1 0 0 0 0
31 36 1 0 0 0 0
29 13 1 0 0 0 0
32 37 1 0 0 0 0
3 38 1 0 0 0 0
10 39 1 0 0 0 0
40 41 2 0 0 0 0
40 42 1 0 0 0 0
9 40 1 0 0 0 0
43 44 1 0 0 0 0
33 43 1 0 0 0 0
M STY 1 1 SUP
M SLB 1 1 1
M SAL 1 3 40 41 42
M SBL 1 1 45
M SMT 1 ^ COOH
M SBV 1 45 0.6955 0.4016
M STY 1 2 SUP
M SLB 1 2 2
M SAL 2 2 43 44
M SBL 2 1 47
M SMT 2 CH2OH
M SBV 2 47 -0.2960 -1.1045
S SKP 5
ID FL1CQUCN0001
FORMULA C30H30O14
EXACTMASS 614.163555668
AVERAGEMASS 614.5508
SMILES C(C(C(=O)C=Cc(c4)ccc(c4)O)=1)(C(O)(C(O3)C(C(C(C3CO)O)O)O)C(=C(C(CC(O)=O)c(c2)ccc(c2)O)C1O)O)=O
M END