Mol:FL1CELNS0003
From Metabolomics.JP
(Difference between revisions)
| Line 1: | Line 1: | ||
| − | + | ||
| − | + | ||
| − | Copyright: ARM project http://www.metabolome.jp/ | + | Copyright: ARM project http://www.metabolome.jp/ |
| − | 29 31 0 0 0 0 0 0 0 0999 V2000 | + | 29 31 0 0 0 0 0 0 0 0999 V2000 |
| − | -2.3731 0.3551 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.3731 0.3551 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.3731 -0.2873 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.3731 -0.2873 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.8168 -0.6085 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.8168 -0.6085 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.2605 -0.2873 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.2605 -0.2873 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.2605 0.3551 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.2605 0.3551 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.8168 0.6763 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.8168 0.6763 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.7042 -0.6085 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.7042 -0.6085 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.1479 -0.2873 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.1479 -0.2873 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.4082 -0.6083 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.4082 -0.6083 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.9643 -0.2873 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.9643 -0.2873 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.5253 -0.6112 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.5253 -0.6112 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.0863 -0.2873 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.0863 -0.2873 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.0863 0.3605 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.0863 0.3605 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.5253 0.6844 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.5253 0.6844 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.9643 0.3605 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.9643 0.3605 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.7042 -1.1098 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.7042 -1.1098 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.8168 -1.2506 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.8168 -1.2506 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.9840 0.5536 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.9840 0.5536 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.3616 0.0339 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.3616 0.0339 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.9840 -0.4858 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.9840 -0.4858 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.5253 -1.2588 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.5253 -1.2588 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.9523 -0.7873 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.9523 -0.7873 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.8183 -1.2873 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.8183 -1.2873 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.7044 0.6761 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.7044 0.6761 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.0101 0.2636 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.0101 0.2636 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.9523 0.8605 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.9523 0.8605 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.8183 1.3605 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.8183 1.3605 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.8057 1.2588 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.8057 1.2588 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.2444 2.1574 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.2444 2.1574 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1 2 1 0 0 0 0 | + | 1 2 1 0 0 0 0 |
| − | 2 3 2 0 0 0 0 | + | 2 3 2 0 0 0 0 |
| − | 3 4 1 0 0 0 0 | + | 3 4 1 0 0 0 0 |
| − | 4 5 2 0 0 0 0 | + | 4 5 2 0 0 0 0 |
| − | 5 6 1 0 0 0 0 | + | 5 6 1 0 0 0 0 |
| − | 6 1 2 0 0 0 0 | + | 6 1 2 0 0 0 0 |
| − | 4 7 1 0 0 0 0 | + | 4 7 1 0 0 0 0 |
| − | 7 8 1 0 0 0 0 | + | 7 8 1 0 0 0 0 |
| − | 8 9 2 0 0 0 0 | + | 8 9 2 0 0 0 0 |
| − | 9 10 1 0 0 0 0 | + | 9 10 1 0 0 0 0 |
| − | 10 11 2 0 0 0 0 | + | 10 11 2 0 0 0 0 |
| − | 11 12 1 0 0 0 0 | + | 11 12 1 0 0 0 0 |
| − | 12 13 2 0 0 0 0 | + | 12 13 2 0 0 0 0 |
| − | 13 14 1 0 0 0 0 | + | 13 14 1 0 0 0 0 |
| − | 14 15 2 0 0 0 0 | + | 14 15 2 0 0 0 0 |
| − | 15 10 1 0 0 0 0 | + | 15 10 1 0 0 0 0 |
| − | 7 16 2 0 0 0 0 | + | 7 16 2 0 0 0 0 |
| − | 3 17 1 0 0 0 0 | + | 3 17 1 0 0 0 0 |
| − | 1 18 1 0 0 0 0 | + | 1 18 1 0 0 0 0 |
| − | 18 19 1 0 0 0 0 | + | 18 19 1 0 0 0 0 |
| − | 19 20 1 0 0 0 0 | + | 19 20 1 0 0 0 0 |
| − | 20 2 1 0 0 0 0 | + | 20 2 1 0 0 0 0 |
| − | 11 21 1 0 0 0 0 | + | 11 21 1 0 0 0 0 |
| − | 12 22 1 0 0 0 0 | + | 12 22 1 0 0 0 0 |
| − | 22 23 1 0 0 0 0 | + | 22 23 1 0 0 0 0 |
| − | 5 24 1 0 0 0 0 | + | 5 24 1 0 0 0 0 |
| − | 24 25 1 0 0 0 0 | + | 24 25 1 0 0 0 0 |
| − | 13 26 1 0 0 0 0 | + | 13 26 1 0 0 0 0 |
| − | 26 27 1 0 0 0 0 | + | 26 27 1 0 0 0 0 |
| − | 14 28 1 0 0 0 0 | + | 14 28 1 0 0 0 0 |
| − | 28 29 1 0 0 0 0 | + | 28 29 1 0 0 0 0 |
| − | M STY 1 4 SUP | + | M STY 1 4 SUP |
| − | M SLB 1 4 4 | + | M SLB 1 4 4 |
| − | M SAL 4 2 28 29 | + | M SAL 4 2 28 29 |
| − | M SBL 4 1 30 | + | M SBL 4 1 30 |
| − | M SMT 4 OCH3 | + | M SMT 4 OCH3 |
| − | M SVB 4 30 1.8057 1.2588 | + | M SVB 4 30 1.8057 1.2588 |
| − | M STY 1 3 SUP | + | M STY 1 3 SUP |
| − | M SLB 1 3 3 | + | M SLB 1 3 3 |
| − | M SAL 3 2 26 27 | + | M SAL 3 2 26 27 |
| − | M SBL 3 1 28 | + | M SBL 3 1 28 |
| − | M SMT 3 OCH3 | + | M SMT 3 OCH3 |
| − | M SVB 3 28 2.6471 0.6843 | + | M SVB 3 28 2.6471 0.6843 |
| − | M STY 1 2 SUP | + | M STY 1 2 SUP |
| − | M SLB 1 2 2 | + | M SLB 1 2 2 |
| − | M SAL 2 2 24 25 | + | M SAL 2 2 24 25 |
| − | M SBL 2 1 26 | + | M SBL 2 1 26 |
| − | M SMT 2 OCH3 | + | M SMT 2 OCH3 |
| − | M SVB 2 26 -0.7044 0.6761 | + | M SVB 2 26 -0.7044 0.6761 |
| − | M STY 1 1 SUP | + | M STY 1 1 SUP |
| − | M SLB 1 1 1 | + | M SLB 1 1 1 |
| − | M SAL 1 2 22 23 | + | M SAL 1 2 22 23 |
| − | M SBL 1 1 24 | + | M SBL 1 1 24 |
| − | M SMT 1 OCH3 | + | M SMT 1 OCH3 |
| − | M SVB 1 24 2.2899 -0.4935 | + | M SVB 1 24 2.2899 -0.4935 |
| − | S SKP 8 | + | S SKP 8 |
| − | ID FL1CELNS0003 | + | ID FL1CELNS0003 |
| − | KNApSAcK_ID C00007031 | + | KNApSAcK_ID C00007031 |
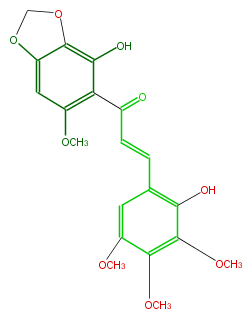
| − | NAME 2,2'-Dihydroxy-3,4,5,6'-tetramethoxy-3',4'-methylenedioxychalcone | + | NAME 2,2'-Dihydroxy-3,4,5,6'-tetramethoxy-3',4'-methylenedioxychalcone |
| − | CAS_RN 121697-07-2 | + | CAS_RN 121697-07-2 |
| − | FORMULA C20H20O9 | + | FORMULA C20H20O9 |
| − | EXACTMASS 404.11073223799997 | + | EXACTMASS 404.11073223799997 |
| − | AVERAGEMASS 404.3674 | + | AVERAGEMASS 404.3674 |
| − | SMILES COc(c3OC)cc(c(c3OC)O)C=CC(=O)c(c2OC)c(c(c1c2)OCO1)O | + | SMILES COc(c3OC)cc(c(c3OC)O)C=CC(=O)c(c2OC)c(c(c1c2)OCO1)O |
M END | M END | ||
| − | |||
Latest revision as of 09:00, 14 March 2009
Copyright: ARM project http://www.metabolome.jp/
29 31 0 0 0 0 0 0 0 0999 V2000
-2.3731 0.3551 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.3731 -0.2873 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.8168 -0.6085 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.2605 -0.2873 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.2605 0.3551 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.8168 0.6763 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.7042 -0.6085 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.1479 -0.2873 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.4082 -0.6083 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.9643 -0.2873 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.5253 -0.6112 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.0863 -0.2873 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.0863 0.3605 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.5253 0.6844 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.9643 0.3605 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.7042 -1.1098 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-1.8168 -1.2506 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-2.9840 0.5536 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-3.3616 0.0339 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.9840 -0.4858 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
1.5253 -1.2588 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
2.9523 -0.7873 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
3.8183 -1.2873 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.7044 0.6761 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
0.0101 0.2636 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.9523 0.8605 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
3.8183 1.3605 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.8057 1.2588 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
2.2444 2.1574 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1 2 1 0 0 0 0
2 3 2 0 0 0 0
3 4 1 0 0 0 0
4 5 2 0 0 0 0
5 6 1 0 0 0 0
6 1 2 0 0 0 0
4 7 1 0 0 0 0
7 8 1 0 0 0 0
8 9 2 0 0 0 0
9 10 1 0 0 0 0
10 11 2 0 0 0 0
11 12 1 0 0 0 0
12 13 2 0 0 0 0
13 14 1 0 0 0 0
14 15 2 0 0 0 0
15 10 1 0 0 0 0
7 16 2 0 0 0 0
3 17 1 0 0 0 0
1 18 1 0 0 0 0
18 19 1 0 0 0 0
19 20 1 0 0 0 0
20 2 1 0 0 0 0
11 21 1 0 0 0 0
12 22 1 0 0 0 0
22 23 1 0 0 0 0
5 24 1 0 0 0 0
24 25 1 0 0 0 0
13 26 1 0 0 0 0
26 27 1 0 0 0 0
14 28 1 0 0 0 0
28 29 1 0 0 0 0
M STY 1 4 SUP
M SLB 1 4 4
M SAL 4 2 28 29
M SBL 4 1 30
M SMT 4 OCH3
M SVB 4 30 1.8057 1.2588
M STY 1 3 SUP
M SLB 1 3 3
M SAL 3 2 26 27
M SBL 3 1 28
M SMT 3 OCH3
M SVB 3 28 2.6471 0.6843
M STY 1 2 SUP
M SLB 1 2 2
M SAL 2 2 24 25
M SBL 2 1 26
M SMT 2 OCH3
M SVB 2 26 -0.7044 0.6761
M STY 1 1 SUP
M SLB 1 1 1
M SAL 1 2 22 23
M SBL 1 1 24
M SMT 1 OCH3
M SVB 1 24 2.2899 -0.4935
S SKP 8
ID FL1CELNS0003
KNApSAcK_ID C00007031
NAME 2,2'-Dihydroxy-3,4,5,6'-tetramethoxy-3',4'-methylenedioxychalcone
CAS_RN 121697-07-2
FORMULA C20H20O9
EXACTMASS 404.11073223799997
AVERAGEMASS 404.3674
SMILES COc(c3OC)cc(c(c3OC)O)C=CC(=O)c(c2OC)c(c(c1c2)OCO1)O
M END