Mol:FL1CAAGS0008
From Metabolomics.JP
(Difference between revisions)
| Line 1: | Line 1: | ||
| − | + | ||
| − | + | ||
| − | Copyright: ARM project http://www.metabolome.jp/ | + | Copyright: ARM project http://www.metabolome.jp/ |
| − | 42 45 0 0 0 0 0 0 0 0999 V2000 | + | 42 45 0 0 0 0 0 0 0 0999 V2000 |
| − | -0.9287 0.6553 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.9287 0.6553 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.9287 -0.1697 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.9287 -0.1697 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.2142 -0.5822 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.2142 -0.5822 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.5002 -0.1697 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.5002 -0.1697 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.5002 0.6553 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.5002 0.6553 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.2142 1.0678 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.2142 1.0678 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.2147 -0.5822 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.2147 -0.5822 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.9292 -0.1697 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.9292 -0.1697 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.9292 0.6553 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.9292 0.6553 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.6437 1.0678 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.6437 1.0678 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.3581 0.6553 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.3581 0.6553 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 4.0726 1.0678 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 4.0726 1.0678 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 4.0726 1.8928 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 4.0726 1.8928 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.3581 2.3053 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.3581 2.3053 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.6437 1.8928 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.6437 1.8928 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.2147 -1.4072 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.2147 -1.4072 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.8991 -1.2624 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.8991 -1.2624 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.4212 -1.9352 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.4212 -1.9352 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.6460 -1.6521 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.6460 -1.6521 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.8348 -1.8035 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.8348 -1.8035 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.3126 -1.1306 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.3126 -1.1306 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.0878 -1.4137 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.0878 -1.4137 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.2991 -0.8452 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.2991 -0.8452 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.3348 -1.7888 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.3348 -1.7888 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.8922 -1.7194 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.8922 -1.7194 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.7567 -2.3053 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.7567 -2.3053 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.2142 -1.2934 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.2142 -1.2934 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.6154 1.0518 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.6154 1.0518 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.8823 -0.5304 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.8823 -0.5304 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.0715 0.9852 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.0715 0.9852 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -4.3181 1.0806 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -4.3181 1.0806 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.8402 0.4078 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.8402 0.4078 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.0650 0.6909 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.0650 0.6909 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.2538 0.5395 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.2538 0.5395 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.7316 1.2124 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.7316 1.2124 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.5068 0.9293 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.5068 0.9293 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.7181 1.4978 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.7181 1.4978 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -4.7538 0.5542 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -4.7538 0.5542 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -4.3112 0.6236 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -4.3112 0.6236 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.1757 0.0377 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.1757 0.0377 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -4.3013 1.8126 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -4.3013 1.8126 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 4.7538 2.2861 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 4.7538 2.2861 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1 2 2 0 0 0 0 | + | 1 2 2 0 0 0 0 |
| − | 2 3 1 0 0 0 0 | + | 2 3 1 0 0 0 0 |
| − | 3 4 2 0 0 0 0 | + | 3 4 2 0 0 0 0 |
| − | 4 5 1 0 0 0 0 | + | 4 5 1 0 0 0 0 |
| − | 5 6 2 0 0 0 0 | + | 5 6 2 0 0 0 0 |
| − | 6 1 1 0 0 0 0 | + | 6 1 1 0 0 0 0 |
| − | 4 7 1 0 0 0 0 | + | 4 7 1 0 0 0 0 |
| − | 7 8 1 0 0 0 0 | + | 7 8 1 0 0 0 0 |
| − | 8 9 2 0 0 0 0 | + | 8 9 2 0 0 0 0 |
| − | 9 10 1 0 0 0 0 | + | 9 10 1 0 0 0 0 |
| − | 10 11 2 0 0 0 0 | + | 10 11 2 0 0 0 0 |
| − | 11 12 1 0 0 0 0 | + | 11 12 1 0 0 0 0 |
| − | 12 13 2 0 0 0 0 | + | 12 13 2 0 0 0 0 |
| − | 13 14 1 0 0 0 0 | + | 13 14 1 0 0 0 0 |
| − | 14 15 2 0 0 0 0 | + | 14 15 2 0 0 0 0 |
| − | 15 10 1 0 0 0 0 | + | 15 10 1 0 0 0 0 |
| − | 7 16 2 0 0 0 0 | + | 7 16 2 0 0 0 0 |
| − | 17 18 1 1 0 0 0 | + | 17 18 1 1 0 0 0 |
| − | 18 19 1 1 0 0 0 | + | 18 19 1 1 0 0 0 |
| − | 20 19 1 1 0 0 0 | + | 20 19 1 1 0 0 0 |
| − | 20 21 1 0 0 0 0 | + | 20 21 1 0 0 0 0 |
| − | 21 22 1 0 0 0 0 | + | 21 22 1 0 0 0 0 |
| − | 22 17 1 0 0 0 0 | + | 22 17 1 0 0 0 0 |
| − | 22 23 1 0 0 0 0 | + | 22 23 1 0 0 0 0 |
| − | 17 24 1 0 0 0 0 | + | 17 24 1 0 0 0 0 |
| − | 18 25 1 0 0 0 0 | + | 18 25 1 0 0 0 0 |
| − | 19 26 1 0 0 0 0 | + | 19 26 1 0 0 0 0 |
| − | 3 27 1 0 0 0 0 | + | 3 27 1 0 0 0 0 |
| − | 1 28 1 0 0 0 0 | + | 1 28 1 0 0 0 0 |
| − | 29 23 1 0 0 0 0 | + | 29 23 1 0 0 0 0 |
| − | 5 30 1 0 0 0 0 | + | 5 30 1 0 0 0 0 |
| − | 27 20 1 0 0 0 0 | + | 27 20 1 0 0 0 0 |
| − | 31 32 1 1 0 0 0 | + | 31 32 1 1 0 0 0 |
| − | 32 33 1 1 0 0 0 | + | 32 33 1 1 0 0 0 |
| − | 34 33 1 1 0 0 0 | + | 34 33 1 1 0 0 0 |
| − | 34 35 1 0 0 0 0 | + | 34 35 1 0 0 0 0 |
| − | 35 36 1 0 0 0 0 | + | 35 36 1 0 0 0 0 |
| − | 36 31 1 0 0 0 0 | + | 36 31 1 0 0 0 0 |
| − | 36 37 1 0 0 0 0 | + | 36 37 1 0 0 0 0 |
| − | 31 38 1 0 0 0 0 | + | 31 38 1 0 0 0 0 |
| − | 32 39 1 0 0 0 0 | + | 32 39 1 0 0 0 0 |
| − | 33 40 1 0 0 0 0 | + | 33 40 1 0 0 0 0 |
| − | 41 37 1 0 0 0 0 | + | 41 37 1 0 0 0 0 |
| − | 28 34 1 0 0 0 0 | + | 28 34 1 0 0 0 0 |
| − | 13 42 1 0 0 0 0 | + | 13 42 1 0 0 0 0 |
| − | S SKP 8 | + | S SKP 8 |
| − | ID FL1CAAGS0008 | + | ID FL1CAAGS0008 |
| − | KNApSAcK_ID C00014504 | + | KNApSAcK_ID C00014504 |
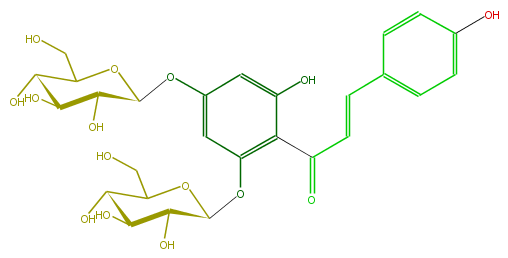
| − | NAME Chalconaringenin 2',4'-di-O-glucoside;4,2',4',6'-Tetrahydroxychalcone 2',4'-di-O-glucoside | + | NAME Chalconaringenin 2',4'-di-O-glucoside;4,2',4',6'-Tetrahydroxychalcone 2',4'-di-O-glucoside |
| − | CAS_RN 6198-23-8 | + | CAS_RN 6198-23-8 |
| − | FORMULA C27H32O15 | + | FORMULA C27H32O15 |
| − | EXACTMASS 596.174120354 | + | EXACTMASS 596.174120354 |
| − | AVERAGEMASS 596.5339799999999 | + | AVERAGEMASS 596.5339799999999 |
| − | SMILES c(c1)(c(C(C=Cc(c4)ccc(O)c4)=O)c(OC(C3O)OC(CO)C(O)C3O)cc1OC(C(O)2)OC(CO)C(O)C2O)O | + | SMILES c(c1)(c(C(C=Cc(c4)ccc(O)c4)=O)c(OC(C3O)OC(CO)C(O)C3O)cc1OC(C(O)2)OC(CO)C(O)C2O)O |
M END | M END | ||
| − | |||
Latest revision as of 09:00, 14 March 2009
Copyright: ARM project http://www.metabolome.jp/
42 45 0 0 0 0 0 0 0 0999 V2000
-0.9287 0.6553 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.9287 -0.1697 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.2142 -0.5822 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.5002 -0.1697 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.5002 0.6553 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.2142 1.0678 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.2147 -0.5822 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.9292 -0.1697 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.9292 0.6553 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.6437 1.0678 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.3581 0.6553 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
4.0726 1.0678 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
4.0726 1.8928 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.3581 2.3053 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.6437 1.8928 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.2147 -1.4072 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-2.8991 -1.2624 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.4212 -1.9352 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.6460 -1.6521 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.8348 -1.8035 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.3126 -1.1306 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-2.0878 -1.4137 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.2991 -0.8452 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-3.3348 -1.7888 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-2.8922 -1.7194 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-1.7567 -2.3053 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-0.2142 -1.2934 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-1.6154 1.0518 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-2.8823 -0.5304 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
1.0715 0.9852 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-4.3181 1.0806 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-3.8402 0.4078 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-3.0650 0.6909 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.2538 0.5395 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.7316 1.2124 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-3.5068 0.9293 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-3.7181 1.4978 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-4.7538 0.5542 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-4.3112 0.6236 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-3.1757 0.0377 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-4.3013 1.8126 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
4.7538 2.2861 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
1 2 2 0 0 0 0
2 3 1 0 0 0 0
3 4 2 0 0 0 0
4 5 1 0 0 0 0
5 6 2 0 0 0 0
6 1 1 0 0 0 0
4 7 1 0 0 0 0
7 8 1 0 0 0 0
8 9 2 0 0 0 0
9 10 1 0 0 0 0
10 11 2 0 0 0 0
11 12 1 0 0 0 0
12 13 2 0 0 0 0
13 14 1 0 0 0 0
14 15 2 0 0 0 0
15 10 1 0 0 0 0
7 16 2 0 0 0 0
17 18 1 1 0 0 0
18 19 1 1 0 0 0
20 19 1 1 0 0 0
20 21 1 0 0 0 0
21 22 1 0 0 0 0
22 17 1 0 0 0 0
22 23 1 0 0 0 0
17 24 1 0 0 0 0
18 25 1 0 0 0 0
19 26 1 0 0 0 0
3 27 1 0 0 0 0
1 28 1 0 0 0 0
29 23 1 0 0 0 0
5 30 1 0 0 0 0
27 20 1 0 0 0 0
31 32 1 1 0 0 0
32 33 1 1 0 0 0
34 33 1 1 0 0 0
34 35 1 0 0 0 0
35 36 1 0 0 0 0
36 31 1 0 0 0 0
36 37 1 0 0 0 0
31 38 1 0 0 0 0
32 39 1 0 0 0 0
33 40 1 0 0 0 0
41 37 1 0 0 0 0
28 34 1 0 0 0 0
13 42 1 0 0 0 0
S SKP 8
ID FL1CAAGS0008
KNApSAcK_ID C00014504
NAME Chalconaringenin 2',4'-di-O-glucoside;4,2',4',6'-Tetrahydroxychalcone 2',4'-di-O-glucoside
CAS_RN 6198-23-8
FORMULA C27H32O15
EXACTMASS 596.174120354
AVERAGEMASS 596.5339799999999
SMILES c(c1)(c(C(C=Cc(c4)ccc(O)c4)=O)c(OC(C3O)OC(CO)C(O)C3O)cc1OC(C(O)2)OC(CO)C(O)C2O)O
M END