Mol:FL1C9AGS0002
From Metabolomics.JP
(Difference between revisions)
| Line 1: | Line 1: | ||
| − | + | ||
| − | + | ||
| − | Copyright: ARM project http://www.metabolome.jp/ | + | Copyright: ARM project http://www.metabolome.jp/ |
| − | 30 32 0 0 0 0 0 0 0 0999 V2000 | + | 30 32 0 0 0 0 0 0 0 0999 V2000 |
| − | -3.0533 0.2062 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.0533 0.2062 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.0533 -0.6188 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.0533 -0.6188 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.3388 -1.0313 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.3388 -1.0313 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.6243 -0.6188 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.6243 -0.6188 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.6243 0.2062 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.6243 0.2062 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.3388 0.6188 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.3388 0.6188 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.9098 -1.0313 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.9098 -1.0313 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.1954 -0.6188 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.1954 -0.6188 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.1954 0.2062 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.1954 0.2062 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.5191 0.6188 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.5191 0.6188 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.2336 0.2062 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.2336 0.2062 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.9480 0.6188 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.9480 0.6188 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.9480 1.4438 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.9480 1.4438 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.2336 1.8563 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.2336 1.8563 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.5191 1.4438 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.5191 1.4438 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.9098 -1.8563 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.9098 -1.8563 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.6366 1.8413 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.6366 1.8413 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.2508 -0.7206 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.2508 -0.7206 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.0550 -0.5354 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.0550 -0.5354 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.0888 0.2892 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.0888 0.2892 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.5381 0.9814 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.5381 0.9814 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.7339 0.7963 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.7339 0.7963 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.6999 -0.0283 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.6999 -0.0283 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.0938 -0.0068 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.0938 -0.0068 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.6884 -0.3845 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.6884 -0.3845 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.5713 -1.3241 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.5713 -1.3241 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.6759 -0.8885 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.6759 -0.8885 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.6505 -0.0622 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.6505 -0.0622 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.3388 -1.7425 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.3388 -1.7425 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.6505 -0.9636 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.6505 -0.9636 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1 2 2 0 0 0 0 | + | 1 2 2 0 0 0 0 |
| − | 2 3 1 0 0 0 0 | + | 2 3 1 0 0 0 0 |
| − | 3 4 2 0 0 0 0 | + | 3 4 2 0 0 0 0 |
| − | 4 5 1 0 0 0 0 | + | 4 5 1 0 0 0 0 |
| − | 5 6 2 0 0 0 0 | + | 5 6 2 0 0 0 0 |
| − | 6 1 1 0 0 0 0 | + | 6 1 1 0 0 0 0 |
| − | 4 7 1 0 0 0 0 | + | 4 7 1 0 0 0 0 |
| − | 7 8 1 0 0 0 0 | + | 7 8 1 0 0 0 0 |
| − | 8 9 2 0 0 0 0 | + | 8 9 2 0 0 0 0 |
| − | 9 10 1 0 0 0 0 | + | 9 10 1 0 0 0 0 |
| − | 10 11 2 0 0 0 0 | + | 10 11 2 0 0 0 0 |
| − | 11 12 1 0 0 0 0 | + | 11 12 1 0 0 0 0 |
| − | 12 13 2 0 0 0 0 | + | 12 13 2 0 0 0 0 |
| − | 13 14 1 0 0 0 0 | + | 13 14 1 0 0 0 0 |
| − | 14 15 2 0 0 0 0 | + | 14 15 2 0 0 0 0 |
| − | 15 10 1 0 0 0 0 | + | 15 10 1 0 0 0 0 |
| − | 7 16 2 0 0 0 0 | + | 7 16 2 0 0 0 0 |
| − | 13 17 1 0 0 0 0 | + | 13 17 1 0 0 0 0 |
| − | 18 19 1 1 0 0 0 | + | 18 19 1 1 0 0 0 |
| − | 19 20 1 1 0 0 0 | + | 19 20 1 1 0 0 0 |
| − | 21 20 1 1 0 0 0 | + | 21 20 1 1 0 0 0 |
| − | 21 22 1 0 0 0 0 | + | 21 22 1 0 0 0 0 |
| − | 22 23 1 0 0 0 0 | + | 22 23 1 0 0 0 0 |
| − | 23 18 1 0 0 0 0 | + | 23 18 1 0 0 0 0 |
| − | 23 24 1 0 0 0 0 | + | 23 24 1 0 0 0 0 |
| − | 24 25 1 0 0 0 0 | + | 24 25 1 0 0 0 0 |
| − | 18 26 1 0 0 0 0 | + | 18 26 1 0 0 0 0 |
| − | 19 27 1 0 0 0 0 | + | 19 27 1 0 0 0 0 |
| − | 20 28 1 0 0 0 0 | + | 20 28 1 0 0 0 0 |
| − | 21 17 1 0 0 0 0 | + | 21 17 1 0 0 0 0 |
| − | 3 29 1 0 0 0 0 | + | 3 29 1 0 0 0 0 |
| − | 2 30 1 0 0 0 0 | + | 2 30 1 0 0 0 0 |
| − | S SKP 8 | + | S SKP 8 |
| − | ID FL1C9AGS0002 | + | ID FL1C9AGS0002 |
| − | KNApSAcK_ID C00014494 | + | KNApSAcK_ID C00014494 |
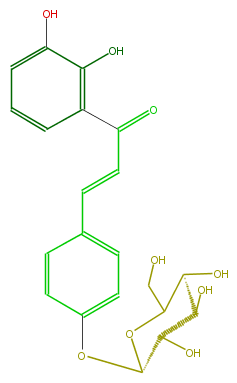
| − | NAME 4,2',3'-Trihydroxychalcone 4-O-glucoside | + | NAME 4,2',3'-Trihydroxychalcone 4-O-glucoside |
| − | CAS_RN 158500-60-8 | + | CAS_RN 158500-60-8 |
| − | FORMULA C21H22O9 | + | FORMULA C21H22O9 |
| − | EXACTMASS 418.126382302 | + | EXACTMASS 418.126382302 |
| − | AVERAGEMASS 418.39398 | + | AVERAGEMASS 418.39398 |
| − | SMILES C(Oc(c2)ccc(C=CC(=O)c(c3O)cccc(O)3)c2)(O1)C(O)C(O)C(O)C1CO | + | SMILES C(Oc(c2)ccc(C=CC(=O)c(c3O)cccc(O)3)c2)(O1)C(O)C(O)C(O)C1CO |
M END | M END | ||
| − | |||
Latest revision as of 09:00, 14 March 2009
Copyright: ARM project http://www.metabolome.jp/
30 32 0 0 0 0 0 0 0 0999 V2000
-3.0533 0.2062 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-3.0533 -0.6188 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.3388 -1.0313 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.6243 -0.6188 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.6243 0.2062 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.3388 0.6188 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.9098 -1.0313 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.1954 -0.6188 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.1954 0.2062 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.5191 0.6188 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.2336 0.2062 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.9480 0.6188 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.9480 1.4438 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.2336 1.8563 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.5191 1.4438 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.9098 -1.8563 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
2.6366 1.8413 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
2.2508 -0.7206 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.0550 -0.5354 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.0888 0.2892 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.5381 0.9814 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.7339 0.7963 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
2.6999 -0.0283 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.0938 -0.0068 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.6884 -0.3845 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
2.5713 -1.3241 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
2.6759 -0.8885 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
3.6505 -0.0622 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-2.3388 -1.7425 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-3.6505 -0.9636 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
1 2 2 0 0 0 0
2 3 1 0 0 0 0
3 4 2 0 0 0 0
4 5 1 0 0 0 0
5 6 2 0 0 0 0
6 1 1 0 0 0 0
4 7 1 0 0 0 0
7 8 1 0 0 0 0
8 9 2 0 0 0 0
9 10 1 0 0 0 0
10 11 2 0 0 0 0
11 12 1 0 0 0 0
12 13 2 0 0 0 0
13 14 1 0 0 0 0
14 15 2 0 0 0 0
15 10 1 0 0 0 0
7 16 2 0 0 0 0
13 17 1 0 0 0 0
18 19 1 1 0 0 0
19 20 1 1 0 0 0
21 20 1 1 0 0 0
21 22 1 0 0 0 0
22 23 1 0 0 0 0
23 18 1 0 0 0 0
23 24 1 0 0 0 0
24 25 1 0 0 0 0
18 26 1 0 0 0 0
19 27 1 0 0 0 0
20 28 1 0 0 0 0
21 17 1 0 0 0 0
3 29 1 0 0 0 0
2 30 1 0 0 0 0
S SKP 8
ID FL1C9AGS0002
KNApSAcK_ID C00014494
NAME 4,2',3'-Trihydroxychalcone 4-O-glucoside
CAS_RN 158500-60-8
FORMULA C21H22O9
EXACTMASS 418.126382302
AVERAGEMASS 418.39398
SMILES C(Oc(c2)ccc(C=CC(=O)c(c3O)cccc(O)3)c2)(O1)C(O)C(O)C(O)C1CO
M END