Mol:FL1C3CGS0009
From Metabolomics.JP
(Difference between revisions)
| Line 1: | Line 1: | ||
| − | + | ||
| − | + | ||
| − | Copyright: ARM project http://www.metabolome.jp/ | + | Copyright: ARM project http://www.metabolome.jp/ |
| − | 49 52 0 0 0 0 0 0 0 0999 V2000 | + | 49 52 0 0 0 0 0 0 0 0999 V2000 |
| − | -0.3479 -0.5479 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.3479 -0.5479 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.3479 -1.1876 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.3479 -1.1876 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.2061 -1.5075 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.2061 -1.5075 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.7601 -1.1876 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.7601 -1.1876 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.7601 -0.5479 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.7601 -0.5479 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.2061 -0.2281 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.2061 -0.2281 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.3138 -1.5073 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.3138 -1.5073 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.8663 -1.1883 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.8663 -1.1883 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.4176 -1.5066 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.4176 -1.5066 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.9678 -1.1890 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.9678 -1.1890 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.5062 -1.4998 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.5062 -1.4998 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 4.0445 -1.1890 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 4.0445 -1.1890 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 4.0445 -0.5674 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 4.0445 -0.5674 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.5062 -0.2565 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.5062 -0.2565 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.9678 -0.5674 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.9678 -0.5674 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.3138 -2.1453 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.3138 -2.1453 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.2061 -2.1469 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.2061 -2.1469 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.9016 -0.2282 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.9016 -0.2282 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 4.5828 -0.2566 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 4.5828 -0.2566 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.8329 -1.5581 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.8329 -1.5581 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 4.5828 -1.4998 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 4.5828 -1.4998 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.9480 -0.4453 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.9480 -0.4453 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.6017 -0.9024 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.6017 -0.9024 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.1032 -0.7085 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.1032 -0.7085 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.5835 -0.7142 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.5835 -0.7142 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.9716 -0.3536 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.9716 -0.3536 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.4810 -0.5365 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.4810 -0.5365 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.3351 -0.6688 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.3351 -0.6688 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.1415 -0.9286 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.1415 -0.9286 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.8175 -1.1880 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.8175 -1.1880 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.9264 -0.0910 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.9264 -0.0910 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.9264 0.5893 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.9264 0.5893 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.4120 1.1037 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.4120 1.1037 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.7400 1.6717 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.7400 1.6717 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.8123 1.1037 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.8123 1.1037 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.5224 1.6057 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.5224 1.6057 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.9436 1.6057 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.9436 1.6057 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.6311 1.0646 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.6311 1.0646 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.0063 1.0646 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.0063 1.0646 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.3061 1.6057 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.3061 1.6057 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.0063 2.1469 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.0063 2.1469 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.6311 2.1469 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.6311 2.1469 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.9304 1.6057 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.9304 1.6057 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -4.0556 -0.2528 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -4.0556 -0.2528 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -4.0556 0.3613 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -4.0556 0.3613 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -4.5828 -0.5572 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -4.5828 -0.5572 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.2918 -1.5523 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.2918 -1.5523 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.9368 -1.5523 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.9368 -1.5523 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.8876 -1.9565 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.8876 -1.9565 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1 2 2 0 0 0 0 | + | 1 2 2 0 0 0 0 |
| − | 2 3 1 0 0 0 0 | + | 2 3 1 0 0 0 0 |
| − | 3 4 2 0 0 0 0 | + | 3 4 2 0 0 0 0 |
| − | 4 5 1 0 0 0 0 | + | 4 5 1 0 0 0 0 |
| − | 5 6 2 0 0 0 0 | + | 5 6 2 0 0 0 0 |
| − | 6 1 1 0 0 0 0 | + | 6 1 1 0 0 0 0 |
| − | 4 7 1 0 0 0 0 | + | 4 7 1 0 0 0 0 |
| − | 7 8 1 0 0 0 0 | + | 7 8 1 0 0 0 0 |
| − | 8 9 2 0 0 0 0 | + | 8 9 2 0 0 0 0 |
| − | 9 10 1 0 0 0 0 | + | 9 10 1 0 0 0 0 |
| − | 10 11 2 0 0 0 0 | + | 10 11 2 0 0 0 0 |
| − | 11 12 1 0 0 0 0 | + | 11 12 1 0 0 0 0 |
| − | 12 13 2 0 0 0 0 | + | 12 13 2 0 0 0 0 |
| − | 13 14 1 0 0 0 0 | + | 13 14 1 0 0 0 0 |
| − | 14 15 2 0 0 0 0 | + | 14 15 2 0 0 0 0 |
| − | 15 10 1 0 0 0 0 | + | 15 10 1 0 0 0 0 |
| − | 7 16 2 0 0 0 0 | + | 7 16 2 0 0 0 0 |
| − | 3 17 1 0 0 0 0 | + | 3 17 1 0 0 0 0 |
| − | 1 18 1 0 0 0 0 | + | 1 18 1 0 0 0 0 |
| − | 13 19 1 0 0 0 0 | + | 13 19 1 0 0 0 0 |
| − | 20 2 1 0 0 0 0 | + | 20 2 1 0 0 0 0 |
| − | 12 21 1 0 0 0 0 | + | 12 21 1 0 0 0 0 |
| − | 22 23 1 1 0 0 0 | + | 22 23 1 1 0 0 0 |
| − | 23 24 1 1 0 0 0 | + | 23 24 1 1 0 0 0 |
| − | 25 24 1 1 0 0 0 | + | 25 24 1 1 0 0 0 |
| − | 25 26 1 0 0 0 0 | + | 25 26 1 0 0 0 0 |
| − | 26 27 1 0 0 0 0 | + | 26 27 1 0 0 0 0 |
| − | 27 22 1 0 0 0 0 | + | 27 22 1 0 0 0 0 |
| − | 22 28 1 0 0 0 0 | + | 22 28 1 0 0 0 0 |
| − | 23 29 1 0 0 0 0 | + | 23 29 1 0 0 0 0 |
| − | 24 30 1 0 0 0 0 | + | 24 30 1 0 0 0 0 |
| − | 27 31 1 0 0 0 0 | + | 27 31 1 0 0 0 0 |
| − | 25 18 1 0 0 0 0 | + | 25 18 1 0 0 0 0 |
| − | 31 32 1 0 0 0 0 | + | 31 32 1 0 0 0 0 |
| − | 32 33 1 0 0 0 0 | + | 32 33 1 0 0 0 0 |
| − | 33 34 2 0 0 0 0 | + | 33 34 2 0 0 0 0 |
| − | 33 35 1 0 0 0 0 | + | 33 35 1 0 0 0 0 |
| − | 35 36 2 0 0 0 0 | + | 35 36 2 0 0 0 0 |
| − | 36 37 1 0 0 0 0 | + | 36 37 1 0 0 0 0 |
| − | 37 38 2 0 0 0 0 | + | 37 38 2 0 0 0 0 |
| − | 38 39 1 0 0 0 0 | + | 38 39 1 0 0 0 0 |
| − | 39 40 2 0 0 0 0 | + | 39 40 2 0 0 0 0 |
| − | 40 41 1 0 0 0 0 | + | 40 41 1 0 0 0 0 |
| − | 41 42 2 0 0 0 0 | + | 41 42 2 0 0 0 0 |
| − | 42 37 1 0 0 0 0 | + | 42 37 1 0 0 0 0 |
| − | 40 43 1 0 0 0 0 | + | 40 43 1 0 0 0 0 |
| − | 28 44 1 0 0 0 0 | + | 28 44 1 0 0 0 0 |
| − | 44 45 2 0 0 0 0 | + | 44 45 2 0 0 0 0 |
| − | 44 46 1 0 0 0 0 | + | 44 46 1 0 0 0 0 |
| − | 47 48 2 0 0 0 0 | + | 47 48 2 0 0 0 0 |
| − | 47 49 1 0 0 0 0 | + | 47 49 1 0 0 0 0 |
| − | 47 29 1 0 0 0 0 | + | 47 29 1 0 0 0 0 |
| − | S SKP 8 | + | S SKP 8 |
| − | ID FL1C3CGS0009 | + | ID FL1C3CGS0009 |
| − | KNApSAcK_ID C00007900 | + | KNApSAcK_ID C00007900 |
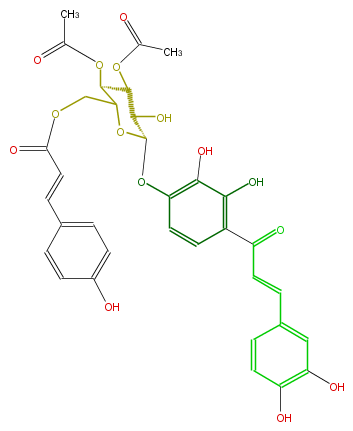
| − | NAME Okanin 4'-(3'',4''-diacetyl-6''-p-coumarylglucoside) | + | NAME Okanin 4'-(3'',4''-diacetyl-6''-p-coumarylglucoside) |
| − | CAS_RN 118853-81-9 | + | CAS_RN 118853-81-9 |
| − | FORMULA C34H32O15 | + | FORMULA C34H32O15 |
| − | EXACTMASS 680.174120354 | + | EXACTMASS 680.174120354 |
| − | AVERAGEMASS 680.60888 | + | AVERAGEMASS 680.60888 |
| − | SMILES C(C)(=O)OC(C2O)C(C(OC(Oc(c3)c(c(c(C(=O)C=Cc(c4)cc(c(O)c4)O)c3)O)O)2)COC(C=Cc(c1)ccc(O)c1)=O)OC(C)=O | + | SMILES C(C)(=O)OC(C2O)C(C(OC(Oc(c3)c(c(c(C(=O)C=Cc(c4)cc(c(O)c4)O)c3)O)O)2)COC(C=Cc(c1)ccc(O)c1)=O)OC(C)=O |
M END | M END | ||
| − | |||
Latest revision as of 09:00, 14 March 2009
Copyright: ARM project http://www.metabolome.jp/
49 52 0 0 0 0 0 0 0 0999 V2000
-0.3479 -0.5479 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.3479 -1.1876 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.2061 -1.5075 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.7601 -1.1876 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.7601 -0.5479 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.2061 -0.2281 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.3138 -1.5073 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.8663 -1.1883 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.4176 -1.5066 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.9678 -1.1890 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.5062 -1.4998 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
4.0445 -1.1890 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
4.0445 -0.5674 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.5062 -0.2565 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.9678 -0.5674 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.3138 -2.1453 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
0.2061 -2.1469 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-0.9016 -0.2282 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
4.5828 -0.2566 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-0.8329 -1.5581 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
4.5828 -1.4998 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-2.9480 -0.4453 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.6017 -0.9024 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.1032 -0.7085 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.5835 -0.7142 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.9716 -0.3536 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-2.4810 -0.5365 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-3.3351 -0.6688 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-3.1415 -0.9286 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-1.8175 -1.1880 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-2.9264 -0.0910 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.9264 0.5893 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-2.4120 1.1037 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.7400 1.6717 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-1.8123 1.1037 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.5224 1.6057 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.9436 1.6057 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.6311 1.0646 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.0063 1.0646 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.3061 1.6057 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.0063 2.1469 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.6311 2.1469 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.9304 1.6057 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-4.0556 -0.2528 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-4.0556 0.3613 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-4.5828 -0.5572 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-3.2918 -1.5523 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-3.9368 -1.5523 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-2.8876 -1.9565 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1 2 2 0 0 0 0
2 3 1 0 0 0 0
3 4 2 0 0 0 0
4 5 1 0 0 0 0
5 6 2 0 0 0 0
6 1 1 0 0 0 0
4 7 1 0 0 0 0
7 8 1 0 0 0 0
8 9 2 0 0 0 0
9 10 1 0 0 0 0
10 11 2 0 0 0 0
11 12 1 0 0 0 0
12 13 2 0 0 0 0
13 14 1 0 0 0 0
14 15 2 0 0 0 0
15 10 1 0 0 0 0
7 16 2 0 0 0 0
3 17 1 0 0 0 0
1 18 1 0 0 0 0
13 19 1 0 0 0 0
20 2 1 0 0 0 0
12 21 1 0 0 0 0
22 23 1 1 0 0 0
23 24 1 1 0 0 0
25 24 1 1 0 0 0
25 26 1 0 0 0 0
26 27 1 0 0 0 0
27 22 1 0 0 0 0
22 28 1 0 0 0 0
23 29 1 0 0 0 0
24 30 1 0 0 0 0
27 31 1 0 0 0 0
25 18 1 0 0 0 0
31 32 1 0 0 0 0
32 33 1 0 0 0 0
33 34 2 0 0 0 0
33 35 1 0 0 0 0
35 36 2 0 0 0 0
36 37 1 0 0 0 0
37 38 2 0 0 0 0
38 39 1 0 0 0 0
39 40 2 0 0 0 0
40 41 1 0 0 0 0
41 42 2 0 0 0 0
42 37 1 0 0 0 0
40 43 1 0 0 0 0
28 44 1 0 0 0 0
44 45 2 0 0 0 0
44 46 1 0 0 0 0
47 48 2 0 0 0 0
47 49 1 0 0 0 0
47 29 1 0 0 0 0
S SKP 8
ID FL1C3CGS0009
KNApSAcK_ID C00007900
NAME Okanin 4'-(3'',4''-diacetyl-6''-p-coumarylglucoside)
CAS_RN 118853-81-9
FORMULA C34H32O15
EXACTMASS 680.174120354
AVERAGEMASS 680.60888
SMILES C(C)(=O)OC(C2O)C(C(OC(Oc(c3)c(c(c(C(=O)C=Cc(c4)cc(c(O)c4)O)c3)O)O)2)COC(C=Cc(c1)ccc(O)c1)=O)OC(C)=O
M END