Mol:FL1C1LNI0005
From Metabolomics.JP
(Difference between revisions)
| Line 1: | Line 1: | ||
| − | + | ||
| − | + | ||
| − | Copyright: ARM project http://www.metabolome.jp/ | + | Copyright: ARM project http://www.metabolome.jp/ |
| − | 39 41 0 0 0 0 0 0 0 0999 V2000 | + | 39 41 0 0 0 0 0 0 0 0999 V2000 |
| − | 1.4289 2.2687 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.4289 2.2687 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.7145 1.8562 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.7145 1.8562 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.7145 1.0312 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.7145 1.0312 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.4289 0.6187 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.4289 0.6187 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.1434 1.0313 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.1434 1.0313 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.1434 1.8563 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.1434 1.8563 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.4289 -0.2062 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.4289 -0.2062 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.7145 -0.6188 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.7145 -0.6188 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.1434 -0.6188 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.1434 -0.6188 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.1434 -1.4438 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.1434 -1.4438 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.8579 -1.8563 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.8579 -1.8563 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.8579 -2.6812 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.8579 -2.6812 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.5724 -3.0938 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.5724 -3.0938 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 4.2868 -2.6812 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 4.2868 -2.6812 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 4.2868 -1.8563 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 4.2868 -1.8563 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.5724 -1.4438 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.5724 -1.4438 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 5.0013 -3.0938 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 5.0013 -3.0938 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.1434 -3.0938 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.1434 -3.0938 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.0000 0.6188 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.0000 0.6188 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.4289 3.0938 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.4289 3.0938 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.0000 2.2687 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.0000 2.2687 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.7145 1.8562 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.7145 1.8562 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.4289 2.2687 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.4289 2.2687 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.4289 3.0937 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.4289 3.0937 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.1434 1.8562 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.1434 1.8562 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.8579 2.2687 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.8579 2.2687 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.5724 1.8562 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.5724 1.8562 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -4.2868 2.2687 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -4.2868 2.2687 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.5724 1.0312 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.5724 1.0312 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -4.2868 0.6187 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -4.2868 0.6187 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -4.2868 -0.2063 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -4.2868 -0.2063 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -5.0013 -0.6188 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -5.0013 -0.6188 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -5.0013 -1.4438 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -5.0013 -1.4438 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -4.2868 -1.8563 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -4.2868 -1.8563 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.5724 -1.4438 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.5724 -1.4438 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.5724 -0.6188 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.5724 -0.6188 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -4.2868 -2.6813 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -4.2868 -2.6813 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.8579 -1.8563 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.8579 -1.8563 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.8579 -2.6813 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.8579 -2.6813 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1 2 2 0 0 0 0 | + | 1 2 2 0 0 0 0 |
| − | 2 3 1 0 0 0 0 | + | 2 3 1 0 0 0 0 |
| − | 3 4 2 0 0 0 0 | + | 3 4 2 0 0 0 0 |
| − | 4 5 1 0 0 0 0 | + | 4 5 1 0 0 0 0 |
| − | 5 6 2 0 0 0 0 | + | 5 6 2 0 0 0 0 |
| − | 6 1 1 0 0 0 0 | + | 6 1 1 0 0 0 0 |
| − | 4 7 1 0 0 0 0 | + | 4 7 1 0 0 0 0 |
| − | 7 8 2 0 0 0 0 | + | 7 8 2 0 0 0 0 |
| − | 7 9 1 0 0 0 0 | + | 7 9 1 0 0 0 0 |
| − | 9 10 2 0 0 0 0 | + | 9 10 2 0 0 0 0 |
| − | 10 11 1 0 0 0 0 | + | 10 11 1 0 0 0 0 |
| − | 11 12 2 0 0 0 0 | + | 11 12 2 0 0 0 0 |
| − | 12 13 1 0 0 0 0 | + | 12 13 1 0 0 0 0 |
| − | 13 14 2 0 0 0 0 | + | 13 14 2 0 0 0 0 |
| − | 14 15 1 0 0 0 0 | + | 14 15 1 0 0 0 0 |
| − | 15 16 2 0 0 0 0 | + | 15 16 2 0 0 0 0 |
| − | 16 11 1 0 0 0 0 | + | 16 11 1 0 0 0 0 |
| − | 14 17 1 0 0 0 0 | + | 14 17 1 0 0 0 0 |
| − | 12 18 1 0 0 0 0 | + | 12 18 1 0 0 0 0 |
| − | 3 19 1 0 0 0 0 | + | 3 19 1 0 0 0 0 |
| − | 1 20 1 0 0 0 0 | + | 1 20 1 0 0 0 0 |
| − | 2 21 1 0 0 0 0 | + | 2 21 1 0 0 0 0 |
| − | 21 22 1 0 0 0 0 | + | 21 22 1 0 0 0 0 |
| − | 22 23 2 0 0 0 0 | + | 22 23 2 0 0 0 0 |
| − | 23 24 1 0 0 0 0 | + | 23 24 1 0 0 0 0 |
| − | 23 25 1 0 0 0 0 | + | 23 25 1 0 0 0 0 |
| − | 25 26 1 0 0 0 0 | + | 25 26 1 0 0 0 0 |
| − | 26 27 1 0 0 0 0 | + | 26 27 1 0 0 0 0 |
| − | 27 28 2 0 0 0 0 | + | 27 28 2 0 0 0 0 |
| − | 27 29 1 0 0 0 0 | + | 27 29 1 0 0 0 0 |
| − | 29 30 2 0 0 0 0 | + | 29 30 2 0 0 0 0 |
| − | 30 31 1 0 0 0 0 | + | 30 31 1 0 0 0 0 |
| − | 31 32 2 0 0 0 0 | + | 31 32 2 0 0 0 0 |
| − | 32 33 1 0 0 0 0 | + | 32 33 1 0 0 0 0 |
| − | 33 34 2 0 0 0 0 | + | 33 34 2 0 0 0 0 |
| − | 34 35 1 0 0 0 0 | + | 34 35 1 0 0 0 0 |
| − | 35 36 2 0 0 0 0 | + | 35 36 2 0 0 0 0 |
| − | 36 31 1 0 0 0 0 | + | 36 31 1 0 0 0 0 |
| − | 34 37 1 0 0 0 0 | + | 34 37 1 0 0 0 0 |
| − | 35 38 1 0 0 0 0 | + | 35 38 1 0 0 0 0 |
| − | 38 39 1 0 0 0 0 | + | 38 39 1 0 0 0 0 |
| − | S SKP 8 | + | S SKP 8 |
| − | ID FL1C1LNI0005 | + | ID FL1C1LNI0005 |
| − | KNApSAcK_ID C00014482 | + | KNApSAcK_ID C00014482 |
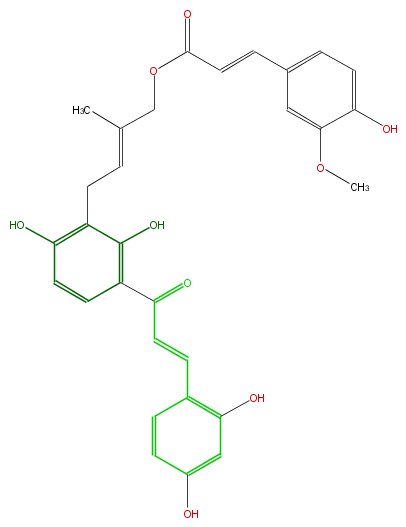
| − | NAME Isogemichalcone C;3'-(4-Feruloyloxy-3-methylbutyl-2(E)-enyl)-2,4,2',4'-tetrahydroxychalcone | + | NAME Isogemichalcone C;3'-(4-Feruloyloxy-3-methylbutyl-2(E)-enyl)-2,4,2',4'-tetrahydroxychalcone |
| − | CAS_RN 376590-14-6 | + | CAS_RN 376590-14-6 |
| − | FORMULA C30H28O9 | + | FORMULA C30H28O9 |
| − | EXACTMASS 532.173332494 | + | EXACTMASS 532.173332494 |
| − | AVERAGEMASS 532.53792 | + | AVERAGEMASS 532.53792 |
| − | SMILES c(O)(c(CC=C(C)COC(C=Cc(c3)cc(OC)c(O)c3)=O)2)c(ccc(O)2)C(=O)C=Cc(c1O)ccc(c1)O | + | SMILES c(O)(c(CC=C(C)COC(C=Cc(c3)cc(OC)c(O)c3)=O)2)c(ccc(O)2)C(=O)C=Cc(c1O)ccc(c1)O |
M END | M END | ||
| − | |||
Latest revision as of 09:00, 14 March 2009
Copyright: ARM project http://www.metabolome.jp/
39 41 0 0 0 0 0 0 0 0999 V2000
1.4289 2.2687 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.7145 1.8562 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.7145 1.0312 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.4289 0.6187 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.1434 1.0313 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.1434 1.8563 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.4289 -0.2062 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.7145 -0.6188 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
2.1434 -0.6188 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.1434 -1.4438 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.8579 -1.8563 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.8579 -2.6812 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.5724 -3.0938 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
4.2868 -2.6812 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
4.2868 -1.8563 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.5724 -1.4438 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
5.0013 -3.0938 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
2.1434 -3.0938 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
0.0000 0.6188 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
1.4289 3.0938 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
0.0000 2.2687 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.7145 1.8562 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.4289 2.2687 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.4289 3.0937 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.1434 1.8562 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.8579 2.2687 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-3.5724 1.8562 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-4.2868 2.2687 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-3.5724 1.0312 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-4.2868 0.6187 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-4.2868 -0.2063 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-5.0013 -0.6188 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-5.0013 -1.4438 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-4.2868 -1.8563 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-3.5724 -1.4438 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-3.5724 -0.6188 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-4.2868 -2.6813 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-2.8579 -1.8563 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-2.8579 -2.6813 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1 2 2 0 0 0 0
2 3 1 0 0 0 0
3 4 2 0 0 0 0
4 5 1 0 0 0 0
5 6 2 0 0 0 0
6 1 1 0 0 0 0
4 7 1 0 0 0 0
7 8 2 0 0 0 0
7 9 1 0 0 0 0
9 10 2 0 0 0 0
10 11 1 0 0 0 0
11 12 2 0 0 0 0
12 13 1 0 0 0 0
13 14 2 0 0 0 0
14 15 1 0 0 0 0
15 16 2 0 0 0 0
16 11 1 0 0 0 0
14 17 1 0 0 0 0
12 18 1 0 0 0 0
3 19 1 0 0 0 0
1 20 1 0 0 0 0
2 21 1 0 0 0 0
21 22 1 0 0 0 0
22 23 2 0 0 0 0
23 24 1 0 0 0 0
23 25 1 0 0 0 0
25 26 1 0 0 0 0
26 27 1 0 0 0 0
27 28 2 0 0 0 0
27 29 1 0 0 0 0
29 30 2 0 0 0 0
30 31 1 0 0 0 0
31 32 2 0 0 0 0
32 33 1 0 0 0 0
33 34 2 0 0 0 0
34 35 1 0 0 0 0
35 36 2 0 0 0 0
36 31 1 0 0 0 0
34 37 1 0 0 0 0
35 38 1 0 0 0 0
38 39 1 0 0 0 0
S SKP 8
ID FL1C1LNI0005
KNApSAcK_ID C00014482
NAME Isogemichalcone C;3'-(4-Feruloyloxy-3-methylbutyl-2(E)-enyl)-2,4,2',4'-tetrahydroxychalcone
CAS_RN 376590-14-6
FORMULA C30H28O9
EXACTMASS 532.173332494
AVERAGEMASS 532.53792
SMILES c(O)(c(CC=C(C)COC(C=Cc(c3)cc(OC)c(O)c3)=O)2)c(ccc(O)2)C(=O)C=Cc(c1O)ccc(c1)O
M END