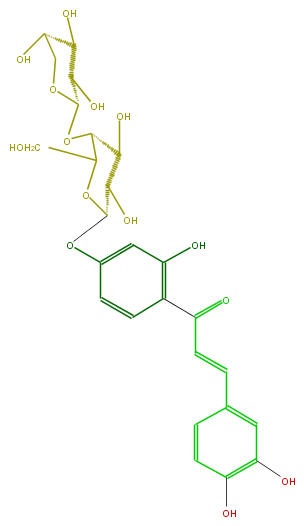
Mol:FL1C1CGS0003
From Metabolomics.JP
(Difference between revisions)
| Line 1: | Line 1: | ||
| − | + | ||
| − | + | ||
| − | Copyright: ARM project http://www.metabolome.jp/ | + | Copyright: ARM project http://www.metabolome.jp/ |
| − | 40 43 0 0 0 0 0 0 0 0999 V2000 | + | 40 43 0 0 0 0 0 0 0 0999 V2000 |
| − | -0.5358 0.7770 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.5358 0.7770 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.5358 -0.0720 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.5358 -0.0720 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.1995 -0.4964 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.1995 -0.4964 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.9345 -0.0720 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.9345 -0.0720 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.9345 0.7770 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.9345 0.7770 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.1995 1.2014 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.1995 1.2014 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.6692 -0.4963 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.6692 -0.4963 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.4025 -0.0729 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.4025 -0.0729 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.1341 -0.4954 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.1341 -0.4954 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.8640 -0.0737 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.8640 -0.0737 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 4.5785 -0.4864 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 4.5785 -0.4864 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 5.2929 -0.0737 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 5.2929 -0.0737 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 5.2929 0.7512 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 5.2929 0.7512 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 4.5785 1.1637 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 4.5785 1.1637 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.8640 0.7512 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.8640 0.7512 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.6692 -1.3429 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.6692 -1.3429 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 5.9898 1.1535 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 5.9898 1.1535 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.1995 -1.3449 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.1995 -1.3449 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.2705 1.2011 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.2705 1.2011 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 6.0266 -0.4155 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 6.0266 -0.4155 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.2718 -0.5396 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.2718 -0.5396 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.5052 -0.8627 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.5052 -0.8627 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.0194 -0.2574 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.0194 -0.2574 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.4187 0.0979 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.4187 0.0979 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.1115 0.2836 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.1115 0.2836 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.6527 -0.3367 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.6527 -0.3367 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.4294 0.0325 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.4294 0.0325 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.3165 -1.3311 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.3165 -1.3311 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.1120 -0.2574 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.1120 -0.2574 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -5.8427 -0.7500 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -5.8427 -0.7500 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -5.2420 -1.2170 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -5.2420 -1.2170 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -4.6696 -0.7972 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -4.6696 -0.7972 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -4.0015 -0.6261 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -4.0015 -0.6261 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -4.6228 -0.2972 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -4.6228 -0.2972 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -5.2129 -0.7065 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -5.2129 -0.7065 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -5.6357 0.0225 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -5.6357 0.0225 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -6.0266 -1.4580 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -6.0266 -1.4580 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.8094 -0.9296 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.8094 -0.9296 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.5709 0.5815 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.5709 0.5815 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.8059 1.4580 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.8059 1.4580 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1 2 2 0 0 0 0 | + | 1 2 2 0 0 0 0 |
| − | 2 3 1 0 0 0 0 | + | 2 3 1 0 0 0 0 |
| − | 3 4 2 0 0 0 0 | + | 3 4 2 0 0 0 0 |
| − | 4 5 1 0 0 0 0 | + | 4 5 1 0 0 0 0 |
| − | 5 6 2 0 0 0 0 | + | 5 6 2 0 0 0 0 |
| − | 6 1 1 0 0 0 0 | + | 6 1 1 0 0 0 0 |
| − | 4 7 1 0 0 0 0 | + | 4 7 1 0 0 0 0 |
| − | 7 8 1 0 0 0 0 | + | 7 8 1 0 0 0 0 |
| − | 8 9 2 0 0 0 0 | + | 8 9 2 0 0 0 0 |
| − | 9 10 1 0 0 0 0 | + | 9 10 1 0 0 0 0 |
| − | 10 11 2 0 0 0 0 | + | 10 11 2 0 0 0 0 |
| − | 11 12 1 0 0 0 0 | + | 11 12 1 0 0 0 0 |
| − | 12 13 2 0 0 0 0 | + | 12 13 2 0 0 0 0 |
| − | 13 14 1 0 0 0 0 | + | 13 14 1 0 0 0 0 |
| − | 14 15 2 0 0 0 0 | + | 14 15 2 0 0 0 0 |
| − | 15 10 1 0 0 0 0 | + | 15 10 1 0 0 0 0 |
| − | 7 16 2 0 0 0 0 | + | 7 16 2 0 0 0 0 |
| − | 17 13 1 0 0 0 0 | + | 17 13 1 0 0 0 0 |
| − | 3 18 1 0 0 0 0 | + | 3 18 1 0 0 0 0 |
| − | 1 19 1 0 0 0 0 | + | 1 19 1 0 0 0 0 |
| − | 12 20 1 0 0 0 0 | + | 12 20 1 0 0 0 0 |
| − | 21 22 1 1 0 0 0 | + | 21 22 1 1 0 0 0 |
| − | 22 23 1 1 0 0 0 | + | 22 23 1 1 0 0 0 |
| − | 24 23 1 1 0 0 0 | + | 24 23 1 1 0 0 0 |
| − | 24 25 1 0 0 0 0 | + | 24 25 1 0 0 0 0 |
| − | 25 26 1 0 0 0 0 | + | 25 26 1 0 0 0 0 |
| − | 26 21 1 0 0 0 0 | + | 26 21 1 0 0 0 0 |
| − | 21 27 1 0 0 0 0 | + | 21 27 1 0 0 0 0 |
| − | 22 28 1 0 0 0 0 | + | 22 28 1 0 0 0 0 |
| − | 23 29 1 0 0 0 0 | + | 23 29 1 0 0 0 0 |
| − | 24 19 1 0 0 0 0 | + | 24 19 1 0 0 0 0 |
| − | 30 31 1 1 0 0 0 | + | 30 31 1 1 0 0 0 |
| − | 31 32 1 1 0 0 0 | + | 31 32 1 1 0 0 0 |
| − | 33 32 1 1 0 0 0 | + | 33 32 1 1 0 0 0 |
| − | 33 34 1 0 0 0 0 | + | 33 34 1 0 0 0 0 |
| − | 34 35 1 0 0 0 0 | + | 34 35 1 0 0 0 0 |
| − | 35 30 1 0 0 0 0 | + | 35 30 1 0 0 0 0 |
| − | 30 36 1 0 0 0 0 | + | 30 36 1 0 0 0 0 |
| − | 31 37 1 0 0 0 0 | + | 31 37 1 0 0 0 0 |
| − | 33 27 1 0 0 0 0 | + | 33 27 1 0 0 0 0 |
| − | 32 38 1 0 0 0 0 | + | 32 38 1 0 0 0 0 |
| − | 39 40 1 0 0 0 0 | + | 39 40 1 0 0 0 0 |
| − | 26 39 1 0 0 0 0 | + | 26 39 1 0 0 0 0 |
| − | M STY 1 1 SUP | + | M STY 1 1 SUP |
| − | M SLB 1 1 1 | + | M SLB 1 1 1 |
| − | M SAL 1 2 39 40 | + | M SAL 1 2 39 40 |
| − | M SBL 1 1 43 | + | M SBL 1 1 43 |
| − | M SMT 1 ^ CH2OH | + | M SMT 1 ^ CH2OH |
| − | M SBV 1 43 0.9182 -0.9182 | + | M SBV 1 43 0.9182 -0.9182 |
| − | S SKP 5 | + | S SKP 5 |
| − | ID FL1C1CGS0003 | + | ID FL1C1CGS0003 |
| − | FORMULA C26H30O14 | + | FORMULA C26H30O14 |
| − | EXACTMASS 566.163555668 | + | EXACTMASS 566.163555668 |
| − | AVERAGEMASS 566.508 | + | AVERAGEMASS 566.508 |
| − | SMILES C(OC(C4O)C(CO)OC(C(O)4)Oc(c2)cc(O)c(C(=O)C=Cc(c3)ccc(c3O)O)c2)(C(O)1)OCC(O)C1O | + | SMILES C(OC(C4O)C(CO)OC(C(O)4)Oc(c2)cc(O)c(C(=O)C=Cc(c3)ccc(c3O)O)c2)(C(O)1)OCC(O)C1O |
M END | M END | ||
| − | |||
Latest revision as of 09:00, 14 March 2009
Copyright: ARM project http://www.metabolome.jp/
40 43 0 0 0 0 0 0 0 0999 V2000
-0.5358 0.7770 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.5358 -0.0720 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.1995 -0.4964 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.9345 -0.0720 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.9345 0.7770 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.1995 1.2014 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.6692 -0.4963 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.4025 -0.0729 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.1341 -0.4954 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.8640 -0.0737 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
4.5785 -0.4864 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
5.2929 -0.0737 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
5.2929 0.7512 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
4.5785 1.1637 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.8640 0.7512 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.6692 -1.3429 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
5.9898 1.1535 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
0.1995 -1.3449 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-1.2705 1.2011 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
6.0266 -0.4155 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-3.2718 -0.5396 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.5052 -0.8627 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.0194 -0.2574 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.4187 0.0979 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.1115 0.2836 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-2.6527 -0.3367 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-3.4294 0.0325 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-3.3165 -1.3311 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-1.1120 -0.2574 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-5.8427 -0.7500 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-5.2420 -1.2170 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-4.6696 -0.7972 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-4.0015 -0.6261 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-4.6228 -0.2972 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-5.2129 -0.7065 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-5.6357 0.0225 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-6.0266 -1.4580 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-3.8094 -0.9296 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-3.5709 0.5815 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-3.8059 1.4580 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
1 2 2 0 0 0 0
2 3 1 0 0 0 0
3 4 2 0 0 0 0
4 5 1 0 0 0 0
5 6 2 0 0 0 0
6 1 1 0 0 0 0
4 7 1 0 0 0 0
7 8 1 0 0 0 0
8 9 2 0 0 0 0
9 10 1 0 0 0 0
10 11 2 0 0 0 0
11 12 1 0 0 0 0
12 13 2 0 0 0 0
13 14 1 0 0 0 0
14 15 2 0 0 0 0
15 10 1 0 0 0 0
7 16 2 0 0 0 0
17 13 1 0 0 0 0
3 18 1 0 0 0 0
1 19 1 0 0 0 0
12 20 1 0 0 0 0
21 22 1 1 0 0 0
22 23 1 1 0 0 0
24 23 1 1 0 0 0
24 25 1 0 0 0 0
25 26 1 0 0 0 0
26 21 1 0 0 0 0
21 27 1 0 0 0 0
22 28 1 0 0 0 0
23 29 1 0 0 0 0
24 19 1 0 0 0 0
30 31 1 1 0 0 0
31 32 1 1 0 0 0
33 32 1 1 0 0 0
33 34 1 0 0 0 0
34 35 1 0 0 0 0
35 30 1 0 0 0 0
30 36 1 0 0 0 0
31 37 1 0 0 0 0
33 27 1 0 0 0 0
32 38 1 0 0 0 0
39 40 1 0 0 0 0
26 39 1 0 0 0 0
M STY 1 1 SUP
M SLB 1 1 1
M SAL 1 2 39 40
M SBL 1 1 43
M SMT 1 ^ CH2OH
M SBV 1 43 0.9182 -0.9182
S SKP 5
ID FL1C1CGS0003
FORMULA C26H30O14
EXACTMASS 566.163555668
AVERAGEMASS 566.508
SMILES C(OC(C4O)C(CO)OC(C(O)4)Oc(c2)cc(O)c(C(=O)C=Cc(c3)ccc(c3O)O)c2)(C(O)1)OCC(O)C1O
M END