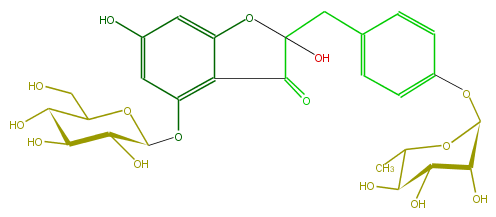
Mol:FL1BAAGS0003
From Metabolomics.JP
(Difference between revisions)
| Line 1: | Line 1: | ||
| − | + | ||
| − | + | ||
| − | Copyright: ARM project http://www.metabolome.jp/ | + | Copyright: ARM project http://www.metabolome.jp/ |
| − | 42 46 0 0 0 0 0 0 0 0999 V2000 | + | 42 46 0 0 0 0 0 0 0 0999 V2000 |
| − | -2.1561 1.3926 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.1561 1.3926 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.1561 0.5676 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.1561 0.5676 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.4417 0.1552 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.4417 0.1552 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.4417 1.8052 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.4417 1.8052 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.7272 0.5676 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.7272 0.5676 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.7272 1.3926 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.7272 1.3926 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.0129 1.8067 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.0129 1.8067 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.7034 1.3914 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.7034 1.3914 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.7025 0.5674 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.7025 0.5674 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.4789 1.9207 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.4789 1.9207 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.2428 1.4570 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.2428 1.4570 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.2538 0.6315 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.2538 0.6315 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.9739 0.2289 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.9739 0.2289 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.6826 0.6512 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.6826 0.6512 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.6713 1.4759 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.6713 1.4759 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.9513 1.8787 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.9513 1.8787 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.4417 -0.5708 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.4417 -0.5708 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 4.3323 0.2877 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 4.3323 0.2877 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.7922 1.7600 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.7922 1.7600 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.1243 0.1457 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.1243 0.1457 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.3739 1.0043 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.3739 1.0043 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -4.0439 -0.0320 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -4.0439 -0.0320 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.6313 -0.7467 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.6313 -0.7467 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.8330 -0.5376 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.8330 -0.5376 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.0395 -0.7644 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.0395 -0.7644 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.4520 -0.0496 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.4520 -0.0496 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.2504 -0.2587 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.2504 -0.2587 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.5801 0.2723 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.5801 0.2723 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -4.2173 0.4392 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -4.2173 0.4392 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -4.5919 -0.3428 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -4.5919 -0.3428 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -4.2230 -0.6747 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -4.2230 -0.6747 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.2496 -1.1209 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.2496 -1.1209 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.1021 -0.8447 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.1021 -0.8447 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.9588 -1.6574 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.9588 -1.6574 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.6269 -1.1730 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.6269 -1.1730 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 4.3980 -1.2113 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 4.3980 -1.2113 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 4.5919 -0.2838 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 4.5919 -0.2838 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.9239 -0.7682 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.9239 -0.7682 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 4.5252 -1.8119 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 4.5252 -1.8119 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.2782 -1.9207 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.2782 -1.9207 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.4164 -1.5618 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.4164 -1.5618 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.5830 -1.2082 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.5830 -1.2082 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1 2 2 0 0 0 0 | + | 1 2 2 0 0 0 0 |
| − | 2 3 1 0 0 0 0 | + | 2 3 1 0 0 0 0 |
| − | 3 5 2 0 0 0 0 | + | 3 5 2 0 0 0 0 |
| − | 6 4 2 0 0 0 0 | + | 6 4 2 0 0 0 0 |
| − | 4 1 1 0 0 0 0 | + | 4 1 1 0 0 0 0 |
| − | 5 6 1 0 0 0 0 | + | 5 6 1 0 0 0 0 |
| − | 6 7 1 0 0 0 0 | + | 6 7 1 0 0 0 0 |
| − | 7 8 1 0 0 0 0 | + | 7 8 1 0 0 0 0 |
| − | 8 9 1 0 0 0 0 | + | 8 9 1 0 0 0 0 |
| − | 5 9 1 0 0 0 0 | + | 5 9 1 0 0 0 0 |
| − | 8 10 1 0 0 0 0 | + | 8 10 1 0 0 0 0 |
| − | 10 11 1 0 0 0 0 | + | 10 11 1 0 0 0 0 |
| − | 11 12 2 0 0 0 0 | + | 11 12 2 0 0 0 0 |
| − | 12 13 1 0 0 0 0 | + | 12 13 1 0 0 0 0 |
| − | 13 14 2 0 0 0 0 | + | 13 14 2 0 0 0 0 |
| − | 14 15 1 0 0 0 0 | + | 14 15 1 0 0 0 0 |
| − | 15 16 2 0 0 0 0 | + | 15 16 2 0 0 0 0 |
| − | 16 11 1 0 0 0 0 | + | 16 11 1 0 0 0 0 |
| − | 3 17 1 0 0 0 0 | + | 3 17 1 0 0 0 0 |
| − | 14 18 1 0 0 0 0 | + | 14 18 1 0 0 0 0 |
| − | 1 19 1 0 0 0 0 | + | 1 19 1 0 0 0 0 |
| − | 9 20 2 0 0 0 0 | + | 9 20 2 0 0 0 0 |
| − | 8 21 1 0 0 0 0 | + | 8 21 1 0 0 0 0 |
| − | 22 23 1 1 0 0 0 | + | 22 23 1 1 0 0 0 |
| − | 23 24 1 1 0 0 0 | + | 23 24 1 1 0 0 0 |
| − | 25 24 1 1 0 0 0 | + | 25 24 1 1 0 0 0 |
| − | 25 26 1 0 0 0 0 | + | 25 26 1 0 0 0 0 |
| − | 26 27 1 0 0 0 0 | + | 26 27 1 0 0 0 0 |
| − | 27 22 1 0 0 0 0 | + | 27 22 1 0 0 0 0 |
| − | 27 28 1 0 0 0 0 | + | 27 28 1 0 0 0 0 |
| − | 28 29 1 0 0 0 0 | + | 28 29 1 0 0 0 0 |
| − | 22 30 1 0 0 0 0 | + | 22 30 1 0 0 0 0 |
| − | 23 31 1 0 0 0 0 | + | 23 31 1 0 0 0 0 |
| − | 24 32 1 0 0 0 0 | + | 24 32 1 0 0 0 0 |
| − | 17 25 1 0 0 0 0 | + | 17 25 1 0 0 0 0 |
| − | 33 34 1 0 0 0 0 | + | 33 34 1 0 0 0 0 |
| − | 34 35 1 1 0 0 0 | + | 34 35 1 1 0 0 0 |
| − | 36 35 1 1 0 0 0 | + | 36 35 1 1 0 0 0 |
| − | 37 36 1 1 0 0 0 | + | 37 36 1 1 0 0 0 |
| − | 37 38 1 0 0 0 0 | + | 37 38 1 0 0 0 0 |
| − | 38 33 1 0 0 0 0 | + | 38 33 1 0 0 0 0 |
| − | 36 39 1 0 0 0 0 | + | 36 39 1 0 0 0 0 |
| − | 35 40 1 0 0 0 0 | + | 35 40 1 0 0 0 0 |
| − | 34 41 1 0 0 0 0 | + | 34 41 1 0 0 0 0 |
| − | 33 42 1 0 0 0 0 | + | 33 42 1 0 0 0 0 |
| − | 37 18 1 0 0 0 0 | + | 37 18 1 0 0 0 0 |
| − | S SKP 5 | + | S SKP 5 |
| − | ID FL1BAAGS0003 | + | ID FL1BAAGS0003 |
| − | FORMULA C27H32O15 | + | FORMULA C27H32O15 |
| − | EXACTMASS 596.174120354 | + | EXACTMASS 596.174120354 |
| − | AVERAGEMASS 596.5339799999999 | + | AVERAGEMASS 596.5339799999999 |
| − | SMILES O(C1Oc(c5)ccc(c5)CC(C4=O)(O)Oc(c42)cc(cc2OC(C3O)OC(CO)C(C3O)O)O)C(C(C(O)C(O)1)O)C | + | SMILES O(C1Oc(c5)ccc(c5)CC(C4=O)(O)Oc(c42)cc(cc2OC(C3O)OC(CO)C(C3O)O)O)C(C(C(O)C(O)1)O)C |
M END | M END | ||
| − | |||
Latest revision as of 09:00, 14 March 2009
Copyright: ARM project http://www.metabolome.jp/
42 46 0 0 0 0 0 0 0 0999 V2000
-2.1561 1.3926 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.1561 0.5676 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.4417 0.1552 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.4417 1.8052 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.7272 0.5676 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.7272 1.3926 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.0129 1.8067 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
0.7034 1.3914 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.7025 0.5674 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.4789 1.9207 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.2428 1.4570 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.2538 0.6315 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.9739 0.2289 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.6826 0.6512 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.6713 1.4759 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.9513 1.8787 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.4417 -0.5708 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
4.3323 0.2877 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-2.7922 1.7600 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
1.1243 0.1457 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
1.3739 1.0043 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-4.0439 -0.0320 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-3.6313 -0.7467 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.8330 -0.5376 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.0395 -0.7644 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.4520 -0.0496 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-3.2504 -0.2587 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-3.5801 0.2723 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-4.2173 0.4392 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-4.5919 -0.3428 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-4.2230 -0.6747 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-2.2496 -1.1209 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
3.1021 -0.8447 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.9588 -1.6574 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.6269 -1.1730 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
4.3980 -1.2113 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
4.5919 -0.2838 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.9239 -0.7682 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
4.5252 -1.8119 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
3.2782 -1.9207 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
2.4164 -1.5618 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
2.5830 -1.2082 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1 2 2 0 0 0 0
2 3 1 0 0 0 0
3 5 2 0 0 0 0
6 4 2 0 0 0 0
4 1 1 0 0 0 0
5 6 1 0 0 0 0
6 7 1 0 0 0 0
7 8 1 0 0 0 0
8 9 1 0 0 0 0
5 9 1 0 0 0 0
8 10 1 0 0 0 0
10 11 1 0 0 0 0
11 12 2 0 0 0 0
12 13 1 0 0 0 0
13 14 2 0 0 0 0
14 15 1 0 0 0 0
15 16 2 0 0 0 0
16 11 1 0 0 0 0
3 17 1 0 0 0 0
14 18 1 0 0 0 0
1 19 1 0 0 0 0
9 20 2 0 0 0 0
8 21 1 0 0 0 0
22 23 1 1 0 0 0
23 24 1 1 0 0 0
25 24 1 1 0 0 0
25 26 1 0 0 0 0
26 27 1 0 0 0 0
27 22 1 0 0 0 0
27 28 1 0 0 0 0
28 29 1 0 0 0 0
22 30 1 0 0 0 0
23 31 1 0 0 0 0
24 32 1 0 0 0 0
17 25 1 0 0 0 0
33 34 1 0 0 0 0
34 35 1 1 0 0 0
36 35 1 1 0 0 0
37 36 1 1 0 0 0
37 38 1 0 0 0 0
38 33 1 0 0 0 0
36 39 1 0 0 0 0
35 40 1 0 0 0 0
34 41 1 0 0 0 0
33 42 1 0 0 0 0
37 18 1 0 0 0 0
S SKP 5
ID FL1BAAGS0003
FORMULA C27H32O15
EXACTMASS 596.174120354
AVERAGEMASS 596.5339799999999
SMILES O(C1Oc(c5)ccc(c5)CC(C4=O)(O)Oc(c42)cc(cc2OC(C3O)OC(CO)C(C3O)O)O)C(C(C(O)C(O)1)O)C
M END