Mol:COX00099
From Metabolomics.JP
(Difference between revisions)
| Line 1: | Line 1: | ||
| − | 941651 | + | 941651 |
| − | CDK 9/16/09,17:16 | + | CDK 9/16/09,17:16 |
| − | + | ||
| − | 59 62 0 0 0 0 0 0 0 0999 V2000 | + | 59 62 0 0 0 0 0 0 0 0999 V2000 |
| − | 8.1962 -1.2085 0.0000 S 0 0 0 0 0 0 0 0 0 0 0 0 | + | 8.1962 -1.2085 0.0000 S 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 4.6660 -3.2327 0.0000 S 0 0 0 0 0 0 0 0 0 0 0 0 | + | 4.6660 -3.2327 0.0000 S 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 7.6928 -0.3444 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 7.6928 -0.3444 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 8.6995 -2.0726 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 8.6995 -2.0726 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 6.3981 1.7673 0.0000 N 0 0 0 0 0 0 0 0 0 0 0 0 | + | 6.3981 1.7673 0.0000 N 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 8.1301 2.7673 0.0000 N 0 0 0 0 0 0 0 0 0 0 0 0 | + | 8.1301 2.7673 0.0000 N 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 9.0603 -0.7052 0.0000 N 0 0 0 0 0 0 0 0 0 0 0 0 | + | 9.0603 -0.7052 0.0000 N 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 7.2641 1.2673 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 7.2641 1.2673 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 6.3981 2.7673 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 6.3981 2.7673 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 8.1301 1.7673 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 8.1301 1.7673 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 7.2641 3.2673 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 7.2641 3.2673 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 5.5321 1.2673 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 5.5321 1.2673 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 5.5321 0.2673 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 5.5321 0.2673 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 8.9962 3.2673 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 8.9962 3.2673 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 4.6660 -0.2327 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 4.6660 -0.2327 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 4.6660 -1.2327 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 4.6660 -1.2327 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 5.5321 -1.7327 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 5.5321 -1.7327 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.8000 -1.7327 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.8000 -1.7327 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 5.5321 -2.7327 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 5.5321 -2.7327 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 6.4260 -1.1980 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 6.4260 -1.1980 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.8000 -2.7327 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.8000 -2.7327 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 7.3321 -1.7119 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 7.3321 -1.7119 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 6.4260 -3.2673 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 6.4260 -3.2673 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.9061 -1.1980 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.9061 -1.1980 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 7.3321 -2.7535 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 7.3321 -2.7535 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.9061 -3.2673 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.9061 -3.2673 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.0000 -1.7119 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.0000 -1.7119 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.0000 -2.7535 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.0000 -2.7535 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 9.9282 -1.2018 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 9.9282 -1.2018 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 9.0564 0.2948 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 9.0564 0.2948 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 7.6626 0.7924 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0 | + | 7.6626 0.7924 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 6.8656 0.7924 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0 | + | 6.8656 0.7924 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 5.7875 2.6597 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0 | + | 5.7875 2.6597 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 6.1860 3.3499 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0 | + | 6.1860 3.3499 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 8.7407 1.8750 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0 | + | 8.7407 1.8750 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 8.3422 1.1847 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0 | + | 8.3422 1.1847 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 6.8656 3.7423 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0 | + | 6.8656 3.7423 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 7.6626 3.7423 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0 | + | 7.6626 3.7423 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 4.9215 1.1597 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0 | + | 4.9215 1.1597 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 5.3200 1.8499 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0 | + | 5.3200 1.8499 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 6.1426 0.3750 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0 | + | 6.1426 0.3750 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 5.7441 -0.3153 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0 | + | 5.7441 -0.3153 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 9.3062 2.7304 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0 | + | 9.3062 2.7304 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 9.5331 3.5773 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0 | + | 9.5331 3.5773 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 8.6862 3.8043 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0 | + | 8.6862 3.8043 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 4.1291 0.0773 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0 | + | 4.1291 0.0773 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 6.4188 -0.5780 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0 | + | 6.4188 -0.5780 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 6.4188 -3.8873 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0 | + | 6.4188 -3.8873 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.9132 -0.5780 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.9132 -0.5780 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 7.8678 -3.0656 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0 | + | 7.8678 -3.0656 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.9132 -3.8873 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.9132 -3.8873 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.4643 -1.3998 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.4643 -1.3998 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.4643 -3.0656 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.4643 -3.0656 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 10.2361 -0.6637 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0 | + | 10.2361 -0.6637 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 10.4663 -1.5098 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0 | + | 10.4663 -1.5098 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 9.6203 -1.7400 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0 | + | 9.6203 -1.7400 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 8.4364 0.2924 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0 | + | 8.4364 0.2924 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 9.0540 0.9148 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0 | + | 9.0540 0.9148 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 9.6764 0.2972 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0 | + | 9.6764 0.2972 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0 |
1 3 2 0 0 0 0 | 1 3 2 0 0 0 0 | ||
1 4 2 0 0 0 0 | 1 4 2 0 0 0 0 | ||
| Line 124: | Line 124: | ||
30 58 1 0 0 0 0 | 30 58 1 0 0 0 0 | ||
30 59 1 0 0 0 0 | 30 59 1 0 0 0 0 | ||
| − | S SKP | + | S SKP 6 |
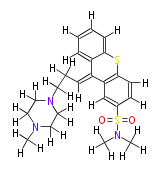
| − | NAME Thiothixene | + | NAME Thiothixene |
| + | ID COX00099 | ||
| + | FORMULA C23H29N3O2S2 | ||
| + | EXACTMASS 443.17011856700003 | ||
| + | AVERAGEMASS 443.62738 | ||
| + | SMILES [H]c(c([H])4)c([H])c(S1)c(c([H])4)C(=C([H])C([H])([H])C([H])([H])N(C([H])([H])3)C([H])([H])C([H])([H])N(C([H])([H])[H])C([H])([H])3)c(c([H])2)c1c([H])c([H])c2S(=O)(=O)N(C([H])([H])[H])C([H])([H])[H] | ||
M END | M END | ||
Latest revision as of 20:48, 21 February 2011
941651
CDK 9/16/09,17:16
59 62 0 0 0 0 0 0 0 0999 V2000
8.1962 -1.2085 0.0000 S 0 0 0 0 0 0 0 0 0 0 0 0
4.6660 -3.2327 0.0000 S 0 0 0 0 0 0 0 0 0 0 0 0
7.6928 -0.3444 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
8.6995 -2.0726 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
6.3981 1.7673 0.0000 N 0 0 0 0 0 0 0 0 0 0 0 0
8.1301 2.7673 0.0000 N 0 0 0 0 0 0 0 0 0 0 0 0
9.0603 -0.7052 0.0000 N 0 0 0 0 0 0 0 0 0 0 0 0
7.2641 1.2673 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
6.3981 2.7673 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
8.1301 1.7673 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
7.2641 3.2673 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
5.5321 1.2673 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
5.5321 0.2673 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
8.9962 3.2673 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
4.6660 -0.2327 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
4.6660 -1.2327 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
5.5321 -1.7327 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.8000 -1.7327 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
5.5321 -2.7327 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
6.4260 -1.1980 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.8000 -2.7327 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
7.3321 -1.7119 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
6.4260 -3.2673 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.9061 -1.1980 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
7.3321 -2.7535 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.9061 -3.2673 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.0000 -1.7119 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.0000 -2.7535 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
9.9282 -1.2018 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
9.0564 0.2948 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
7.6626 0.7924 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0
6.8656 0.7924 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0
5.7875 2.6597 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0
6.1860 3.3499 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0
8.7407 1.8750 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0
8.3422 1.1847 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0
6.8656 3.7423 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0
7.6626 3.7423 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0
4.9215 1.1597 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0
5.3200 1.8499 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0
6.1426 0.3750 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0
5.7441 -0.3153 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0
9.3062 2.7304 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0
9.5331 3.5773 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0
8.6862 3.8043 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0
4.1291 0.0773 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0
6.4188 -0.5780 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0
6.4188 -3.8873 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0
2.9132 -0.5780 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0
7.8678 -3.0656 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0
2.9132 -3.8873 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0
1.4643 -1.3998 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0
1.4643 -3.0656 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0
10.2361 -0.6637 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0
10.4663 -1.5098 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0
9.6203 -1.7400 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0
8.4364 0.2924 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0
9.0540 0.9148 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0
9.6764 0.2972 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0
1 3 2 0 0 0 0
1 4 2 0 0 0 0
1 7 1 0 0 0 0
1 22 1 0 0 0 0
2 19 1 0 0 0 0
2 21 1 0 0 0 0
5 8 1 0 0 0 0
5 9 1 0 0 0 0
5 12 1 0 0 0 0
6 10 1 0 0 0 0
6 11 1 0 0 0 0
6 14 1 0 0 0 0
7 29 1 0 0 0 0
7 30 1 0 0 0 0
8 10 1 0 0 0 0
8 31 1 0 0 0 0
8 32 1 0 0 0 0
9 11 1 0 0 0 0
9 33 1 0 0 0 0
9 34 1 0 0 0 0
10 35 1 0 0 0 0
10 36 1 0 0 0 0
11 37 1 0 0 0 0
11 38 1 0 0 0 0
12 13 1 0 0 0 0
12 39 1 0 0 0 0
12 40 1 0 0 0 0
13 15 1 0 0 0 0
13 41 1 0 0 0 0
13 42 1 0 0 0 0
14 43 1 0 0 0 0
14 44 1 0 0 0 0
14 45 1 0 0 0 0
15 16 2 0 0 0 0
15 46 1 0 0 0 0
16 17 1 0 0 0 0
16 18 1 0 0 0 0
17 19 2 0 0 0 0
17 20 1 0 0 0 0
18 21 1 0 0 0 0
18 24 2 0 0 0 0
19 23 1 0 0 0 0
20 22 2 0 0 0 0
20 47 1 0 0 0 0
21 26 2 0 0 0 0
22 25 1 0 0 0 0
23 25 2 0 0 0 0
23 48 1 0 0 0 0
24 27 1 0 0 0 0
24 49 1 0 0 0 0
25 50 1 0 0 0 0
26 28 1 0 0 0 0
26 51 1 0 0 0 0
27 28 2 0 0 0 0
27 52 1 0 0 0 0
28 53 1 0 0 0 0
29 54 1 0 0 0 0
29 55 1 0 0 0 0
29 56 1 0 0 0 0
30 57 1 0 0 0 0
30 58 1 0 0 0 0
30 59 1 0 0 0 0
S SKP 6
NAME Thiothixene
ID COX00099
FORMULA C23H29N3O2S2
EXACTMASS 443.17011856700003
AVERAGEMASS 443.62738
SMILES [H]c(c([H])4)c([H])c(S1)c(c([H])4)C(=C([H])C([H])([H])C([H])([H])N(C([H])([H])3)C([H])([H])C([H])([H])N(C([H])([H])[H])C([H])([H])3)c(c([H])2)c1c([H])c([H])c2S(=O)(=O)N(C([H])([H])[H])C([H])([H])[H]
M END