Mol:BMMCBZ1Sa018
From Metabolomics.JP
(Difference between revisions)
| Line 1: | Line 1: | ||
| − | + | ||
| − | + | ||
| − | Copyright: ARM project http://www.metabolome.jp/ | + | Copyright: ARM project http://www.metabolome.jp/ |
| − | 63 66 0 0 1 0 0 0 0 0999 V2000 | + | 63 66 0 0 1 0 0 0 0 0999 V2000 |
| − | 7.3713 -3.1287 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 7.3713 -3.1287 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 6.3931 -2.9208 0.0000 C 0 0 3 0 0 0 0 0 0 0 0 0 | + | 6.3931 -2.9208 0.0000 C 0 0 3 0 0 0 0 0 0 0 0 0 |
| − | 6.0841 -1.9698 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 6.0841 -1.9698 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 5.1060 -1.7619 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 5.1060 -1.7619 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 4.9344 -4.5409 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 4.9344 -4.5409 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.9563 -4.3330 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.9563 -4.3330 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.2872 -3.5898 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.2872 -3.5898 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.3090 -3.7977 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.3090 -3.7977 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.0000 -4.7488 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.0000 -4.7488 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.6691 -5.4919 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.6691 -5.4919 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.6473 -5.2840 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.6473 -5.2840 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 4.4369 -2.5050 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 4.4369 -2.5050 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 4.7970 -0.8108 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 4.7970 -0.8108 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 5.2435 -5.4919 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 5.2435 -5.4919 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 7.6803 -4.0798 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 7.6803 -4.0798 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 24.7284 -2.0988 0.0000 N 0 0 0 0 0 0 0 0 0 0 0 0 | + | 24.7284 -2.0988 0.0000 N 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 23.8624 -1.5988 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 23.8624 -1.5988 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 23.8624 -0.5988 0.0000 N 0 0 0 0 0 0 0 0 0 0 0 0 | + | 23.8624 -0.5988 0.0000 N 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 24.7284 -0.0988 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 24.7284 -0.0988 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 25.5944 -0.5988 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 25.5944 -0.5988 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 25.5944 -1.5988 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 25.5944 -1.5988 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 26.3376 0.0703 0.0000 N 0 0 0 0 0 0 0 0 0 0 0 0 | + | 26.3376 0.0703 0.0000 N 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 25.9308 0.9839 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 25.9308 0.9839 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 24.9363 0.8793 0.0000 N 0 0 0 0 0 0 0 0 0 0 0 0 | + | 24.9363 0.8793 0.0000 N 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 26.4605 -2.0988 0.0000 N 0 0 0 0 0 0 0 0 0 0 0 0 | + | 26.4605 -2.0988 0.0000 N 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 24.2672 1.6225 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0 | + | 24.2672 1.6225 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0 |
| − | 24.4751 2.6006 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0 | + | 24.4751 2.6006 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0 |
| − | 23.6091 3.1006 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0 | + | 23.6091 3.1006 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0 |
| − | 22.8659 2.4315 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0 | + | 22.8659 2.4315 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0 |
| − | 21.8878 2.6394 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 21.8878 2.6394 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 25.3886 3.0074 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 25.3886 3.0074 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 23.5045 4.0951 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 23.5045 4.0951 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 23.2727 1.5179 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 23.2727 1.5179 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 21.2187 1.8963 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 21.2187 1.8963 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 24.3136 4.6829 0.0000 P 0 0 0 0 0 0 0 0 0 0 0 0 | + | 24.3136 4.6829 0.0000 P 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 24.9013 3.8739 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 24.9013 3.8739 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 23.7258 5.4919 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 23.7258 5.4919 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 25.1226 5.2707 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 25.1226 5.2707 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 20.2405 2.1042 0.0000 P 0 0 0 0 0 0 0 0 0 0 0 0 | + | 20.2405 2.1042 0.0000 P 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 20.0326 1.1260 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 20.0326 1.1260 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 20.4484 3.0823 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 20.4484 3.0823 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 19.2624 2.3121 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 19.2624 2.3121 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 18.5932 1.5689 0.0000 P 0 0 0 0 0 0 0 0 0 0 0 0 | + | 18.5932 1.5689 0.0000 P 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 19.3364 0.8998 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 19.3364 0.8998 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 17.8501 2.2381 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 17.8501 2.2381 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 17.9241 0.8258 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 17.9241 0.8258 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 16.9459 1.0337 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 16.9459 1.0337 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 16.2768 0.2906 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 16.2768 0.2906 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 17.0200 -0.3786 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 17.0200 -0.3786 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 15.5337 0.9597 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 15.5337 0.9597 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 15.6077 -0.4526 0.0000 C 0 0 2 0 0 0 0 0 0 0 0 0 | + | 15.6077 -0.4526 0.0000 C 0 0 2 0 0 0 0 0 0 0 0 0 |
| − | 14.6295 -0.2447 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 14.6295 -0.2447 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 13.9604 -0.9878 0.0000 N 0 0 0 0 0 0 0 0 0 0 0 0 | + | 13.9604 -0.9878 0.0000 N 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 12.9823 -0.7799 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 12.9823 -0.7799 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 12.3131 -1.5230 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 12.3131 -1.5230 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 11.3350 -1.3151 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 11.3350 -1.3151 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 10.6659 -2.0583 0.0000 N 0 0 0 0 0 0 0 0 0 0 0 0 | + | 10.6659 -2.0583 0.0000 N 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 9.6877 -1.8504 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 9.6877 -1.8504 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 9.0186 -2.5935 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 9.0186 -2.5935 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 8.0404 -2.3856 0.0000 S 0 0 0 0 0 0 0 0 0 0 0 0 | + | 8.0404 -2.3856 0.0000 S 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 15.9167 -1.4036 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 15.9167 -1.4036 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 14.3205 0.7064 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 14.3205 0.7064 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 11.0260 -0.3641 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 11.0260 -0.3641 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 16 21 2 0 0 0 0 | + | 16 21 2 0 0 0 0 |
| − | 16 17 1 0 0 0 0 | + | 16 17 1 0 0 0 0 |
| − | 17 18 2 0 0 0 0 | + | 17 18 2 0 0 0 0 |
| − | 18 19 1 0 0 0 0 | + | 18 19 1 0 0 0 0 |
| − | 20 21 1 0 0 0 0 | + | 20 21 1 0 0 0 0 |
| − | 20 19 2 0 0 0 0 | + | 20 19 2 0 0 0 0 |
| − | 24 23 1 0 0 0 0 | + | 24 23 1 0 0 0 0 |
| − | 23 22 2 0 0 0 0 | + | 23 22 2 0 0 0 0 |
| − | 22 20 1 0 0 0 0 | + | 22 20 1 0 0 0 0 |
| − | 19 24 1 0 0 0 0 | + | 19 24 1 0 0 0 0 |
| − | 21 25 1 0 0 0 0 | + | 21 25 1 0 0 0 0 |
| − | 29 33 1 6 0 0 0 | + | 29 33 1 6 0 0 0 |
| − | 29 28 1 0 0 0 0 | + | 29 28 1 0 0 0 0 |
| − | 26 33 1 6 0 0 0 | + | 26 33 1 6 0 0 0 |
| − | 28 27 1 0 0 0 0 | + | 28 27 1 0 0 0 0 |
| − | 26 27 1 0 0 0 0 | + | 26 27 1 0 0 0 0 |
| − | 29 30 1 0 0 0 0 | + | 29 30 1 0 0 0 0 |
| − | 30 34 1 0 0 0 0 | + | 30 34 1 0 0 0 0 |
| − | 26 24 1 0 0 0 0 | + | 26 24 1 0 0 0 0 |
| − | 34 39 1 0 0 0 0 | + | 34 39 1 0 0 0 0 |
| − | 39 41 1 0 0 0 0 | + | 39 41 1 0 0 0 0 |
| − | 39 40 2 0 0 0 0 | + | 39 40 2 0 0 0 0 |
| − | 39 42 1 0 0 0 0 | + | 39 42 1 0 0 0 0 |
| − | 46 43 1 0 0 0 0 | + | 46 43 1 0 0 0 0 |
| − | 43 44 2 0 0 0 0 | + | 43 44 2 0 0 0 0 |
| − | 43 45 1 0 0 0 0 | + | 43 45 1 0 0 0 0 |
| − | 46 47 1 0 0 0 0 | + | 46 47 1 0 0 0 0 |
| − | 43 42 1 0 0 0 0 | + | 43 42 1 0 0 0 0 |
| − | 47 48 1 0 0 0 0 | + | 47 48 1 0 0 0 0 |
| − | 48 51 1 0 0 0 0 | + | 48 51 1 0 0 0 0 |
| − | 48 50 1 0 0 0 0 | + | 48 50 1 0 0 0 0 |
| − | 48 49 1 0 0 0 0 | + | 48 49 1 0 0 0 0 |
| − | 51 52 1 0 0 0 0 | + | 51 52 1 0 0 0 0 |
| − | 51 61 1 1 0 0 0 | + | 51 61 1 1 0 0 0 |
| − | 52 62 2 0 0 0 0 | + | 52 62 2 0 0 0 0 |
| − | 52 53 1 0 0 0 0 | + | 52 53 1 0 0 0 0 |
| − | 53 54 1 0 0 0 0 | + | 53 54 1 0 0 0 0 |
| − | 54 55 1 0 0 0 0 | + | 54 55 1 0 0 0 0 |
| − | 55 56 1 0 0 0 0 | + | 55 56 1 0 0 0 0 |
| − | 56 57 1 0 0 0 0 | + | 56 57 1 0 0 0 0 |
| − | 56 63 2 0 0 0 0 | + | 56 63 2 0 0 0 0 |
| − | 57 58 1 0 0 0 0 | + | 57 58 1 0 0 0 0 |
| − | 58 59 1 0 0 0 0 | + | 58 59 1 0 0 0 0 |
| − | 59 60 1 0 0 0 0 | + | 59 60 1 0 0 0 0 |
| − | 27 31 1 1 0 0 0 | + | 27 31 1 1 0 0 0 |
| − | 28 32 1 1 0 0 0 | + | 28 32 1 1 0 0 0 |
| − | 38 35 1 0 0 0 0 | + | 38 35 1 0 0 0 0 |
| − | 35 36 1 0 0 0 0 | + | 35 36 1 0 0 0 0 |
| − | 35 37 2 0 0 0 0 | + | 35 37 2 0 0 0 0 |
| − | 32 35 1 0 0 0 0 | + | 32 35 1 0 0 0 0 |
| − | 60 1 1 0 0 0 0 | + | 60 1 1 0 0 0 0 |
| − | 1 2 1 0 0 0 0 | + | 1 2 1 0 0 0 0 |
| − | 1 15 2 0 0 0 0 | + | 1 15 2 0 0 0 0 |
| − | 2 3 1 4 0 0 0 | + | 2 3 1 4 0 0 0 |
| − | 2 5 1 0 0 0 0 | + | 2 5 1 0 0 0 0 |
| − | 5 6 1 0 0 0 0 | + | 5 6 1 0 0 0 0 |
| − | 6 7 2 0 0 0 0 | + | 6 7 2 0 0 0 0 |
| − | 7 8 1 0 0 0 0 | + | 7 8 1 0 0 0 0 |
| − | 8 9 2 0 0 0 0 | + | 8 9 2 0 0 0 0 |
| − | 9 10 1 0 0 0 0 | + | 9 10 1 0 0 0 0 |
| − | 10 11 2 0 0 0 0 | + | 10 11 2 0 0 0 0 |
| − | 11 6 1 0 0 0 0 | + | 11 6 1 0 0 0 0 |
| − | 5 14 2 0 0 0 0 | + | 5 14 2 0 0 0 0 |
| − | 3 4 1 0 0 0 0 | + | 3 4 1 0 0 0 0 |
| − | 4 13 1 0 0 0 0 | + | 4 13 1 0 0 0 0 |
| − | 4 12 2 0 0 0 0 | + | 4 12 2 0 0 0 0 |
| − | S SKP 7 | + | S SKP 7 |
| − | ID BMMCBZ1Sa018 | + | ID BMMCBZ1Sa018 |
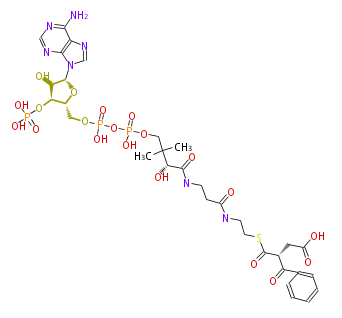
| − | NAME Benzoyl-succinyl-CoA | + | NAME Benzoyl-succinyl-CoA |
| − | FORMULA C32H44N7O20P3S | + | FORMULA C32H44N7O20P3S |
| − | EXACTMASS 971.1574 | + | EXACTMASS 971.1574 |
| − | AVERAGEMASS 971.7142 | + | AVERAGEMASS 971.7142 |
| − | SMILES C(OP(OP(O)(=O)OC[C@@H](O2)[C@@H](OP(O)(O)=O)[C@H]([C@@H]2n(c34)cnc3c(ncn4)N)O)(O)=O)C(C)(C)[C@H](C(=O)NCCC(=O)NCCSC(=O)C(CC(O)=O)C(c(c1)cccc1)=O)O | + | SMILES C(OP(OP(O)(=O)OC[C@@H](O2)[C@@H](OP(O)(O)=O)[C@H]([C@@H]2n(c34)cnc3c(ncn4)N)O)(O)=O)C(C)(C)[C@H](C(=O)NCCC(=O)NCCSC(=O)C(CC(O)=O)C(c(c1)cccc1)=O)O |
| − | KEGG http://www.genome.jp/dbget-bin/www_bget?compound+C09820 | + | KEGG http://www.genome.jp/dbget-bin/www_bget?compound+C09820 |
M END | M END | ||
| − | |||
Revision as of 09:00, 14 March 2009
Copyright: ARM project http://www.metabolome.jp/
63 66 0 0 1 0 0 0 0 0999 V2000
7.3713 -3.1287 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
6.3931 -2.9208 0.0000 C 0 0 3 0 0 0 0 0 0 0 0 0
6.0841 -1.9698 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
5.1060 -1.7619 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
4.9344 -4.5409 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.9563 -4.3330 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.2872 -3.5898 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.3090 -3.7977 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.0000 -4.7488 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.6691 -5.4919 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.6473 -5.2840 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
4.4369 -2.5050 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
4.7970 -0.8108 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
5.2435 -5.4919 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
7.6803 -4.0798 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
24.7284 -2.0988 0.0000 N 0 0 0 0 0 0 0 0 0 0 0 0
23.8624 -1.5988 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
23.8624 -0.5988 0.0000 N 0 0 0 0 0 0 0 0 0 0 0 0
24.7284 -0.0988 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
25.5944 -0.5988 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
25.5944 -1.5988 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
26.3376 0.0703 0.0000 N 0 0 0 0 0 0 0 0 0 0 0 0
25.9308 0.9839 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
24.9363 0.8793 0.0000 N 0 0 0 0 0 0 0 0 0 0 0 0
26.4605 -2.0988 0.0000 N 0 0 0 0 0 0 0 0 0 0 0 0
24.2672 1.6225 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0
24.4751 2.6006 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0
23.6091 3.1006 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0
22.8659 2.4315 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0
21.8878 2.6394 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
25.3886 3.0074 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
23.5045 4.0951 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
23.2727 1.5179 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
21.2187 1.8963 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
24.3136 4.6829 0.0000 P 0 0 0 0 0 0 0 0 0 0 0 0
24.9013 3.8739 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
23.7258 5.4919 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
25.1226 5.2707 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
20.2405 2.1042 0.0000 P 0 0 0 0 0 0 0 0 0 0 0 0
20.0326 1.1260 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
20.4484 3.0823 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
19.2624 2.3121 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
18.5932 1.5689 0.0000 P 0 0 0 0 0 0 0 0 0 0 0 0
19.3364 0.8998 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
17.8501 2.2381 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
17.9241 0.8258 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
16.9459 1.0337 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
16.2768 0.2906 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
17.0200 -0.3786 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
15.5337 0.9597 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
15.6077 -0.4526 0.0000 C 0 0 2 0 0 0 0 0 0 0 0 0
14.6295 -0.2447 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
13.9604 -0.9878 0.0000 N 0 0 0 0 0 0 0 0 0 0 0 0
12.9823 -0.7799 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
12.3131 -1.5230 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
11.3350 -1.3151 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
10.6659 -2.0583 0.0000 N 0 0 0 0 0 0 0 0 0 0 0 0
9.6877 -1.8504 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
9.0186 -2.5935 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
8.0404 -2.3856 0.0000 S 0 0 0 0 0 0 0 0 0 0 0 0
15.9167 -1.4036 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
14.3205 0.7064 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
11.0260 -0.3641 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
16 21 2 0 0 0 0
16 17 1 0 0 0 0
17 18 2 0 0 0 0
18 19 1 0 0 0 0
20 21 1 0 0 0 0
20 19 2 0 0 0 0
24 23 1 0 0 0 0
23 22 2 0 0 0 0
22 20 1 0 0 0 0
19 24 1 0 0 0 0
21 25 1 0 0 0 0
29 33 1 6 0 0 0
29 28 1 0 0 0 0
26 33 1 6 0 0 0
28 27 1 0 0 0 0
26 27 1 0 0 0 0
29 30 1 0 0 0 0
30 34 1 0 0 0 0
26 24 1 0 0 0 0
34 39 1 0 0 0 0
39 41 1 0 0 0 0
39 40 2 0 0 0 0
39 42 1 0 0 0 0
46 43 1 0 0 0 0
43 44 2 0 0 0 0
43 45 1 0 0 0 0
46 47 1 0 0 0 0
43 42 1 0 0 0 0
47 48 1 0 0 0 0
48 51 1 0 0 0 0
48 50 1 0 0 0 0
48 49 1 0 0 0 0
51 52 1 0 0 0 0
51 61 1 1 0 0 0
52 62 2 0 0 0 0
52 53 1 0 0 0 0
53 54 1 0 0 0 0
54 55 1 0 0 0 0
55 56 1 0 0 0 0
56 57 1 0 0 0 0
56 63 2 0 0 0 0
57 58 1 0 0 0 0
58 59 1 0 0 0 0
59 60 1 0 0 0 0
27 31 1 1 0 0 0
28 32 1 1 0 0 0
38 35 1 0 0 0 0
35 36 1 0 0 0 0
35 37 2 0 0 0 0
32 35 1 0 0 0 0
60 1 1 0 0 0 0
1 2 1 0 0 0 0
1 15 2 0 0 0 0
2 3 1 4 0 0 0
2 5 1 0 0 0 0
5 6 1 0 0 0 0
6 7 2 0 0 0 0
7 8 1 0 0 0 0
8 9 2 0 0 0 0
9 10 1 0 0 0 0
10 11 2 0 0 0 0
11 6 1 0 0 0 0
5 14 2 0 0 0 0
3 4 1 0 0 0 0
4 13 1 0 0 0 0
4 12 2 0 0 0 0
S SKP 7
ID BMMCBZ1Sa018
NAME Benzoyl-succinyl-CoA
FORMULA C32H44N7O20P3S
EXACTMASS 971.1574
AVERAGEMASS 971.7142
SMILES C(OP(OP(O)(=O)OC[C@@H](O2)[C@@H](OP(O)(O)=O)[C@H]([C@@H]2n(c34)cnc3c(ncn4)N)O)(O)=O)C(C)(C)[C@H](C(=O)NCCC(=O)NCCSC(=O)C(CC(O)=O)C(c(c1)cccc1)=O)O
KEGG http://www.genome.jp/dbget-bin/www_bget?compound+C09820
M END