Mol:BMFYS9ESa003
From Metabolomics.JP
(Difference between revisions)
| Line 1: | Line 1: | ||
| − | + | ||
| − | + | ||
| − | Copyright: ARM project http://www.metabolome.jp/ | + | Copyright: ARM project http://www.metabolome.jp/ |
| − | 58 60 0 0 1 0 0 0 0 0999 V2000 | + | 58 60 0 0 1 0 0 0 0 0999 V2000 |
| − | 8.5891 -3.2399 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 8.5891 -3.2399 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 7.6110 -3.0320 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 7.6110 -3.0320 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 6.9418 -3.7751 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 6.9418 -3.7751 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 5.9637 -3.5672 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 5.9637 -3.5672 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 5.2946 -4.3103 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 5.2946 -4.3103 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 4.3164 -4.1024 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 4.3164 -4.1024 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.6473 -4.8456 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.6473 -4.8456 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.6691 -4.6377 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.6691 -4.6377 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.0000 -5.3808 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.0000 -5.3808 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 8.8981 -4.1909 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 8.8981 -4.1909 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 25.9462 -2.2099 0.0000 N 0 0 0 0 0 0 0 0 0 0 0 0 | + | 25.9462 -2.2099 0.0000 N 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 25.0802 -1.7099 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 25.0802 -1.7099 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 25.0802 -0.7099 0.0000 N 0 0 0 0 0 0 0 0 0 0 0 0 | + | 25.0802 -0.7099 0.0000 N 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 25.9462 -0.2099 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 25.9462 -0.2099 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 26.8123 -0.7099 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 26.8123 -0.7099 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 26.8123 -1.7099 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 26.8123 -1.7099 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 27.5554 -0.0408 0.0000 N 0 0 0 0 0 0 0 0 0 0 0 0 | + | 27.5554 -0.0408 0.0000 N 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 27.1487 0.8727 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 27.1487 0.8727 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 26.1541 0.7682 0.0000 N 0 0 0 0 0 0 0 0 0 0 0 0 | + | 26.1541 0.7682 0.0000 N 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 27.6783 -2.2099 0.0000 N 0 0 0 0 0 0 0 0 0 0 0 0 | + | 27.6783 -2.2099 0.0000 N 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 25.4850 1.5113 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0 | + | 25.4850 1.5113 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0 |
| − | 25.6929 2.4895 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0 | + | 25.6929 2.4895 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0 |
| − | 24.8269 2.9895 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0 | + | 24.8269 2.9895 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0 |
| − | 24.0837 2.3204 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0 | + | 24.0837 2.3204 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0 |
| − | 23.1056 2.5283 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 23.1056 2.5283 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 26.6065 2.8962 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 26.6065 2.8962 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 24.7224 3.9840 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 24.7224 3.9840 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 24.4905 1.4068 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 24.4905 1.4068 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 22.4365 1.7851 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 22.4365 1.7851 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 25.5314 4.5718 0.0000 P 0 0 0 0 0 0 0 0 0 0 0 0 | + | 25.5314 4.5718 0.0000 P 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 26.1192 3.7628 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 26.1192 3.7628 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 24.9436 5.3808 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 24.9436 5.3808 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 26.3404 5.1596 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 26.3404 5.1596 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 21.4583 1.9930 0.0000 P 0 0 0 0 0 0 0 0 0 0 0 0 | + | 21.4583 1.9930 0.0000 P 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 21.2504 1.0149 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 21.2504 1.0149 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 21.6662 2.9712 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 21.6662 2.9712 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 20.4802 2.2010 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 20.4802 2.2010 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 19.8110 1.4578 0.0000 P 0 0 0 0 0 0 0 0 0 0 0 0 | + | 19.8110 1.4578 0.0000 P 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 20.5542 0.7887 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 20.5542 0.7887 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 19.0679 2.1269 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 19.0679 2.1269 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 19.1419 0.7147 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 19.1419 0.7147 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 18.1638 0.9226 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 18.1638 0.9226 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 17.4946 0.1794 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 17.4946 0.1794 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 18.2378 -0.4897 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 18.2378 -0.4897 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 16.7515 0.8486 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 16.7515 0.8486 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 16.8255 -0.5637 0.0000 C 0 0 2 0 0 0 0 0 0 0 0 0 | + | 16.8255 -0.5637 0.0000 C 0 0 2 0 0 0 0 0 0 0 0 0 |
| − | 15.8474 -0.3558 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 15.8474 -0.3558 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 15.1782 -1.0990 0.0000 N 0 0 0 0 0 0 0 0 0 0 0 0 | + | 15.1782 -1.0990 0.0000 N 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 14.2001 -0.8910 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 14.2001 -0.8910 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 13.5309 -1.6342 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 13.5309 -1.6342 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 12.5528 -1.4263 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 12.5528 -1.4263 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 11.8837 -2.1694 0.0000 N 0 0 0 0 0 0 0 0 0 0 0 0 | + | 11.8837 -2.1694 0.0000 N 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 10.9055 -1.9615 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 10.9055 -1.9615 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 10.2364 -2.7046 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 10.2364 -2.7046 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 9.2582 -2.4967 0.0000 S 0 0 0 0 0 0 0 0 0 0 0 0 | + | 9.2582 -2.4967 0.0000 S 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 17.1345 -1.5148 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 17.1345 -1.5148 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 15.5383 0.5953 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 15.5383 0.5953 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 12.2438 -0.4752 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 12.2438 -0.4752 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 11 16 2 0 0 0 0 | + | 11 16 2 0 0 0 0 |
| − | 11 12 1 0 0 0 0 | + | 11 12 1 0 0 0 0 |
| − | 12 13 2 0 0 0 0 | + | 12 13 2 0 0 0 0 |
| − | 13 14 1 0 0 0 0 | + | 13 14 1 0 0 0 0 |
| − | 15 16 1 0 0 0 0 | + | 15 16 1 0 0 0 0 |
| − | 15 14 2 0 0 0 0 | + | 15 14 2 0 0 0 0 |
| − | 19 18 1 0 0 0 0 | + | 19 18 1 0 0 0 0 |
| − | 18 17 2 0 0 0 0 | + | 18 17 2 0 0 0 0 |
| − | 17 15 1 0 0 0 0 | + | 17 15 1 0 0 0 0 |
| − | 14 19 1 0 0 0 0 | + | 14 19 1 0 0 0 0 |
| − | 16 20 1 0 0 0 0 | + | 16 20 1 0 0 0 0 |
| − | 24 28 1 6 0 0 0 | + | 24 28 1 6 0 0 0 |
| − | 23 24 1 0 0 0 0 | + | 23 24 1 0 0 0 0 |
| − | 21 28 1 6 0 0 0 | + | 21 28 1 6 0 0 0 |
| − | 23 22 1 0 0 0 0 | + | 23 22 1 0 0 0 0 |
| − | 21 22 1 0 0 0 0 | + | 21 22 1 0 0 0 0 |
| − | 24 25 1 0 0 0 0 | + | 24 25 1 0 0 0 0 |
| − | 25 29 1 0 0 0 0 | + | 25 29 1 0 0 0 0 |
| − | 21 19 1 0 0 0 0 | + | 21 19 1 0 0 0 0 |
| − | 29 34 1 0 0 0 0 | + | 29 34 1 0 0 0 0 |
| − | 34 36 1 0 0 0 0 | + | 34 36 1 0 0 0 0 |
| − | 34 35 2 0 0 0 0 | + | 34 35 2 0 0 0 0 |
| − | 34 37 1 0 0 0 0 | + | 34 37 1 0 0 0 0 |
| − | 41 38 1 0 0 0 0 | + | 41 38 1 0 0 0 0 |
| − | 38 39 2 0 0 0 0 | + | 38 39 2 0 0 0 0 |
| − | 38 40 1 0 0 0 0 | + | 38 40 1 0 0 0 0 |
| − | 41 42 1 0 0 0 0 | + | 41 42 1 0 0 0 0 |
| − | 38 37 1 0 0 0 0 | + | 38 37 1 0 0 0 0 |
| − | 42 43 1 0 0 0 0 | + | 42 43 1 0 0 0 0 |
| − | 43 46 1 0 0 0 0 | + | 43 46 1 0 0 0 0 |
| − | 43 44 1 0 0 0 0 | + | 43 44 1 0 0 0 0 |
| − | 43 45 1 0 0 0 0 | + | 43 45 1 0 0 0 0 |
| − | 46 47 1 0 0 0 0 | + | 46 47 1 0 0 0 0 |
| − | 46 56 1 1 0 0 0 | + | 46 56 1 1 0 0 0 |
| − | 47 57 2 0 0 0 0 | + | 47 57 2 0 0 0 0 |
| − | 47 48 1 0 0 0 0 | + | 47 48 1 0 0 0 0 |
| − | 48 49 1 0 0 0 0 | + | 48 49 1 0 0 0 0 |
| − | 49 50 1 0 0 0 0 | + | 49 50 1 0 0 0 0 |
| − | 50 51 1 0 0 0 0 | + | 50 51 1 0 0 0 0 |
| − | 51 52 1 0 0 0 0 | + | 51 52 1 0 0 0 0 |
| − | 51 58 2 0 0 0 0 | + | 51 58 2 0 0 0 0 |
| − | 52 53 1 0 0 0 0 | + | 52 53 1 0 0 0 0 |
| − | 53 54 1 0 0 0 0 | + | 53 54 1 0 0 0 0 |
| − | 54 55 1 0 0 0 0 | + | 54 55 1 0 0 0 0 |
| − | 22 26 1 1 0 0 0 | + | 22 26 1 1 0 0 0 |
| − | 23 27 1 1 0 0 0 | + | 23 27 1 1 0 0 0 |
| − | 31 30 1 0 0 0 0 | + | 31 30 1 0 0 0 0 |
| − | 30 32 1 0 0 0 0 | + | 30 32 1 0 0 0 0 |
| − | 30 33 2 0 0 0 0 | + | 30 33 2 0 0 0 0 |
| − | 27 30 1 0 0 0 0 | + | 27 30 1 0 0 0 0 |
| − | 55 1 1 0 0 0 0 | + | 55 1 1 0 0 0 0 |
| − | 1 2 1 0 0 0 0 | + | 1 2 1 0 0 0 0 |
| − | 2 3 1 0 0 0 0 | + | 2 3 1 0 0 0 0 |
| − | 3 4 1 0 0 0 0 | + | 3 4 1 0 0 0 0 |
| − | 4 5 1 0 0 0 0 | + | 4 5 1 0 0 0 0 |
| − | 5 6 1 0 0 0 0 | + | 5 6 1 0 0 0 0 |
| − | 6 7 1 0 0 0 0 | + | 6 7 1 0 0 0 0 |
| − | 7 8 1 0 0 0 0 | + | 7 8 1 0 0 0 0 |
| − | 1 10 2 0 0 0 0 | + | 1 10 2 0 0 0 0 |
| − | 8 9 1 0 0 0 0 | + | 8 9 1 0 0 0 0 |
| − | S SKP 7 | + | S SKP 7 |
| − | ID BMFYS9ESa003 | + | ID BMFYS9ESa003 |
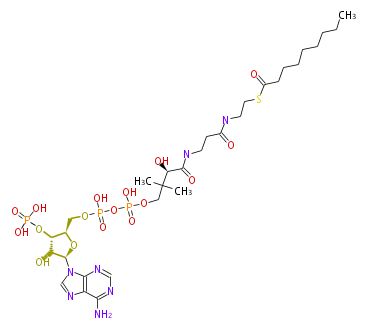
| − | NAME Nonanoyl-CoA | + | NAME Nonanoyl-CoA |
| − | FORMULA C30H52N7O17P3S | + | FORMULA C30H52N7O17P3S |
| − | EXACTMASS 907.2353 | + | EXACTMASS 907.2353 |
| − | AVERAGEMASS 907.7581 | + | AVERAGEMASS 907.7581 |
| − | SMILES n(c(N)1)cnc(n([C@H](O3)[C@@H]([C@@H]([C@H]3COP(O)(=O)OP(O)(=O)OCC(C)(C)[C@@H](O)C(=O)NCCC(NCCSC(=O)CCCCCCCC)=O)OP(O)(O)=O)O)2)c(nc2)1 | + | SMILES n(c(N)1)cnc(n([C@H](O3)[C@@H]([C@@H]([C@H]3COP(O)(=O)OP(O)(=O)OCC(C)(C)[C@@H](O)C(=O)NCCC(NCCSC(=O)CCCCCCCC)=O)OP(O)(O)=O)O)2)c(nc2)1 |
| − | KEGG http://www.genome.jp/dbget-bin/www_bget?compound+C01942 | + | KEGG http://www.genome.jp/dbget-bin/www_bget?compound+C01942 |
M END | M END | ||
| − | |||
Latest revision as of 09:00, 14 March 2009
Copyright: ARM project http://www.metabolome.jp/
58 60 0 0 1 0 0 0 0 0999 V2000
8.5891 -3.2399 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
7.6110 -3.0320 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
6.9418 -3.7751 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
5.9637 -3.5672 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
5.2946 -4.3103 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
4.3164 -4.1024 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.6473 -4.8456 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.6691 -4.6377 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.0000 -5.3808 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
8.8981 -4.1909 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
25.9462 -2.2099 0.0000 N 0 0 0 0 0 0 0 0 0 0 0 0
25.0802 -1.7099 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
25.0802 -0.7099 0.0000 N 0 0 0 0 0 0 0 0 0 0 0 0
25.9462 -0.2099 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
26.8123 -0.7099 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
26.8123 -1.7099 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
27.5554 -0.0408 0.0000 N 0 0 0 0 0 0 0 0 0 0 0 0
27.1487 0.8727 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
26.1541 0.7682 0.0000 N 0 0 0 0 0 0 0 0 0 0 0 0
27.6783 -2.2099 0.0000 N 0 0 0 0 0 0 0 0 0 0 0 0
25.4850 1.5113 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0
25.6929 2.4895 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0
24.8269 2.9895 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0
24.0837 2.3204 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0
23.1056 2.5283 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
26.6065 2.8962 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
24.7224 3.9840 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
24.4905 1.4068 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
22.4365 1.7851 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
25.5314 4.5718 0.0000 P 0 0 0 0 0 0 0 0 0 0 0 0
26.1192 3.7628 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
24.9436 5.3808 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
26.3404 5.1596 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
21.4583 1.9930 0.0000 P 0 0 0 0 0 0 0 0 0 0 0 0
21.2504 1.0149 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
21.6662 2.9712 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
20.4802 2.2010 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
19.8110 1.4578 0.0000 P 0 0 0 0 0 0 0 0 0 0 0 0
20.5542 0.7887 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
19.0679 2.1269 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
19.1419 0.7147 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
18.1638 0.9226 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
17.4946 0.1794 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
18.2378 -0.4897 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
16.7515 0.8486 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
16.8255 -0.5637 0.0000 C 0 0 2 0 0 0 0 0 0 0 0 0
15.8474 -0.3558 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
15.1782 -1.0990 0.0000 N 0 0 0 0 0 0 0 0 0 0 0 0
14.2001 -0.8910 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
13.5309 -1.6342 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
12.5528 -1.4263 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
11.8837 -2.1694 0.0000 N 0 0 0 0 0 0 0 0 0 0 0 0
10.9055 -1.9615 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
10.2364 -2.7046 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
9.2582 -2.4967 0.0000 S 0 0 0 0 0 0 0 0 0 0 0 0
17.1345 -1.5148 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
15.5383 0.5953 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
12.2438 -0.4752 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
11 16 2 0 0 0 0
11 12 1 0 0 0 0
12 13 2 0 0 0 0
13 14 1 0 0 0 0
15 16 1 0 0 0 0
15 14 2 0 0 0 0
19 18 1 0 0 0 0
18 17 2 0 0 0 0
17 15 1 0 0 0 0
14 19 1 0 0 0 0
16 20 1 0 0 0 0
24 28 1 6 0 0 0
23 24 1 0 0 0 0
21 28 1 6 0 0 0
23 22 1 0 0 0 0
21 22 1 0 0 0 0
24 25 1 0 0 0 0
25 29 1 0 0 0 0
21 19 1 0 0 0 0
29 34 1 0 0 0 0
34 36 1 0 0 0 0
34 35 2 0 0 0 0
34 37 1 0 0 0 0
41 38 1 0 0 0 0
38 39 2 0 0 0 0
38 40 1 0 0 0 0
41 42 1 0 0 0 0
38 37 1 0 0 0 0
42 43 1 0 0 0 0
43 46 1 0 0 0 0
43 44 1 0 0 0 0
43 45 1 0 0 0 0
46 47 1 0 0 0 0
46 56 1 1 0 0 0
47 57 2 0 0 0 0
47 48 1 0 0 0 0
48 49 1 0 0 0 0
49 50 1 0 0 0 0
50 51 1 0 0 0 0
51 52 1 0 0 0 0
51 58 2 0 0 0 0
52 53 1 0 0 0 0
53 54 1 0 0 0 0
54 55 1 0 0 0 0
22 26 1 1 0 0 0
23 27 1 1 0 0 0
31 30 1 0 0 0 0
30 32 1 0 0 0 0
30 33 2 0 0 0 0
27 30 1 0 0 0 0
55 1 1 0 0 0 0
1 2 1 0 0 0 0
2 3 1 0 0 0 0
3 4 1 0 0 0 0
4 5 1 0 0 0 0
5 6 1 0 0 0 0
6 7 1 0 0 0 0
7 8 1 0 0 0 0
1 10 2 0 0 0 0
8 9 1 0 0 0 0
S SKP 7
ID BMFYS9ESa003
NAME Nonanoyl-CoA
FORMULA C30H52N7O17P3S
EXACTMASS 907.2353
AVERAGEMASS 907.7581
SMILES n(c(N)1)cnc(n([C@H](O3)[C@@H]([C@@H]([C@H]3COP(O)(=O)OP(O)(=O)OCC(C)(C)[C@@H](O)C(=O)NCCC(NCCSC(=O)CCCCCCCC)=O)OP(O)(O)=O)O)2)c(nc2)1
KEGG http://www.genome.jp/dbget-bin/www_bget?compound+C01942
M END