Mol:BMFYS8ESa001
From Metabolomics.JP
(Difference between revisions)
| Line 1: | Line 1: | ||
| − | + | ||
| − | + | ||
| − | Copyright: ARM project http://www.metabolome.jp/ | + | Copyright: ARM project http://www.metabolome.jp/ |
| − | 58 60 0 0 1 0 0 0 0 0999 V2000 | + | 58 60 0 0 1 0 0 0 0 0999 V2000 |
| − | 7.9200 -3.5075 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 7.9200 -3.5075 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 6.9418 -3.2996 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 6.9418 -3.2996 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 6.2727 -4.0427 0.0000 C 0 0 2 0 0 0 0 0 0 0 0 0 | + | 6.2727 -4.0427 0.0000 C 0 0 2 0 0 0 0 0 0 0 0 0 |
| − | 5.2946 -3.8348 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 5.2946 -3.8348 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 4.6254 -4.5780 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 4.6254 -4.5780 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.6473 -4.3701 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.6473 -4.3701 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.9781 -5.1132 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.9781 -5.1132 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.0000 -4.9053 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.0000 -4.9053 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 8.2290 -4.4586 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 8.2290 -4.4586 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 6.5817 -4.9938 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 6.5817 -4.9938 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 25.2771 -2.4776 0.0000 N 0 0 0 0 0 0 0 0 0 0 0 0 | + | 25.2771 -2.4776 0.0000 N 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 24.4111 -1.9776 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 24.4111 -1.9776 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 24.4111 -0.9776 0.0000 N 0 0 0 0 0 0 0 0 0 0 0 0 | + | 24.4111 -0.9776 0.0000 N 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 25.2771 -0.4776 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 25.2771 -0.4776 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 26.1431 -0.9776 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 26.1431 -0.9776 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 26.1431 -1.9776 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 26.1431 -1.9776 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 26.8863 -0.3084 0.0000 N 0 0 0 0 0 0 0 0 0 0 0 0 | + | 26.8863 -0.3084 0.0000 N 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 26.4795 0.6051 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 26.4795 0.6051 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 25.4850 0.5006 0.0000 N 0 0 0 0 0 0 0 0 0 0 0 0 | + | 25.4850 0.5006 0.0000 N 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 27.0091 -2.4776 0.0000 N 0 0 0 0 0 0 0 0 0 0 0 0 | + | 27.0091 -2.4776 0.0000 N 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 24.8159 1.2437 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0 | + | 24.8159 1.2437 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0 |
| − | 25.0238 2.2219 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0 | + | 25.0238 2.2219 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0 |
| − | 24.1578 2.7219 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0 | + | 24.1578 2.7219 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0 |
| − | 23.4146 2.0527 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0 | + | 23.4146 2.0527 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0 |
| − | 22.4365 2.2607 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 22.4365 2.2607 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 25.9373 2.6286 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 25.9373 2.6286 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 24.0532 3.7164 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 24.0532 3.7164 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 23.8214 1.1392 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 23.8214 1.1392 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 21.7673 1.5175 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 21.7673 1.5175 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 24.8622 4.3042 0.0000 P 0 0 0 0 0 0 0 0 0 0 0 0 | + | 24.8622 4.3042 0.0000 P 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 25.4500 3.4952 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 25.4500 3.4952 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 24.2745 5.1132 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 24.2745 5.1132 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 25.6713 4.8920 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 25.6713 4.8920 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 20.7892 1.7254 0.0000 P 0 0 0 0 0 0 0 0 0 0 0 0 | + | 20.7892 1.7254 0.0000 P 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 20.5813 0.7473 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 20.5813 0.7473 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 20.9971 2.7036 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 20.9971 2.7036 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 19.8110 1.9333 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 19.8110 1.9333 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 19.1419 1.1902 0.0000 P 0 0 0 0 0 0 0 0 0 0 0 0 | + | 19.1419 1.1902 0.0000 P 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 19.8851 0.5211 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 19.8851 0.5211 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 18.3988 1.8593 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 18.3988 1.8593 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 18.4728 0.4470 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 18.4728 0.4470 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 17.4946 0.6550 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 17.4946 0.6550 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 16.8255 -0.0882 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 16.8255 -0.0882 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 17.5686 -0.7573 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 17.5686 -0.7573 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 16.0824 0.5809 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 16.0824 0.5809 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 16.1564 -0.8313 0.0000 C 0 0 2 0 0 0 0 0 0 0 0 0 | + | 16.1564 -0.8313 0.0000 C 0 0 2 0 0 0 0 0 0 0 0 0 |
| − | 15.1782 -0.6234 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 15.1782 -0.6234 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 14.5091 -1.3666 0.0000 N 0 0 0 0 0 0 0 0 0 0 0 0 | + | 14.5091 -1.3666 0.0000 N 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 13.5309 -1.1587 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 13.5309 -1.1587 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 12.8618 -1.9018 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 12.8618 -1.9018 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 11.8837 -1.6939 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 11.8837 -1.6939 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 11.2145 -2.4370 0.0000 N 0 0 0 0 0 0 0 0 0 0 0 0 | + | 11.2145 -2.4370 0.0000 N 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 10.2364 -2.2291 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 10.2364 -2.2291 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 9.5673 -2.9723 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 9.5673 -2.9723 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 8.5891 -2.7644 0.0000 S 0 0 0 0 0 0 0 0 0 0 0 0 | + | 8.5891 -2.7644 0.0000 S 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 16.4654 -1.7824 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 16.4654 -1.7824 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 14.8692 0.3276 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 14.8692 0.3276 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 11.5747 -0.7428 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 11.5747 -0.7428 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 11 16 2 0 0 0 0 | + | 11 16 2 0 0 0 0 |
| − | 11 12 1 0 0 0 0 | + | 11 12 1 0 0 0 0 |
| − | 12 13 2 0 0 0 0 | + | 12 13 2 0 0 0 0 |
| − | 13 14 1 0 0 0 0 | + | 13 14 1 0 0 0 0 |
| − | 15 16 1 0 0 0 0 | + | 15 16 1 0 0 0 0 |
| − | 15 14 2 0 0 0 0 | + | 15 14 2 0 0 0 0 |
| − | 19 18 1 0 0 0 0 | + | 19 18 1 0 0 0 0 |
| − | 18 17 2 0 0 0 0 | + | 18 17 2 0 0 0 0 |
| − | 17 15 1 0 0 0 0 | + | 17 15 1 0 0 0 0 |
| − | 14 19 1 0 0 0 0 | + | 14 19 1 0 0 0 0 |
| − | 16 20 1 0 0 0 0 | + | 16 20 1 0 0 0 0 |
| − | 24 28 1 6 0 0 0 | + | 24 28 1 6 0 0 0 |
| − | 23 24 1 0 0 0 0 | + | 23 24 1 0 0 0 0 |
| − | 21 28 1 6 0 0 0 | + | 21 28 1 6 0 0 0 |
| − | 23 22 1 0 0 0 0 | + | 23 22 1 0 0 0 0 |
| − | 21 22 1 0 0 0 0 | + | 21 22 1 0 0 0 0 |
| − | 24 25 1 0 0 0 0 | + | 24 25 1 0 0 0 0 |
| − | 25 29 1 0 0 0 0 | + | 25 29 1 0 0 0 0 |
| − | 21 19 1 0 0 0 0 | + | 21 19 1 0 0 0 0 |
| − | 29 34 1 0 0 0 0 | + | 29 34 1 0 0 0 0 |
| − | 34 36 1 0 0 0 0 | + | 34 36 1 0 0 0 0 |
| − | 34 35 2 0 0 0 0 | + | 34 35 2 0 0 0 0 |
| − | 34 37 1 0 0 0 0 | + | 34 37 1 0 0 0 0 |
| − | 41 38 1 0 0 0 0 | + | 41 38 1 0 0 0 0 |
| − | 38 40 2 0 0 0 0 | + | 38 40 2 0 0 0 0 |
| − | 38 39 1 0 0 0 0 | + | 38 39 1 0 0 0 0 |
| − | 41 42 1 0 0 0 0 | + | 41 42 1 0 0 0 0 |
| − | 38 37 1 0 0 0 0 | + | 38 37 1 0 0 0 0 |
| − | 42 43 1 0 0 0 0 | + | 42 43 1 0 0 0 0 |
| − | 43 46 1 0 0 0 0 | + | 43 46 1 0 0 0 0 |
| − | 43 45 1 0 0 0 0 | + | 43 45 1 0 0 0 0 |
| − | 43 44 1 0 0 0 0 | + | 43 44 1 0 0 0 0 |
| − | 46 47 1 0 0 0 0 | + | 46 47 1 0 0 0 0 |
| − | 46 56 1 1 0 0 0 | + | 46 56 1 1 0 0 0 |
| − | 47 57 2 0 0 0 0 | + | 47 57 2 0 0 0 0 |
| − | 47 48 1 0 0 0 0 | + | 47 48 1 0 0 0 0 |
| − | 48 49 1 0 0 0 0 | + | 48 49 1 0 0 0 0 |
| − | 49 50 1 0 0 0 0 | + | 49 50 1 0 0 0 0 |
| − | 50 51 1 0 0 0 0 | + | 50 51 1 0 0 0 0 |
| − | 51 52 1 0 0 0 0 | + | 51 52 1 0 0 0 0 |
| − | 51 58 2 0 0 0 0 | + | 51 58 2 0 0 0 0 |
| − | 52 53 1 0 0 0 0 | + | 52 53 1 0 0 0 0 |
| − | 53 54 1 0 0 0 0 | + | 53 54 1 0 0 0 0 |
| − | 54 55 1 0 0 0 0 | + | 54 55 1 0 0 0 0 |
| − | 22 26 1 1 0 0 0 | + | 22 26 1 1 0 0 0 |
| − | 23 27 1 1 0 0 0 | + | 23 27 1 1 0 0 0 |
| − | 31 30 1 0 0 0 0 | + | 31 30 1 0 0 0 0 |
| − | 30 33 1 0 0 0 0 | + | 30 33 1 0 0 0 0 |
| − | 30 32 2 0 0 0 0 | + | 30 32 2 0 0 0 0 |
| − | 27 30 1 0 0 0 0 | + | 27 30 1 0 0 0 0 |
| − | 55 1 1 0 0 0 0 | + | 55 1 1 0 0 0 0 |
| − | 1 2 1 0 0 0 0 | + | 1 2 1 0 0 0 0 |
| − | 2 3 1 0 0 0 0 | + | 2 3 1 0 0 0 0 |
| − | 3 4 1 0 0 0 0 | + | 3 4 1 0 0 0 0 |
| − | 4 5 1 0 0 0 0 | + | 4 5 1 0 0 0 0 |
| − | 5 6 1 0 0 0 0 | + | 5 6 1 0 0 0 0 |
| − | 6 7 1 0 0 0 0 | + | 6 7 1 0 0 0 0 |
| − | 7 8 1 0 0 0 0 | + | 7 8 1 0 0 0 0 |
| − | 1 9 2 0 0 0 0 | + | 1 9 2 0 0 0 0 |
| − | 3 10 1 1 0 0 0 | + | 3 10 1 1 0 0 0 |
| − | S SKP 7 | + | S SKP 7 |
| − | ID BMFYS8ESa001 | + | ID BMFYS8ESa001 |
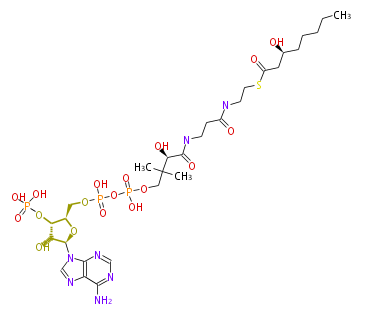
| − | NAME (S)-3-Hydroxy-octanoyl-CoA | + | NAME (S)-3-Hydroxy-octanoyl-CoA |
| − | FORMULA C29H50N7O18P3S | + | FORMULA C29H50N7O18P3S |
| − | EXACTMASS 909.2145 | + | EXACTMASS 909.2145 |
| − | AVERAGEMASS 909.7309 | + | AVERAGEMASS 909.7309 |
| − | SMILES C(C([C@H](C(=O)NCCC(NCCSC(=O)C[C@H](CCCCC)O)=O)O)(C)C)OP(OP(OC[C@H]([C@@H](OP(O)(O)=O)1)O[C@@H](n(c23)cnc2c(ncn3)N)[C@@H]1O)(O)=O)(O)=O | + | SMILES C(C([C@H](C(=O)NCCC(NCCSC(=O)C[C@H](CCCCC)O)=O)O)(C)C)OP(OP(OC[C@H]([C@@H](OP(O)(O)=O)1)O[C@@H](n(c23)cnc2c(ncn3)N)[C@@H]1O)(O)=O)(O)=O |
| − | KEGG http://www.genome.jp/dbget-bin/www_bget?compound+C05266 | + | KEGG http://www.genome.jp/dbget-bin/www_bget?compound+C05266 |
M END | M END | ||
| − | |||
Latest revision as of 09:00, 14 March 2009
Copyright: ARM project http://www.metabolome.jp/
58 60 0 0 1 0 0 0 0 0999 V2000
7.9200 -3.5075 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
6.9418 -3.2996 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
6.2727 -4.0427 0.0000 C 0 0 2 0 0 0 0 0 0 0 0 0
5.2946 -3.8348 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
4.6254 -4.5780 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.6473 -4.3701 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.9781 -5.1132 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.0000 -4.9053 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
8.2290 -4.4586 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
6.5817 -4.9938 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
25.2771 -2.4776 0.0000 N 0 0 0 0 0 0 0 0 0 0 0 0
24.4111 -1.9776 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
24.4111 -0.9776 0.0000 N 0 0 0 0 0 0 0 0 0 0 0 0
25.2771 -0.4776 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
26.1431 -0.9776 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
26.1431 -1.9776 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
26.8863 -0.3084 0.0000 N 0 0 0 0 0 0 0 0 0 0 0 0
26.4795 0.6051 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
25.4850 0.5006 0.0000 N 0 0 0 0 0 0 0 0 0 0 0 0
27.0091 -2.4776 0.0000 N 0 0 0 0 0 0 0 0 0 0 0 0
24.8159 1.2437 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0
25.0238 2.2219 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0
24.1578 2.7219 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0
23.4146 2.0527 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0
22.4365 2.2607 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
25.9373 2.6286 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
24.0532 3.7164 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
23.8214 1.1392 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
21.7673 1.5175 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
24.8622 4.3042 0.0000 P 0 0 0 0 0 0 0 0 0 0 0 0
25.4500 3.4952 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
24.2745 5.1132 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
25.6713 4.8920 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
20.7892 1.7254 0.0000 P 0 0 0 0 0 0 0 0 0 0 0 0
20.5813 0.7473 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
20.9971 2.7036 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
19.8110 1.9333 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
19.1419 1.1902 0.0000 P 0 0 0 0 0 0 0 0 0 0 0 0
19.8851 0.5211 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
18.3988 1.8593 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
18.4728 0.4470 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
17.4946 0.6550 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
16.8255 -0.0882 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
17.5686 -0.7573 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
16.0824 0.5809 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
16.1564 -0.8313 0.0000 C 0 0 2 0 0 0 0 0 0 0 0 0
15.1782 -0.6234 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
14.5091 -1.3666 0.0000 N 0 0 0 0 0 0 0 0 0 0 0 0
13.5309 -1.1587 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
12.8618 -1.9018 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
11.8837 -1.6939 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
11.2145 -2.4370 0.0000 N 0 0 0 0 0 0 0 0 0 0 0 0
10.2364 -2.2291 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
9.5673 -2.9723 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
8.5891 -2.7644 0.0000 S 0 0 0 0 0 0 0 0 0 0 0 0
16.4654 -1.7824 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
14.8692 0.3276 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
11.5747 -0.7428 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
11 16 2 0 0 0 0
11 12 1 0 0 0 0
12 13 2 0 0 0 0
13 14 1 0 0 0 0
15 16 1 0 0 0 0
15 14 2 0 0 0 0
19 18 1 0 0 0 0
18 17 2 0 0 0 0
17 15 1 0 0 0 0
14 19 1 0 0 0 0
16 20 1 0 0 0 0
24 28 1 6 0 0 0
23 24 1 0 0 0 0
21 28 1 6 0 0 0
23 22 1 0 0 0 0
21 22 1 0 0 0 0
24 25 1 0 0 0 0
25 29 1 0 0 0 0
21 19 1 0 0 0 0
29 34 1 0 0 0 0
34 36 1 0 0 0 0
34 35 2 0 0 0 0
34 37 1 0 0 0 0
41 38 1 0 0 0 0
38 40 2 0 0 0 0
38 39 1 0 0 0 0
41 42 1 0 0 0 0
38 37 1 0 0 0 0
42 43 1 0 0 0 0
43 46 1 0 0 0 0
43 45 1 0 0 0 0
43 44 1 0 0 0 0
46 47 1 0 0 0 0
46 56 1 1 0 0 0
47 57 2 0 0 0 0
47 48 1 0 0 0 0
48 49 1 0 0 0 0
49 50 1 0 0 0 0
50 51 1 0 0 0 0
51 52 1 0 0 0 0
51 58 2 0 0 0 0
52 53 1 0 0 0 0
53 54 1 0 0 0 0
54 55 1 0 0 0 0
22 26 1 1 0 0 0
23 27 1 1 0 0 0
31 30 1 0 0 0 0
30 33 1 0 0 0 0
30 32 2 0 0 0 0
27 30 1 0 0 0 0
55 1 1 0 0 0 0
1 2 1 0 0 0 0
2 3 1 0 0 0 0
3 4 1 0 0 0 0
4 5 1 0 0 0 0
5 6 1 0 0 0 0
6 7 1 0 0 0 0
7 8 1 0 0 0 0
1 9 2 0 0 0 0
3 10 1 1 0 0 0
S SKP 7
ID BMFYS8ESa001
NAME (S)-3-Hydroxy-octanoyl-CoA
FORMULA C29H50N7O18P3S
EXACTMASS 909.2145
AVERAGEMASS 909.7309
SMILES C(C([C@H](C(=O)NCCC(NCCSC(=O)C[C@H](CCCCC)O)=O)O)(C)C)OP(OP(OC[C@H]([C@@H](OP(O)(O)=O)1)O[C@@H](n(c23)cnc2c(ncn3)N)[C@@H]1O)(O)=O)(O)=O
KEGG http://www.genome.jp/dbget-bin/www_bget?compound+C05266
M END