Mol:BMFYB4HOa001
From Metabolomics.JP
(Difference between revisions)
| Line 1: | Line 1: | ||
| − | + | ||
| − | + | ||
| − | Copyright: ARM project http://www.metabolome.jp/ | + | Copyright: ARM project http://www.metabolome.jp/ |
| − | 55 57 0 0 1 0 0 0 0 0999 V2000 | + | 55 57 0 0 1 0 0 0 0 0999 V2000 |
| − | 4.3904 -3.6712 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 4.3904 -3.6712 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.4123 -3.4632 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.4123 -3.4632 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.7431 -4.2064 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.7431 -4.2064 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.4863 -4.8755 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.4863 -4.8755 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.0000 -3.5373 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.0000 -3.5373 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.0740 -4.9495 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.0740 -4.9495 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 4.6994 -4.6222 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 4.6994 -4.6222 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 21.7475 -2.6412 0.0000 N 0 0 0 0 0 0 0 0 0 0 0 0 | + | 21.7475 -2.6412 0.0000 N 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 20.8815 -2.1412 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 20.8815 -2.1412 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 20.8815 -1.1412 0.0000 N 0 0 0 0 0 0 0 0 0 0 0 0 | + | 20.8815 -1.1412 0.0000 N 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 21.7475 -0.6412 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 21.7475 -0.6412 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 22.6136 -1.1412 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 22.6136 -1.1412 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 22.6136 -2.1412 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 22.6136 -2.1412 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 23.3567 -0.4721 0.0000 N 0 0 0 0 0 0 0 0 0 0 0 0 | + | 23.3567 -0.4721 0.0000 N 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 22.9500 0.4414 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 22.9500 0.4414 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 21.9554 0.3369 0.0000 N 0 0 0 0 0 0 0 0 0 0 0 0 | + | 21.9554 0.3369 0.0000 N 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 23.4796 -2.6412 0.0000 N 0 0 0 0 0 0 0 0 0 0 0 0 | + | 23.4796 -2.6412 0.0000 N 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 21.2863 1.0801 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0 | + | 21.2863 1.0801 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0 |
| − | 21.4942 2.0582 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0 | + | 21.4942 2.0582 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0 |
| − | 20.6282 2.5582 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0 | + | 20.6282 2.5582 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0 |
| − | 19.8851 1.8891 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0 | + | 19.8851 1.8891 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0 |
| − | 18.9069 2.0970 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 18.9069 2.0970 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 22.4078 2.4650 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 22.4078 2.4650 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 20.5237 3.5527 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 20.5237 3.5527 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 20.2918 0.9755 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 20.2918 0.9755 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 18.2378 1.3539 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 18.2378 1.3539 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 21.3327 4.1405 0.0000 P 0 0 0 0 0 0 0 0 0 0 0 0 | + | 21.3327 4.1405 0.0000 P 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 21.9205 3.3315 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 21.9205 3.3315 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 20.7449 4.9495 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 20.7449 4.9495 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 22.1417 4.7283 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 22.1417 4.7283 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 17.2596 1.5618 0.0000 P 0 0 0 0 0 0 0 0 0 0 0 0 | + | 17.2596 1.5618 0.0000 P 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 17.0517 0.5836 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 17.0517 0.5836 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 17.4675 2.5399 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 17.4675 2.5399 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 16.2815 1.7697 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 16.2815 1.7697 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 15.6124 1.0265 0.0000 P 0 0 0 0 0 0 0 0 0 0 0 0 | + | 15.6124 1.0265 0.0000 P 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 16.3555 0.3574 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 16.3555 0.3574 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 14.8692 1.6957 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 14.8692 1.6957 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 14.9432 0.2834 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 14.9432 0.2834 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 13.9651 0.4913 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 13.9651 0.4913 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 13.2959 -0.2518 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 13.2959 -0.2518 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 14.0391 -0.9210 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 14.0391 -0.9210 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 12.5528 0.4173 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 12.5528 0.4173 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 12.6268 -0.9950 0.0000 C 0 0 2 0 0 0 0 0 0 0 0 0 | + | 12.6268 -0.9950 0.0000 C 0 0 2 0 0 0 0 0 0 0 0 0 |
| − | 11.6487 -0.7871 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 11.6487 -0.7871 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 10.9795 -1.5302 0.0000 N 0 0 0 0 0 0 0 0 0 0 0 0 | + | 10.9795 -1.5302 0.0000 N 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 10.0014 -1.3223 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 10.0014 -1.3223 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 9.3323 -2.0655 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 9.3323 -2.0655 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 8.3541 -1.8575 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 8.3541 -1.8575 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 7.6850 -2.6007 0.0000 N 0 0 0 0 0 0 0 0 0 0 0 0 | + | 7.6850 -2.6007 0.0000 N 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 6.7068 -2.3928 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 6.7068 -2.3928 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 6.0377 -3.1359 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 6.0377 -3.1359 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 5.0596 -2.9280 0.0000 S 0 0 0 0 0 0 0 0 0 0 0 0 | + | 5.0596 -2.9280 0.0000 S 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 12.9358 -1.9461 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 12.9358 -1.9461 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 11.3396 0.1640 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 11.3396 0.1640 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 8.0451 -0.9065 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 8.0451 -0.9065 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 35 36 2 0 0 0 0 | + | 35 36 2 0 0 0 0 |
| − | 11 10 1 0 0 0 0 | + | 11 10 1 0 0 0 0 |
| − | 35 38 1 0 0 0 0 | + | 35 38 1 0 0 0 0 |
| − | 15 14 2 0 0 0 0 | + | 15 14 2 0 0 0 0 |
| − | 38 39 1 0 0 0 0 | + | 38 39 1 0 0 0 0 |
| − | 19 20 1 0 0 0 0 | + | 19 20 1 0 0 0 0 |
| − | 39 40 1 0 0 0 0 | + | 39 40 1 0 0 0 0 |
| − | 19 23 1 1 0 0 0 | + | 19 23 1 1 0 0 0 |
| − | 40 43 1 0 0 0 0 | + | 40 43 1 0 0 0 0 |
| − | 21 25 1 6 0 0 0 | + | 21 25 1 6 0 0 0 |
| − | 43 44 1 0 0 0 0 | + | 43 44 1 0 0 0 0 |
| − | 12 13 1 0 0 0 0 | + | 12 13 1 0 0 0 0 |
| − | 44 45 1 0 0 0 0 | + | 44 45 1 0 0 0 0 |
| − | 10 9 2 0 0 0 0 | + | 10 9 2 0 0 0 0 |
| − | 45 46 1 0 0 0 0 | + | 45 46 1 0 0 0 0 |
| − | 20 24 1 1 0 0 0 | + | 20 24 1 1 0 0 0 |
| − | 46 47 1 0 0 0 0 | + | 46 47 1 0 0 0 0 |
| − | 21 22 1 0 0 0 0 | + | 21 22 1 0 0 0 0 |
| − | 47 48 1 0 0 0 0 | + | 47 48 1 0 0 0 0 |
| − | 13 8 2 0 0 0 0 | + | 13 8 2 0 0 0 0 |
| − | 48 49 1 0 0 0 0 | + | 48 49 1 0 0 0 0 |
| − | 13 17 1 0 0 0 0 | + | 13 17 1 0 0 0 0 |
| − | 49 50 1 0 0 0 0 | + | 49 50 1 0 0 0 0 |
| − | 22 26 1 0 0 0 0 | + | 22 26 1 0 0 0 0 |
| − | 43 53 1 1 0 0 0 | + | 43 53 1 1 0 0 0 |
| − | 12 14 1 0 0 0 0 | + | 12 14 1 0 0 0 0 |
| − | 44 54 2 0 0 0 0 | + | 44 54 2 0 0 0 0 |
| − | 20 21 1 0 0 0 0 | + | 20 21 1 0 0 0 0 |
| − | 9 8 1 0 0 0 0 | + | 9 8 1 0 0 0 0 |
| − | 50 51 1 0 0 0 0 | + | 50 51 1 0 0 0 0 |
| − | 51 52 1 0 0 0 0 | + | 51 52 1 0 0 0 0 |
| − | 26 31 1 0 0 0 0 | + | 26 31 1 0 0 0 0 |
| − | 40 42 1 0 0 0 0 | + | 40 42 1 0 0 0 0 |
| − | 18 16 1 0 0 0 0 | + | 18 16 1 0 0 0 0 |
| − | 40 41 1 0 0 0 0 | + | 40 41 1 0 0 0 0 |
| − | 31 34 1 0 0 0 0 | + | 31 34 1 0 0 0 0 |
| − | 52 1 1 0 0 0 0 | + | 52 1 1 0 0 0 0 |
| − | 16 11 1 0 0 0 0 | + | 16 11 1 0 0 0 0 |
| − | 1 2 1 0 0 0 0 | + | 1 2 1 0 0 0 0 |
| − | 34 35 1 0 0 0 0 | + | 34 35 1 0 0 0 0 |
| − | 2 3 1 0 0 0 0 | + | 2 3 1 0 0 0 0 |
| − | 16 15 1 0 0 0 0 | + | 16 15 1 0 0 0 0 |
| − | 3 6 1 0 0 0 0 | + | 3 6 1 0 0 0 0 |
| − | 31 33 1 0 0 0 0 | + | 31 33 1 0 0 0 0 |
| − | 3 5 1 0 0 0 0 | + | 3 5 1 0 0 0 0 |
| − | 19 18 1 0 0 0 0 | + | 19 18 1 0 0 0 0 |
| − | 3 4 1 0 0 0 0 | + | 3 4 1 0 0 0 0 |
| − | 35 37 1 0 0 0 0 | + | 35 37 1 0 0 0 0 |
| − | 24 27 1 0 0 0 0 | + | 24 27 1 0 0 0 0 |
| − | 18 25 1 6 0 0 0 | + | 18 25 1 6 0 0 0 |
| − | 27 28 1 0 0 0 0 | + | 27 28 1 0 0 0 0 |
| − | 31 32 2 0 0 0 0 | + | 31 32 2 0 0 0 0 |
| − | 27 30 2 0 0 0 0 | + | 27 30 2 0 0 0 0 |
| − | 11 12 2 0 0 0 0 | + | 11 12 2 0 0 0 0 |
| − | 27 29 1 0 0 0 0 | + | 27 29 1 0 0 0 0 |
| − | 1 7 2 0 0 0 0 | + | 1 7 2 0 0 0 0 |
| − | 48 55 2 0 0 0 0 | + | 48 55 2 0 0 0 0 |
| − | S SKP 7 | + | S SKP 7 |
| − | ID BMFYB4HOa001 | + | ID BMFYB4HOa001 |
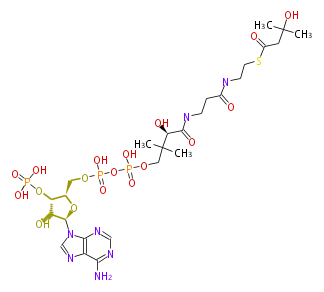
| − | NAME 3-Hydroxy-isopentyl-CoA | + | NAME 3-Hydroxy-isopentyl-CoA |
| − | FORMULA C26H44N7O18P3S | + | FORMULA C26H44N7O18P3S |
| − | EXACTMASS 867.1676 | + | EXACTMASS 867.1676 |
| − | AVERAGEMASS 867.6512 | + | AVERAGEMASS 867.6512 |
| − | SMILES C([C@H](C(NCCC(NCCSC(CC(C)(C)O)=O)=O)=O)O)(C)(C)COP(OP(OC[C@H]([C@@H](OP(O)(O)=O)3)O[C@H]([C@@H]3O)n(c21)cnc(c(N)ncn2)1)(O)=O)(O)=O | + | SMILES C([C@H](C(NCCC(NCCSC(CC(C)(C)O)=O)=O)=O)O)(C)(C)COP(OP(OC[C@H]([C@@H](OP(O)(O)=O)3)O[C@H]([C@@H]3O)n(c21)cnc(c(N)ncn2)1)(O)=O)(O)=O |
| − | KEGG http://www.genome.jp/dbget-bin/www_bget?compound+C05997 | + | KEGG http://www.genome.jp/dbget-bin/www_bget?compound+C05997 |
M END | M END | ||
| − | |||
Revision as of 09:00, 14 March 2009
Copyright: ARM project http://www.metabolome.jp/
55 57 0 0 1 0 0 0 0 0999 V2000
4.3904 -3.6712 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.4123 -3.4632 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.7431 -4.2064 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.4863 -4.8755 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.0000 -3.5373 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.0740 -4.9495 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
4.6994 -4.6222 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
21.7475 -2.6412 0.0000 N 0 0 0 0 0 0 0 0 0 0 0 0
20.8815 -2.1412 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
20.8815 -1.1412 0.0000 N 0 0 0 0 0 0 0 0 0 0 0 0
21.7475 -0.6412 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
22.6136 -1.1412 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
22.6136 -2.1412 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
23.3567 -0.4721 0.0000 N 0 0 0 0 0 0 0 0 0 0 0 0
22.9500 0.4414 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
21.9554 0.3369 0.0000 N 0 0 0 0 0 0 0 0 0 0 0 0
23.4796 -2.6412 0.0000 N 0 0 0 0 0 0 0 0 0 0 0 0
21.2863 1.0801 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0
21.4942 2.0582 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0
20.6282 2.5582 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0
19.8851 1.8891 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0
18.9069 2.0970 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
22.4078 2.4650 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
20.5237 3.5527 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
20.2918 0.9755 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
18.2378 1.3539 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
21.3327 4.1405 0.0000 P 0 0 0 0 0 0 0 0 0 0 0 0
21.9205 3.3315 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
20.7449 4.9495 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
22.1417 4.7283 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
17.2596 1.5618 0.0000 P 0 0 0 0 0 0 0 0 0 0 0 0
17.0517 0.5836 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
17.4675 2.5399 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
16.2815 1.7697 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
15.6124 1.0265 0.0000 P 0 0 0 0 0 0 0 0 0 0 0 0
16.3555 0.3574 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
14.8692 1.6957 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
14.9432 0.2834 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
13.9651 0.4913 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
13.2959 -0.2518 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
14.0391 -0.9210 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
12.5528 0.4173 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
12.6268 -0.9950 0.0000 C 0 0 2 0 0 0 0 0 0 0 0 0
11.6487 -0.7871 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
10.9795 -1.5302 0.0000 N 0 0 0 0 0 0 0 0 0 0 0 0
10.0014 -1.3223 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
9.3323 -2.0655 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
8.3541 -1.8575 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
7.6850 -2.6007 0.0000 N 0 0 0 0 0 0 0 0 0 0 0 0
6.7068 -2.3928 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
6.0377 -3.1359 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
5.0596 -2.9280 0.0000 S 0 0 0 0 0 0 0 0 0 0 0 0
12.9358 -1.9461 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
11.3396 0.1640 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
8.0451 -0.9065 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
35 36 2 0 0 0 0
11 10 1 0 0 0 0
35 38 1 0 0 0 0
15 14 2 0 0 0 0
38 39 1 0 0 0 0
19 20 1 0 0 0 0
39 40 1 0 0 0 0
19 23 1 1 0 0 0
40 43 1 0 0 0 0
21 25 1 6 0 0 0
43 44 1 0 0 0 0
12 13 1 0 0 0 0
44 45 1 0 0 0 0
10 9 2 0 0 0 0
45 46 1 0 0 0 0
20 24 1 1 0 0 0
46 47 1 0 0 0 0
21 22 1 0 0 0 0
47 48 1 0 0 0 0
13 8 2 0 0 0 0
48 49 1 0 0 0 0
13 17 1 0 0 0 0
49 50 1 0 0 0 0
22 26 1 0 0 0 0
43 53 1 1 0 0 0
12 14 1 0 0 0 0
44 54 2 0 0 0 0
20 21 1 0 0 0 0
9 8 1 0 0 0 0
50 51 1 0 0 0 0
51 52 1 0 0 0 0
26 31 1 0 0 0 0
40 42 1 0 0 0 0
18 16 1 0 0 0 0
40 41 1 0 0 0 0
31 34 1 0 0 0 0
52 1 1 0 0 0 0
16 11 1 0 0 0 0
1 2 1 0 0 0 0
34 35 1 0 0 0 0
2 3 1 0 0 0 0
16 15 1 0 0 0 0
3 6 1 0 0 0 0
31 33 1 0 0 0 0
3 5 1 0 0 0 0
19 18 1 0 0 0 0
3 4 1 0 0 0 0
35 37 1 0 0 0 0
24 27 1 0 0 0 0
18 25 1 6 0 0 0
27 28 1 0 0 0 0
31 32 2 0 0 0 0
27 30 2 0 0 0 0
11 12 2 0 0 0 0
27 29 1 0 0 0 0
1 7 2 0 0 0 0
48 55 2 0 0 0 0
S SKP 7
ID BMFYB4HOa001
NAME 3-Hydroxy-isopentyl-CoA
FORMULA C26H44N7O18P3S
EXACTMASS 867.1676
AVERAGEMASS 867.6512
SMILES C([C@H](C(NCCC(NCCSC(CC(C)(C)O)=O)=O)=O)O)(C)(C)COP(OP(OC[C@H]([C@@H](OP(O)(O)=O)3)O[C@H]([C@@H]3O)n(c21)cnc(c(N)ncn2)1)(O)=O)(O)=O
KEGG http://www.genome.jp/dbget-bin/www_bget?compound+C05997
M END