Mol:BMCCPUAPS502
From Metabolomics.JP
(Difference between revisions)
| Line 1: | Line 1: | ||
| − | + | ||
| − | + | ||
| − | Copyright: ARM project http://www.metabolome.jp/ | + | Copyright: ARM project http://www.metabolome.jp/ |
| − | 36 39 0 0 1 0 0 0 0 0999 V2000 | + | 36 39 0 0 1 0 0 0 0 0999 V2000 |
| − | 2.9781 -1.4750 0.0000 C 0 0 2 0 0 0 0 0 0 0 0 0 | + | 2.9781 -1.4750 0.0000 C 0 0 2 0 0 0 0 0 0 0 0 0 |
| − | 3.7213 -2.1441 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0 | + | 3.7213 -2.1441 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0 |
| − | 4.5873 -1.6441 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0 | + | 4.5873 -1.6441 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0 |
| − | 4.3794 -0.6660 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0 | + | 4.3794 -0.6660 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0 |
| − | 5.0485 0.0772 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 5.0485 0.0772 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.0000 -1.6829 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.0000 -1.6829 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.6168 -3.1386 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.6168 -3.1386 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 5.5009 -2.0508 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 5.5009 -2.0508 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.3849 -0.5614 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.3849 -0.5614 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 6.0267 -0.1307 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 6.0267 -0.1307 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 12.8310 -3.0553 0.0000 N 0 0 0 0 0 0 0 0 0 0 0 0 | + | 12.8310 -3.0553 0.0000 N 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 11.9650 -2.5553 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 11.9650 -2.5553 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 11.9650 -1.5553 0.0000 N 0 0 0 0 0 0 0 0 0 0 0 0 | + | 11.9650 -1.5553 0.0000 N 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 12.8310 -1.0553 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 12.8310 -1.0553 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 13.6970 -1.5553 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 13.6970 -1.5553 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 13.6970 -2.5553 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 13.6970 -2.5553 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 14.4402 -0.8862 0.0000 N 0 0 0 0 0 0 0 0 0 0 0 0 | + | 14.4402 -0.8862 0.0000 N 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 14.0334 0.0273 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 14.0334 0.0273 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 13.0389 -0.0772 0.0000 N 0 0 0 0 0 0 0 0 0 0 0 0 | + | 13.0389 -0.0772 0.0000 N 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 14.5630 -3.0553 0.0000 N 0 0 0 0 0 0 0 0 0 0 0 0 | + | 14.5630 -3.0553 0.0000 N 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 12.3698 0.6660 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0 | + | 12.3698 0.6660 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0 |
| − | 12.5777 1.6441 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0 | + | 12.5777 1.6441 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0 |
| − | 11.7117 2.1441 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0 | + | 11.7117 2.1441 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0 |
| − | 10.9685 1.4750 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0 | + | 10.9685 1.4750 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0 |
| − | 9.9904 1.6829 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 9.9904 1.6829 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 13.4912 2.0508 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 13.4912 2.0508 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 11.6071 3.1386 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 11.6071 3.1386 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 11.3753 0.5614 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 11.3753 0.5614 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 9.3212 0.9397 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 9.3212 0.9397 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 8.3431 1.1477 0.0000 P 0 0 0 0 0 0 0 0 0 0 0 0 | + | 8.3431 1.1477 0.0000 P 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 8.1352 0.1695 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 8.1352 0.1695 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 8.5510 2.1258 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 8.5510 2.1258 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 7.3649 1.3556 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 7.3649 1.3556 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 6.6958 0.6124 0.0000 P 0 0 0 0 0 0 0 0 0 0 0 0 | + | 6.6958 0.6124 0.0000 P 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 7.4390 -0.0567 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 7.4390 -0.0567 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 5.9527 1.2816 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 5.9527 1.2816 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 14 19 1 0 0 0 0 | + | 14 19 1 0 0 0 0 |
| − | 2 7 1 1 0 0 0 | + | 2 7 1 1 0 0 0 |
| − | 19 18 1 0 0 0 0 | + | 19 18 1 0 0 0 0 |
| − | 3 8 1 1 0 0 0 | + | 3 8 1 1 0 0 0 |
| − | 18 17 2 0 0 0 0 | + | 18 17 2 0 0 0 0 |
| − | 5 10 1 0 0 0 0 | + | 5 10 1 0 0 0 0 |
| − | 16 11 1 0 0 0 0 | + | 16 11 1 0 0 0 0 |
| − | 10 34 1 0 0 0 0 | + | 10 34 1 0 0 0 0 |
| − | 16 20 1 0 0 0 0 | + | 16 20 1 0 0 0 0 |
| − | 34 35 2 0 0 0 0 | + | 34 35 2 0 0 0 0 |
| − | 15 14 1 0 0 0 0 | + | 15 14 1 0 0 0 0 |
| − | 34 36 1 0 0 0 0 | + | 34 36 1 0 0 0 0 |
| − | 34 33 1 0 0 0 0 | + | 34 33 1 0 0 0 0 |
| − | 11 12 2 0 0 0 0 | + | 11 12 2 0 0 0 0 |
| − | 33 30 1 0 0 0 0 | + | 33 30 1 0 0 0 0 |
| − | 12 13 1 0 0 0 0 | + | 12 13 1 0 0 0 0 |
| − | 30 32 1 0 0 0 0 | + | 30 32 1 0 0 0 0 |
| − | 13 14 2 0 0 0 0 | + | 13 14 2 0 0 0 0 |
| − | 30 31 2 0 0 0 0 | + | 30 31 2 0 0 0 0 |
| − | 3 4 1 0 0 0 0 | + | 3 4 1 0 0 0 0 |
| − | 30 29 1 0 0 0 0 | + | 30 29 1 0 0 0 0 |
| − | 4 9 1 6 0 0 0 | + | 4 9 1 6 0 0 0 |
| − | 9 1 1 0 0 0 0 | + | 9 1 1 0 0 0 0 |
| − | 1 2 1 0 0 0 0 | + | 1 2 1 0 0 0 0 |
| − | 2 3 1 0 0 0 0 | + | 2 3 1 0 0 0 0 |
| − | 15 16 2 0 0 0 0 | + | 15 16 2 0 0 0 0 |
| − | 22 21 1 0 0 0 0 | + | 22 21 1 0 0 0 0 |
| − | 21 28 1 6 0 0 0 | + | 21 28 1 6 0 0 0 |
| − | 24 28 1 6 0 0 0 | + | 24 28 1 6 0 0 0 |
| − | 24 23 1 0 0 0 0 | + | 24 23 1 0 0 0 0 |
| − | 23 22 1 0 0 0 0 | + | 23 22 1 0 0 0 0 |
| − | 4 5 1 0 0 0 0 | + | 4 5 1 0 0 0 0 |
| − | 22 26 1 1 0 0 0 | + | 22 26 1 1 0 0 0 |
| − | 17 15 1 0 0 0 0 | + | 17 15 1 0 0 0 0 |
| − | 23 27 1 1 0 0 0 | + | 23 27 1 1 0 0 0 |
| − | 24 25 1 0 0 0 0 | + | 24 25 1 0 0 0 0 |
| − | 29 25 1 0 0 0 0 | + | 29 25 1 0 0 0 0 |
| − | 21 19 1 0 0 0 0 | + | 21 19 1 0 0 0 0 |
| − | 1 6 1 6 0 0 0 | + | 1 6 1 6 0 0 0 |
| − | S SKP 7 | + | S SKP 7 |
| − | ID BMCCPUAPS502 | + | ID BMCCPUAPS502 |
| − | NAME ADP-5-ribose | + | NAME ADP-5-ribose |
| − | FORMULA C15H23N5O14P2 | + | FORMULA C15H23N5O14P2 |
| − | EXACTMASS 559.0716 | + | EXACTMASS 559.0716 |
| − | AVERAGEMASS 559.3159 | + | AVERAGEMASS 559.3159 |
| − | SMILES Nc(n4)c(n3)c(nc4)n(c3)[C@H](O1)[C@H](O)[C@H](O)[C@@H](COP(O)(=O)OP(O)(=O)OC[C@@H](O2)[C@@H](O)[C@@H](O)[C@H](O)2)1 | + | SMILES Nc(n4)c(n3)c(nc4)n(c3)[C@H](O1)[C@H](O)[C@H](O)[C@@H](COP(O)(=O)OP(O)(=O)OC[C@@H](O2)[C@@H](O)[C@@H](O)[C@H](O)2)1 |
| − | KEGG http://www.genome.jp/dbget-bin/www_bget?compound+C00301 | + | KEGG http://www.genome.jp/dbget-bin/www_bget?compound+C00301 |
M END | M END | ||
| − | |||
Latest revision as of 09:00, 14 March 2009
Copyright: ARM project http://www.metabolome.jp/
36 39 0 0 1 0 0 0 0 0999 V2000
2.9781 -1.4750 0.0000 C 0 0 2 0 0 0 0 0 0 0 0 0
3.7213 -2.1441 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0
4.5873 -1.6441 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0
4.3794 -0.6660 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0
5.0485 0.0772 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.0000 -1.6829 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
3.6168 -3.1386 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
5.5009 -2.0508 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
3.3849 -0.5614 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
6.0267 -0.1307 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
12.8310 -3.0553 0.0000 N 0 0 0 0 0 0 0 0 0 0 0 0
11.9650 -2.5553 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
11.9650 -1.5553 0.0000 N 0 0 0 0 0 0 0 0 0 0 0 0
12.8310 -1.0553 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
13.6970 -1.5553 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
13.6970 -2.5553 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
14.4402 -0.8862 0.0000 N 0 0 0 0 0 0 0 0 0 0 0 0
14.0334 0.0273 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
13.0389 -0.0772 0.0000 N 0 0 0 0 0 0 0 0 0 0 0 0
14.5630 -3.0553 0.0000 N 0 0 0 0 0 0 0 0 0 0 0 0
12.3698 0.6660 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0
12.5777 1.6441 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0
11.7117 2.1441 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0
10.9685 1.4750 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0
9.9904 1.6829 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
13.4912 2.0508 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
11.6071 3.1386 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
11.3753 0.5614 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
9.3212 0.9397 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
8.3431 1.1477 0.0000 P 0 0 0 0 0 0 0 0 0 0 0 0
8.1352 0.1695 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
8.5510 2.1258 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
7.3649 1.3556 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
6.6958 0.6124 0.0000 P 0 0 0 0 0 0 0 0 0 0 0 0
7.4390 -0.0567 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
5.9527 1.2816 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
14 19 1 0 0 0 0
2 7 1 1 0 0 0
19 18 1 0 0 0 0
3 8 1 1 0 0 0
18 17 2 0 0 0 0
5 10 1 0 0 0 0
16 11 1 0 0 0 0
10 34 1 0 0 0 0
16 20 1 0 0 0 0
34 35 2 0 0 0 0
15 14 1 0 0 0 0
34 36 1 0 0 0 0
34 33 1 0 0 0 0
11 12 2 0 0 0 0
33 30 1 0 0 0 0
12 13 1 0 0 0 0
30 32 1 0 0 0 0
13 14 2 0 0 0 0
30 31 2 0 0 0 0
3 4 1 0 0 0 0
30 29 1 0 0 0 0
4 9 1 6 0 0 0
9 1 1 0 0 0 0
1 2 1 0 0 0 0
2 3 1 0 0 0 0
15 16 2 0 0 0 0
22 21 1 0 0 0 0
21 28 1 6 0 0 0
24 28 1 6 0 0 0
24 23 1 0 0 0 0
23 22 1 0 0 0 0
4 5 1 0 0 0 0
22 26 1 1 0 0 0
17 15 1 0 0 0 0
23 27 1 1 0 0 0
24 25 1 0 0 0 0
29 25 1 0 0 0 0
21 19 1 0 0 0 0
1 6 1 6 0 0 0
S SKP 7
ID BMCCPUAPS502
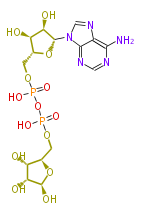
NAME ADP-5-ribose
FORMULA C15H23N5O14P2
EXACTMASS 559.0716
AVERAGEMASS 559.3159
SMILES Nc(n4)c(n3)c(nc4)n(c3)[C@H](O1)[C@H](O)[C@H](O)[C@@H](COP(O)(=O)OP(O)(=O)OC[C@@H](O2)[C@@H](O)[C@@H](O)[C@H](O)2)1
KEGG http://www.genome.jp/dbget-bin/www_bget?compound+C00301
M END