Mol:BMCCPTFLe002
From Metabolomics.JP
(Difference between revisions)
| Line 1: | Line 1: | ||
| − | + | ||
| − | + | ||
| − | Copyright: ARM project http://www.metabolome.jp/ | + | Copyright: ARM project http://www.metabolome.jp/ |
| − | 53 55 0 0 1 0 0 0 0 0999 V2000 | + | 53 55 0 0 1 0 0 0 0 0999 V2000 |
| − | 6.9478 -2.5260 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 6.9478 -2.5260 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 6.9478 -1.6919 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 6.9478 -1.6919 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 6.2334 -2.9385 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 6.2334 -2.9385 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 5.5097 -1.6919 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 5.5097 -1.6919 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 4.7810 -1.2731 0.0000 N 0 0 0 0 0 0 0 0 0 0 0 0 | + | 4.7810 -1.2731 0.0000 N 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 4.7867 -2.9500 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 4.7867 -2.9500 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 5.5097 -2.5260 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 5.5097 -2.5260 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 4.0706 -1.6747 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 4.0706 -1.6747 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.3129 -1.2704 0.0000 N 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.3129 -1.2704 0.0000 N 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.5794 -1.6951 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.5794 -1.6951 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.5910 -2.5467 0.0000 N 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.5910 -2.5467 0.0000 N 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.3303 -2.9648 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.3303 -2.9648 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 4.0580 -2.5313 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 4.0580 -2.5313 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 6.2238 -1.2786 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 6.2238 -1.2786 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.3122 -3.7897 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.3122 -3.7897 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.8619 -1.2880 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.8619 -1.2880 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 4.8145 0.0790 0.0000 C 0 0 3 0 0 0 0 0 0 0 0 0 | + | 4.8145 0.0790 0.0000 C 0 0 3 0 0 0 0 0 0 0 0 0 |
| − | 4.8212 0.9023 0.0000 C 0 0 3 0 0 0 0 0 0 0 0 0 | + | 4.8212 0.9023 0.0000 C 0 0 3 0 0 0 0 0 0 0 0 0 |
| − | 4.8212 1.7273 0.0000 C 0 0 3 0 0 0 0 0 0 0 0 0 | + | 4.8212 1.7273 0.0000 C 0 0 3 0 0 0 0 0 0 0 0 0 |
| − | 4.8212 2.5523 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 4.8212 2.5523 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.9895 0.0832 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.9895 0.0832 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.9962 0.9040 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.9962 0.9040 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.9962 1.7273 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.9962 1.7273 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 4.1068 2.9648 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 4.1068 2.9648 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.2859 2.9648 0.0000 P 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.2859 2.9648 0.0000 P 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.4609 2.9648 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.4609 2.9648 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.2859 2.1398 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.2859 2.1398 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.2900 3.7897 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.2900 3.7897 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.7504 2.5476 0.0000 C 0 0 2 0 0 0 0 0 0 0 0 0 | + | 1.7504 2.5476 0.0000 C 0 0 2 0 0 0 0 0 0 0 0 0 |
| − | 1.0330 2.9550 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.0330 2.9550 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.0270 3.7799 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.0270 3.7799 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.2479 2.9373 0.0000 N 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.2479 2.9373 0.0000 N 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.9548 2.4909 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0 | + | -0.9548 2.4909 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0 |
| − | -1.6851 2.8749 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.6851 2.8749 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.3827 2.4345 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.3827 2.4345 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.1129 2.8185 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.1129 2.8185 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.9934 1.5323 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.9934 1.5323 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.6925 1.0943 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.6925 1.0943 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.2620 1.1507 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.2620 1.1507 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.1454 3.6428 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.1454 3.6428 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -4.0932 2.3048 0.0000 N 0 0 0 0 0 0 0 0 0 0 0 0 | + | -4.0932 2.3048 0.0000 N 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -4.8035 1.8877 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0 | + | -4.8035 1.8877 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0 |
| − | -5.5209 2.2950 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -5.5209 2.2950 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -6.2324 1.8773 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -6.2324 1.8773 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -6.9498 2.2845 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -6.9498 2.2845 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -7.6612 1.8669 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -7.6612 1.8669 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -6.9558 3.1095 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -6.9558 3.1095 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -4.7975 1.0628 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -4.7975 1.0628 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -5.5102 0.6474 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -5.5102 0.6474 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -4.0786 0.6578 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -4.0786 0.6578 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 7.6612 -1.2775 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 7.6612 -1.2775 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.7565 1.7228 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.7565 1.7228 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 5.5178 -0.5093 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 5.5178 -0.5093 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1 3 1 0 0 0 0 | + | 1 3 1 0 0 0 0 |
| − | 1 2 2 0 0 0 0 | + | 1 2 2 0 0 0 0 |
| − | 2 14 1 0 0 0 0 | + | 2 14 1 0 0 0 0 |
| − | 7 3 2 0 0 0 0 | + | 7 3 2 0 0 0 0 |
| − | 4 7 1 0 0 0 0 | + | 4 7 1 0 0 0 0 |
| − | 4 5 1 0 0 0 0 | + | 4 5 1 0 0 0 0 |
| − | 5 8 1 0 0 0 0 | + | 5 8 1 0 0 0 0 |
| − | 13 6 2 0 0 0 0 | + | 13 6 2 0 0 0 0 |
| − | 6 7 1 0 0 0 0 | + | 6 7 1 0 0 0 0 |
| − | 8 13 1 0 0 0 0 | + | 8 13 1 0 0 0 0 |
| − | 8 9 2 0 0 0 0 | + | 8 9 2 0 0 0 0 |
| − | 9 10 1 0 0 0 0 | + | 9 10 1 0 0 0 0 |
| − | 10 11 1 0 0 0 0 | + | 10 11 1 0 0 0 0 |
| − | 11 12 1 0 0 0 0 | + | 11 12 1 0 0 0 0 |
| − | 12 13 1 0 0 0 0 | + | 12 13 1 0 0 0 0 |
| − | 4 14 2 0 0 0 0 | + | 4 14 2 0 0 0 0 |
| − | 12 15 2 0 0 0 0 | + | 12 15 2 0 0 0 0 |
| − | 10 16 2 0 0 0 0 | + | 10 16 2 0 0 0 0 |
| − | 17 18 1 0 0 0 0 | + | 17 18 1 0 0 0 0 |
| − | 18 19 1 0 0 0 0 | + | 18 19 1 0 0 0 0 |
| − | 19 20 1 0 0 0 0 | + | 19 20 1 0 0 0 0 |
| − | 17 21 1 4 0 0 0 | + | 17 21 1 4 0 0 0 |
| − | 18 22 1 4 0 0 0 | + | 18 22 1 4 0 0 0 |
| − | 19 23 1 4 0 0 0 | + | 19 23 1 4 0 0 0 |
| − | 20 24 1 0 0 0 0 | + | 20 24 1 0 0 0 0 |
| − | 25 24 1 0 0 0 0 | + | 25 24 1 0 0 0 0 |
| − | 25 26 1 0 0 0 0 | + | 25 26 1 0 0 0 0 |
| − | 25 27 2 0 0 0 0 | + | 25 27 2 0 0 0 0 |
| − | 25 28 1 0 0 0 0 | + | 25 28 1 0 0 0 0 |
| − | 26 29 1 0 0 0 0 | + | 26 29 1 0 0 0 0 |
| − | 29 30 1 0 0 0 0 | + | 29 30 1 0 0 0 0 |
| − | 30 31 2 0 0 0 0 | + | 30 31 2 0 0 0 0 |
| − | 30 32 1 0 0 0 0 | + | 30 32 1 0 0 0 0 |
| − | 32 33 1 0 0 0 0 | + | 32 33 1 0 0 0 0 |
| − | 33 34 1 0 0 0 0 | + | 33 34 1 0 0 0 0 |
| − | 34 35 1 0 0 0 0 | + | 34 35 1 0 0 0 0 |
| − | 35 36 1 0 0 0 0 | + | 35 36 1 0 0 0 0 |
| − | 37 38 1 0 0 0 0 | + | 37 38 1 0 0 0 0 |
| − | 37 39 2 0 0 0 0 | + | 37 39 2 0 0 0 0 |
| − | 36 40 2 0 0 0 0 | + | 36 40 2 0 0 0 0 |
| − | 41 42 1 0 0 0 0 | + | 41 42 1 0 0 0 0 |
| − | 42 43 1 0 0 0 0 | + | 42 43 1 0 0 0 0 |
| − | 43 44 1 0 0 0 0 | + | 43 44 1 0 0 0 0 |
| − | 44 45 1 0 0 0 0 | + | 44 45 1 0 0 0 0 |
| − | 45 46 2 0 0 0 0 | + | 45 46 2 0 0 0 0 |
| − | 45 47 1 0 0 0 0 | + | 45 47 1 0 0 0 0 |
| − | 48 49 1 0 0 0 0 | + | 48 49 1 0 0 0 0 |
| − | 48 50 2 0 0 0 0 | + | 48 50 2 0 0 0 0 |
| − | 42 48 1 6 0 0 0 | + | 42 48 1 6 0 0 0 |
| − | 2 51 1 0 0 0 0 | + | 2 51 1 0 0 0 0 |
| − | 29 52 1 1 0 0 0 | + | 29 52 1 1 0 0 0 |
| − | 36 41 1 0 0 0 0 | + | 36 41 1 0 0 0 0 |
| − | 5 53 1 0 0 0 0 | + | 5 53 1 0 0 0 0 |
| − | 53 17 1 0 0 0 0 | + | 53 17 1 0 0 0 0 |
| − | 33 37 1 6 0 0 0 | + | 33 37 1 6 0 0 0 |
| − | S SKP 7 | + | S SKP 7 |
| − | ID BMCCPTFLe002 | + | ID BMCCPTFLe002 |
| − | NAME Coenzyme F420 | + | NAME Coenzyme F420 |
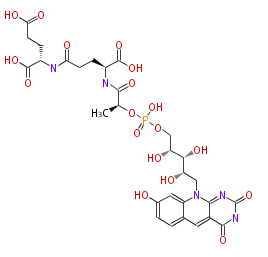
| − | FORMULA C29H36N5O18P | + | FORMULA C29H36N5O18P |
| − | EXACTMASS 773.1792 | + | EXACTMASS 773.1792 |
| − | AVERAGEMASS 773.5928 | + | AVERAGEMASS 773.5928 |
| − | SMILES P(O)(=O)(O[C@H](C(N[C@@H](CCC(N[C@H](C(O)=O)CCC(O)=O)=O)C(O)=O)=O)C)OCC(C(O)C(CN(C1=2)c(c3)c(ccc(O)3)C=C(C(NC(=O)N2)=O)1)O)O | + | SMILES P(O)(=O)(O[C@H](C(N[C@@H](CCC(N[C@H](C(O)=O)CCC(O)=O)=O)C(O)=O)=O)C)OCC(C(O)C(CN(C1=2)c(c3)c(ccc(O)3)C=C(C(NC(=O)N2)=O)1)O)O |
| − | KEGG http://www.genome.jp/dbget-bin/www_bget?compound+C00876 | + | KEGG http://www.genome.jp/dbget-bin/www_bget?compound+C00876 |
M END | M END | ||
| − | |||
Latest revision as of 09:00, 14 March 2009
Copyright: ARM project http://www.metabolome.jp/
53 55 0 0 1 0 0 0 0 0999 V2000
6.9478 -2.5260 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
6.9478 -1.6919 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
6.2334 -2.9385 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
5.5097 -1.6919 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
4.7810 -1.2731 0.0000 N 0 0 0 0 0 0 0 0 0 0 0 0
4.7867 -2.9500 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
5.5097 -2.5260 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
4.0706 -1.6747 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.3129 -1.2704 0.0000 N 0 0 0 0 0 0 0 0 0 0 0 0
2.5794 -1.6951 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.5910 -2.5467 0.0000 N 0 0 0 0 0 0 0 0 0 0 0 0
3.3303 -2.9648 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
4.0580 -2.5313 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
6.2238 -1.2786 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.3122 -3.7897 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
1.8619 -1.2880 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
4.8145 0.0790 0.0000 C 0 0 3 0 0 0 0 0 0 0 0 0
4.8212 0.9023 0.0000 C 0 0 3 0 0 0 0 0 0 0 0 0
4.8212 1.7273 0.0000 C 0 0 3 0 0 0 0 0 0 0 0 0
4.8212 2.5523 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.9895 0.0832 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
3.9962 0.9040 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
3.9962 1.7273 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
4.1068 2.9648 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
3.2859 2.9648 0.0000 P 0 0 0 0 0 0 0 0 0 0 0 0
2.4609 2.9648 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
3.2859 2.1398 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
3.2900 3.7897 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
1.7504 2.5476 0.0000 C 0 0 2 0 0 0 0 0 0 0 0 0
1.0330 2.9550 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.0270 3.7799 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-0.2479 2.9373 0.0000 N 0 0 0 0 0 0 0 0 0 0 0 0
-0.9548 2.4909 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0
-1.6851 2.8749 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.3827 2.4345 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-3.1129 2.8185 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.9934 1.5323 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.6925 1.0943 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-0.2620 1.1507 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-3.1454 3.6428 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-4.0932 2.3048 0.0000 N 0 0 0 0 0 0 0 0 0 0 0 0
-4.8035 1.8877 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0
-5.5209 2.2950 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-6.2324 1.8773 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-6.9498 2.2845 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-7.6612 1.8669 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-6.9558 3.1095 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-4.7975 1.0628 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-5.5102 0.6474 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-4.0786 0.6578 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
7.6612 -1.2775 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
1.7565 1.7228 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
5.5178 -0.5093 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1 3 1 0 0 0 0
1 2 2 0 0 0 0
2 14 1 0 0 0 0
7 3 2 0 0 0 0
4 7 1 0 0 0 0
4 5 1 0 0 0 0
5 8 1 0 0 0 0
13 6 2 0 0 0 0
6 7 1 0 0 0 0
8 13 1 0 0 0 0
8 9 2 0 0 0 0
9 10 1 0 0 0 0
10 11 1 0 0 0 0
11 12 1 0 0 0 0
12 13 1 0 0 0 0
4 14 2 0 0 0 0
12 15 2 0 0 0 0
10 16 2 0 0 0 0
17 18 1 0 0 0 0
18 19 1 0 0 0 0
19 20 1 0 0 0 0
17 21 1 4 0 0 0
18 22 1 4 0 0 0
19 23 1 4 0 0 0
20 24 1 0 0 0 0
25 24 1 0 0 0 0
25 26 1 0 0 0 0
25 27 2 0 0 0 0
25 28 1 0 0 0 0
26 29 1 0 0 0 0
29 30 1 0 0 0 0
30 31 2 0 0 0 0
30 32 1 0 0 0 0
32 33 1 0 0 0 0
33 34 1 0 0 0 0
34 35 1 0 0 0 0
35 36 1 0 0 0 0
37 38 1 0 0 0 0
37 39 2 0 0 0 0
36 40 2 0 0 0 0
41 42 1 0 0 0 0
42 43 1 0 0 0 0
43 44 1 0 0 0 0
44 45 1 0 0 0 0
45 46 2 0 0 0 0
45 47 1 0 0 0 0
48 49 1 0 0 0 0
48 50 2 0 0 0 0
42 48 1 6 0 0 0
2 51 1 0 0 0 0
29 52 1 1 0 0 0
36 41 1 0 0 0 0
5 53 1 0 0 0 0
53 17 1 0 0 0 0
33 37 1 6 0 0 0
S SKP 7
ID BMCCPTFLe002
NAME Coenzyme F420
FORMULA C29H36N5O18P
EXACTMASS 773.1792
AVERAGEMASS 773.5928
SMILES P(O)(=O)(O[C@H](C(N[C@@H](CCC(N[C@H](C(O)=O)CCC(O)=O)=O)C(O)=O)=O)C)OCC(C(O)C(CN(C1=2)c(c3)c(ccc(O)3)C=C(C(NC(=O)N2)=O)1)O)O
KEGG http://www.genome.jp/dbget-bin/www_bget?compound+C00876
M END