Mol:BMCCPPPC0003
From Metabolomics.JP
(Difference between revisions)
| Line 1: | Line 1: | ||
| − | + | ||
| − | + | ||
| − | Copyright: ARM project http://www.metabolome.jp/ | + | Copyright: ARM project http://www.metabolome.jp/ |
| − | 64 69 0 0 1 0 0 0 0 0999 V2000 | + | 64 69 0 0 1 0 0 0 0 0999 V2000 |
| − | 10.1537 -0.7295 0.0000 C 0 0 2 0 0 0 0 0 0 0 0 0 | + | 10.1537 -0.7295 0.0000 C 0 0 2 0 0 0 0 0 0 0 0 0 |
| − | 11.0197 -0.2295 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0 | + | 11.0197 -0.2295 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0 |
| − | 10.6889 0.7885 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0 | + | 10.6889 0.7885 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0 |
| − | 9.6185 0.7885 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 9.6185 0.7885 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 9.1995 1.3717 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 9.1995 1.3717 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 8.5893 1.7502 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 8.5893 1.7502 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 8.2924 2.5686 0.0000 C 0 0 2 0 0 0 0 0 0 0 0 0 | + | 8.2924 2.5686 0.0000 C 0 0 2 0 0 0 0 0 0 0 0 0 |
| − | 7.4223 2.5391 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 7.4223 2.5391 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 7.1815 1.7025 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 7.1815 1.7025 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 6.5983 1.2836 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 6.5983 1.2836 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 6.2198 0.6733 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 6.2198 0.6733 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 5.4014 0.3764 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 5.4014 0.3764 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 5.4309 -0.4937 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 5.4309 -0.4937 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 6.2675 -0.7345 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 6.2675 -0.7345 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 6.6864 -1.3177 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 6.6864 -1.3177 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 7.2967 -1.6962 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 7.2967 -1.6962 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 7.5936 -2.5146 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 7.5936 -2.5146 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 8.4637 -2.4851 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 8.4637 -2.4851 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 8.7045 -1.6485 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 8.7045 -1.6485 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 9.2877 -1.2295 0.0000 C 0 0 2 0 0 0 0 0 0 0 0 0 | + | 9.2877 -1.2295 0.0000 C 0 0 2 0 0 0 0 0 0 0 0 0 |
| − | 9.2877 -0.2295 0.0000 N 0 0 0 0 0 0 0 0 0 0 0 0 | + | 9.2877 -0.2295 0.0000 N 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 7.8806 1.8664 0.0000 N 0 0 0 0 0 0 0 0 0 0 0 0 | + | 7.8806 1.8664 0.0000 N 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 6.1036 -0.0353 0.0000 N 0 0 0 0 0 0 0 0 0 0 0 0 | + | 6.1036 -0.0353 0.0000 N 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 8.0053 -1.8124 0.0000 N 0 0 0 0 0 0 0 0 0 0 0 0 | + | 8.0053 -1.8124 0.0000 N 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 11.6075 -1.0386 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 11.6075 -1.0386 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 11.6075 -0.4205 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 11.6075 -0.4205 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 11.6625 0.5365 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 11.6625 0.5365 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 11.2767 1.5975 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 11.2767 1.5975 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 12.2712 1.4930 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 12.2712 1.4930 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 12.8590 2.3020 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 12.8590 2.3020 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 9.2324 2.9097 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 9.2324 2.9097 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 8.2585 3.5680 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 8.2585 3.5680 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 9.1071 4.0971 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 9.1071 4.0971 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 6.8074 3.3277 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 6.8074 3.3277 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 7.1830 4.2545 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 7.1830 4.2545 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 6.5681 5.0432 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 6.5681 5.0432 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 4.5729 0.9364 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 4.5729 0.9364 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 4.6437 1.9339 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 4.6437 1.9339 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 4.6422 -1.1085 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 4.6422 -1.1085 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.7154 -0.7330 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.7154 -0.7330 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.9268 -1.3478 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.9268 -1.3478 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 7.0335 -3.3430 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 7.0335 -3.3430 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 7.4710 -4.2423 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 7.4710 -4.2423 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 6.9110 -5.0707 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 6.9110 -5.0707 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 9.8040 -4.2043 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 9.8040 -4.2043 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 9.4285 -5.1311 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 9.4285 -5.1311 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 10.1011 -1.8110 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 10.1011 -1.8110 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.0650 -2.3382 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.0650 -2.3382 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.0000 -0.9723 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.0000 -0.9723 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 5.5777 4.9050 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 5.5777 4.9050 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 6.9437 5.9700 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 6.9437 5.9700 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 9.9895 3.6267 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 9.9895 3.6267 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 9.0732 5.0965 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 9.0732 5.0965 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 12.4523 3.2156 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 12.4523 3.2156 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 13.8535 2.1975 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 13.8535 2.1975 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 5.5429 2.3714 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 5.5429 2.3714 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.8152 2.4940 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.8152 2.4940 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 5.9135 -5.0000 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 5.9135 -5.0000 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 7.3484 -5.9700 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 7.3484 -5.9700 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 8.4381 -5.2693 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 8.4381 -5.2693 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 10.0433 -5.9198 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 10.0433 -5.9198 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 12.4165 -1.6263 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 12.4165 -1.6263 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 11.0197 -1.2295 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 11.0197 -1.2295 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 9.3216 -2.2290 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 9.3216 -2.2290 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1 20 1 0 0 0 0 | + | 1 20 1 0 0 0 0 |
| − | 39 40 1 0 0 0 0 | + | 39 40 1 0 0 0 0 |
| − | 7 32 1 6 0 0 0 | + | 7 32 1 6 0 0 0 |
| − | 40 41 1 0 0 0 0 | + | 40 41 1 0 0 0 0 |
| − | 8 34 1 0 0 0 0 | + | 8 34 1 0 0 0 0 |
| − | 34 35 1 0 0 0 0 | + | 34 35 1 0 0 0 0 |
| − | 41 48 2 0 0 0 0 | + | 41 48 2 0 0 0 0 |
| − | 14 23 1 0 0 0 0 | + | 14 23 1 0 0 0 0 |
| − | 41 49 1 0 0 0 0 | + | 41 49 1 0 0 0 0 |
| − | 17 42 1 0 0 0 0 | + | 17 42 1 0 0 0 0 |
| − | 11 12 2 0 0 0 0 | + | 11 12 2 0 0 0 0 |
| − | 35 36 1 0 0 0 0 | + | 35 36 1 0 0 0 0 |
| − | 12 13 1 0 0 0 0 | + | 12 13 1 0 0 0 0 |
| − | 36 50 1 0 0 0 0 | + | 36 50 1 0 0 0 0 |
| − | 13 14 2 0 0 0 0 | + | 13 14 2 0 0 0 0 |
| − | 36 51 2 0 0 0 0 | + | 36 51 2 0 0 0 0 |
| − | 23 11 1 0 0 0 0 | + | 23 11 1 0 0 0 0 |
| − | 32 33 1 0 0 0 0 | + | 32 33 1 0 0 0 0 |
| − | 18 19 2 0 0 0 0 | + | 18 19 2 0 0 0 0 |
| − | 33 52 1 0 0 0 0 | + | 33 52 1 0 0 0 0 |
| − | 28 29 1 0 0 0 0 | + | 28 29 1 0 0 0 0 |
| − | 33 53 2 0 0 0 0 | + | 33 53 2 0 0 0 0 |
| − | 42 43 1 0 0 0 0 | + | 42 43 1 0 0 0 0 |
| − | 29 30 1 0 0 0 0 | + | 29 30 1 0 0 0 0 |
| − | 24 16 1 0 0 0 0 | + | 24 16 1 0 0 0 0 |
| − | 30 54 1 0 0 0 0 | + | 30 54 1 0 0 0 0 |
| − | 16 17 2 0 0 0 0 | + | 16 17 2 0 0 0 0 |
| − | 30 55 2 0 0 0 0 | + | 30 55 2 0 0 0 0 |
| − | 17 18 1 0 0 0 0 | + | 17 18 1 0 0 0 0 |
| − | 19 24 1 0 0 0 0 | + | 19 24 1 0 0 0 0 |
| − | 16 15 1 0 0 0 0 | + | 16 15 1 0 0 0 0 |
| − | 15 14 1 0 0 0 0 | + | 15 14 1 0 0 0 0 |
| − | 12 37 1 0 0 0 0 | + | 12 37 1 0 0 0 0 |
| − | 43 44 1 0 0 0 0 | + | 43 44 1 0 0 0 0 |
| − | 37 38 1 0 0 0 0 | + | 37 38 1 0 0 0 0 |
| − | 11 10 1 0 0 0 0 | + | 11 10 1 0 0 0 0 |
| − | 38 56 1 0 0 0 0 | + | 38 56 1 0 0 0 0 |
| − | 10 9 1 0 0 0 0 | + | 10 9 1 0 0 0 0 |
| − | 38 57 2 0 0 0 0 | + | 38 57 2 0 0 0 0 |
| − | 6 22 2 0 0 0 0 | + | 6 22 2 0 0 0 0 |
| − | 7 31 1 1 0 0 0 | + | 7 31 1 1 0 0 0 |
| − | 20 19 1 0 0 0 0 | + | 20 19 1 0 0 0 0 |
| − | 3 28 1 1 0 0 0 | + | 3 28 1 1 0 0 0 |
| − | 44 58 1 0 0 0 0 | + | 44 58 1 0 0 0 0 |
| − | 9 8 2 0 0 0 0 | + | 9 8 2 0 0 0 0 |
| − | 44 59 2 0 0 0 0 | + | 44 59 2 0 0 0 0 |
| − | 8 7 1 0 0 0 0 | + | 8 7 1 0 0 0 0 |
| − | 18 45 1 0 0 0 0 | + | 18 45 1 0 0 0 0 |
| − | 7 6 1 0 0 0 0 | + | 7 6 1 0 0 0 0 |
| − | 45 46 1 0 0 0 0 | + | 45 46 1 0 0 0 0 |
| − | 22 9 1 0 0 0 0 | + | 22 9 1 0 0 0 0 |
| − | 46 60 1 0 0 0 0 | + | 46 60 1 0 0 0 0 |
| − | 2 1 1 0 0 0 0 | + | 2 1 1 0 0 0 0 |
| − | 46 61 2 0 0 0 0 | + | 46 61 2 0 0 0 0 |
| − | 13 39 1 0 0 0 0 | + | 13 39 1 0 0 0 0 |
| − | 2 26 1 6 0 0 0 | + | 2 26 1 6 0 0 0 |
| − | 21 4 1 0 0 0 0 | + | 21 4 1 0 0 0 0 |
| − | 26 25 1 0 0 0 0 | + | 26 25 1 0 0 0 0 |
| − | 4 3 1 0 0 0 0 | + | 4 3 1 0 0 0 0 |
| − | 25 62 2 0 0 0 0 | + | 25 62 2 0 0 0 0 |
| − | 3 2 1 0 0 0 0 | + | 3 2 1 0 0 0 0 |
| − | 25 63 1 0 0 0 0 | + | 25 63 1 0 0 0 0 |
| − | 1 63 1 6 0 0 0 | + | 1 63 1 6 0 0 0 |
| − | 1 21 1 1 0 0 0 | + | 1 21 1 1 0 0 0 |
| − | 2 27 1 1 0 0 0 | + | 2 27 1 1 0 0 0 |
| − | 4 5 2 0 0 0 0 | + | 4 5 2 0 0 0 0 |
| − | 20 64 1 1 0 0 0 | + | 20 64 1 1 0 0 0 |
| − | 5 6 1 0 0 0 0 | + | 5 6 1 0 0 0 0 |
| − | 20 47 1 6 0 0 0 | + | 20 47 1 6 0 0 0 |
| − | S SKP 7 | + | S SKP 7 |
| − | ID BMCCPPPC0003 | + | ID BMCCPPPC0003 |
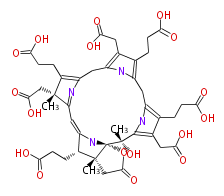
| − | NAME Precorrin 3B | + | NAME Precorrin 3B |
| − | FORMULA C43H50N4O17 | + | FORMULA C43H50N4O17 |
| − | EXACTMASS 894.317 | + | EXACTMASS 894.317 |
| − | AVERAGEMASS 894.8738 | + | AVERAGEMASS 894.8738 |
| − | SMILES [C@@](CC(O)=O)(C)(C6=1)C(=C(Cc(n2)c(c(CCC(O)=O)c2Cc(n3)c(c(CC(O)=O)c3[C@@](C)(O)[C@@]([C@]5(C)4)(NC(=C6)[C@@H](CCC(O)=O)5)OC(=O)C4)CCC(O)=O)CC(O)=O)N1)CCC(O)=O | + | SMILES [C@@](CC(O)=O)(C)(C6=1)C(=C(Cc(n2)c(c(CCC(O)=O)c2Cc(n3)c(c(CC(O)=O)c3[C@@](C)(O)[C@@]([C@]5(C)4)(NC(=C6)[C@@H](CCC(O)=O)5)OC(=O)C4)CCC(O)=O)CC(O)=O)N1)CCC(O)=O |
| − | KEGG http://www.genome.jp/dbget-bin/www_bget?compound+C06406 | + | KEGG http://www.genome.jp/dbget-bin/www_bget?compound+C06406 |
M END | M END | ||
| − | |||
Revision as of 09:00, 14 March 2009
Copyright: ARM project http://www.metabolome.jp/
64 69 0 0 1 0 0 0 0 0999 V2000
10.1537 -0.7295 0.0000 C 0 0 2 0 0 0 0 0 0 0 0 0
11.0197 -0.2295 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0
10.6889 0.7885 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0
9.6185 0.7885 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
9.1995 1.3717 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
8.5893 1.7502 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
8.2924 2.5686 0.0000 C 0 0 2 0 0 0 0 0 0 0 0 0
7.4223 2.5391 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
7.1815 1.7025 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
6.5983 1.2836 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
6.2198 0.6733 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
5.4014 0.3764 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
5.4309 -0.4937 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
6.2675 -0.7345 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
6.6864 -1.3177 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
7.2967 -1.6962 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
7.5936 -2.5146 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
8.4637 -2.4851 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
8.7045 -1.6485 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
9.2877 -1.2295 0.0000 C 0 0 2 0 0 0 0 0 0 0 0 0
9.2877 -0.2295 0.0000 N 0 0 0 0 0 0 0 0 0 0 0 0
7.8806 1.8664 0.0000 N 0 0 0 0 0 0 0 0 0 0 0 0
6.1036 -0.0353 0.0000 N 0 0 0 0 0 0 0 0 0 0 0 0
8.0053 -1.8124 0.0000 N 0 0 0 0 0 0 0 0 0 0 0 0
11.6075 -1.0386 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
11.6075 -0.4205 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
11.6625 0.5365 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
11.2767 1.5975 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
12.2712 1.4930 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
12.8590 2.3020 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
9.2324 2.9097 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
8.2585 3.5680 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
9.1071 4.0971 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
6.8074 3.3277 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
7.1830 4.2545 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
6.5681 5.0432 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
4.5729 0.9364 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
4.6437 1.9339 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
4.6422 -1.1085 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.7154 -0.7330 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.9268 -1.3478 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
7.0335 -3.3430 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
7.4710 -4.2423 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
6.9110 -5.0707 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
9.8040 -4.2043 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
9.4285 -5.1311 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
10.1011 -1.8110 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.0650 -2.3382 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
2.0000 -0.9723 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
5.5777 4.9050 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
6.9437 5.9700 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
9.9895 3.6267 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
9.0732 5.0965 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
12.4523 3.2156 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
13.8535 2.1975 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
5.5429 2.3714 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
3.8152 2.4940 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
5.9135 -5.0000 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
7.3484 -5.9700 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
8.4381 -5.2693 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
10.0433 -5.9198 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
12.4165 -1.6263 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
11.0197 -1.2295 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
9.3216 -2.2290 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
1 20 1 0 0 0 0
39 40 1 0 0 0 0
7 32 1 6 0 0 0
40 41 1 0 0 0 0
8 34 1 0 0 0 0
34 35 1 0 0 0 0
41 48 2 0 0 0 0
14 23 1 0 0 0 0
41 49 1 0 0 0 0
17 42 1 0 0 0 0
11 12 2 0 0 0 0
35 36 1 0 0 0 0
12 13 1 0 0 0 0
36 50 1 0 0 0 0
13 14 2 0 0 0 0
36 51 2 0 0 0 0
23 11 1 0 0 0 0
32 33 1 0 0 0 0
18 19 2 0 0 0 0
33 52 1 0 0 0 0
28 29 1 0 0 0 0
33 53 2 0 0 0 0
42 43 1 0 0 0 0
29 30 1 0 0 0 0
24 16 1 0 0 0 0
30 54 1 0 0 0 0
16 17 2 0 0 0 0
30 55 2 0 0 0 0
17 18 1 0 0 0 0
19 24 1 0 0 0 0
16 15 1 0 0 0 0
15 14 1 0 0 0 0
12 37 1 0 0 0 0
43 44 1 0 0 0 0
37 38 1 0 0 0 0
11 10 1 0 0 0 0
38 56 1 0 0 0 0
10 9 1 0 0 0 0
38 57 2 0 0 0 0
6 22 2 0 0 0 0
7 31 1 1 0 0 0
20 19 1 0 0 0 0
3 28 1 1 0 0 0
44 58 1 0 0 0 0
9 8 2 0 0 0 0
44 59 2 0 0 0 0
8 7 1 0 0 0 0
18 45 1 0 0 0 0
7 6 1 0 0 0 0
45 46 1 0 0 0 0
22 9 1 0 0 0 0
46 60 1 0 0 0 0
2 1 1 0 0 0 0
46 61 2 0 0 0 0
13 39 1 0 0 0 0
2 26 1 6 0 0 0
21 4 1 0 0 0 0
26 25 1 0 0 0 0
4 3 1 0 0 0 0
25 62 2 0 0 0 0
3 2 1 0 0 0 0
25 63 1 0 0 0 0
1 63 1 6 0 0 0
1 21 1 1 0 0 0
2 27 1 1 0 0 0
4 5 2 0 0 0 0
20 64 1 1 0 0 0
5 6 1 0 0 0 0
20 47 1 6 0 0 0
S SKP 7
ID BMCCPPPC0003
NAME Precorrin 3B
FORMULA C43H50N4O17
EXACTMASS 894.317
AVERAGEMASS 894.8738
SMILES [C@@](CC(O)=O)(C)(C6=1)C(=C(Cc(n2)c(c(CCC(O)=O)c2Cc(n3)c(c(CC(O)=O)c3[C@@](C)(O)[C@@]([C@]5(C)4)(NC(=C6)[C@@H](CCC(O)=O)5)OC(=O)C4)CCC(O)=O)CC(O)=O)N1)CCC(O)=O
KEGG http://www.genome.jp/dbget-bin/www_bget?compound+C06406
M END