Mol:BMCCPPHM0004
From Metabolomics.JP
(Difference between revisions)
| (One intermediate revision by one user not shown) | |||
| Line 1: | Line 1: | ||
| − | + | ||
| − | + | ||
| − | Copyright: ARM project http://www.metabolome.jp/ | + | Copyright: ARM project http://www.metabolome.jp/ |
| − | 42 49 0 0 0 0 0 0 0 0999 V2000 | + | 42 49 0 0 0 0 0 0 0 0999 V2000 |
| − | 4.4013 0.7407 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 4.4013 0.7407 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.7226 0.9612 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.7226 0.9612 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.7226 1.6749 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.7226 1.6749 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 4.4013 1.8954 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 4.4013 1.8954 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 4.6126 2.6841 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 4.6126 2.6841 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 5.4013 2.8954 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 5.4013 2.8954 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 5.6218 3.5741 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 5.6218 3.5741 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 6.3355 3.5741 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 6.3355 3.5741 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 6.5560 2.8954 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 6.5560 2.8954 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 7.3447 2.6841 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 7.3447 2.6841 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 7.5560 1.8954 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 7.5560 1.8954 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 8.2347 1.6749 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 8.2347 1.6749 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 8.2347 0.9612 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 8.2347 0.9612 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 7.5560 0.7407 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 7.5560 0.7407 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 7.3447 -0.0480 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 7.3447 -0.0480 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 6.5560 -0.2593 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 6.5560 -0.2593 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 6.3355 -0.9380 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 6.3355 -0.9380 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 5.6218 -0.9380 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 5.6218 -0.9380 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 5.4013 -0.2593 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 5.4013 -0.2593 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 4.6126 -0.0480 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 4.6126 -0.0480 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 4.9786 1.3181 0.0000 N 0 0 0 0 0 0 0 0 0 0 0 0 | + | 4.9786 1.3181 0.0000 N 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 5.9786 2.3181 0.0000 N 0 0 0 0 0 0 0 0 0 0 0 0 | + | 5.9786 2.3181 0.0000 N 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 6.9786 1.3181 0.0000 N 0 0 0 0 0 0 0 0 0 0 0 0 | + | 6.9786 1.3181 0.0000 N 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 5.9786 0.3181 0.0000 N 0 0 0 0 0 0 0 0 0 0 0 0 | + | 5.9786 0.3181 0.0000 N 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.9135 0.3734 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.9135 0.3734 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.9135 2.2627 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.9135 2.2627 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.0000 1.8559 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.0000 1.8559 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 5.0340 4.3831 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 5.0340 4.3831 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 6.9232 4.3831 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 6.9232 4.3831 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 9.2347 1.6749 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 9.2347 1.6749 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 9.0437 0.3734 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 9.0437 0.3734 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 9.9573 0.7802 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 9.9573 0.7802 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 10.7663 0.1924 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 10.7663 0.1924 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 11.6798 0.5991 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 11.6798 0.5991 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 6.9232 -1.7470 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 6.9232 -1.7470 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 6.5165 -2.6606 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 6.5165 -2.6606 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 7.1043 -3.4696 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 7.1043 -3.4696 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 8.0988 -3.3651 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 8.0988 -3.3651 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 5.0340 -1.7470 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 5.0340 -1.7470 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 5.9786 1.3181 0.0000 Fe 0 2 0 0 0 0 0 0 0 0 0 0 | + | 5.9786 1.3181 0.0000 Fe 0 2 0 0 0 0 0 0 0 0 0 0 |
| − | 10.6617 -0.8021 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 10.6617 -0.8021 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 6.6975 -4.3831 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 6.6975 -4.3831 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 4 21 1 0 0 0 0 | + | 4 21 1 0 0 0 0 |
| − | 4 5 1 0 0 0 0 | + | 4 5 1 0 0 0 0 |
| − | 5 6 2 0 0 0 0 | + | 5 6 2 0 0 0 0 |
| − | 6 22 1 0 0 0 0 | + | 6 22 1 0 0 0 0 |
| − | 21 1 1 0 0 0 0 | + | 21 1 1 0 0 0 0 |
| − | 1 20 1 0 0 0 0 | + | 1 20 1 0 0 0 0 |
| − | 20 19 2 0 0 0 0 | + | 20 19 2 0 0 0 0 |
| − | 19 24 1 0 0 0 0 | + | 19 24 1 0 0 0 0 |
| − | 1 2 2 0 0 0 0 | + | 1 2 2 0 0 0 0 |
| − | 2 3 1 0 0 0 0 | + | 2 3 1 0 0 0 0 |
| − | 3 4 2 0 0 0 0 | + | 3 4 2 0 0 0 0 |
| − | 24 16 2 0 0 0 0 | + | 24 16 2 0 0 0 0 |
| − | 16 15 1 0 0 0 0 | + | 16 15 1 0 0 0 0 |
| − | 15 14 2 0 0 0 0 | + | 15 14 2 0 0 0 0 |
| − | 14 23 1 0 0 0 0 | + | 14 23 1 0 0 0 0 |
| − | 23 11 1 0 0 0 0 | + | 23 11 1 0 0 0 0 |
| − | 11 10 2 0 0 0 0 | + | 11 10 2 0 0 0 0 |
| − | 10 9 1 0 0 0 0 | + | 10 9 1 0 0 0 0 |
| − | 9 22 2 0 0 0 0 | + | 9 22 2 0 0 0 0 |
| − | 19 18 1 0 0 0 0 | + | 19 18 1 0 0 0 0 |
| − | 18 17 2 0 0 0 0 | + | 18 17 2 0 0 0 0 |
| − | 17 16 1 0 0 0 0 | + | 17 16 1 0 0 0 0 |
| − | 14 13 1 0 0 0 0 | + | 14 13 1 0 0 0 0 |
| − | 13 12 2 0 0 0 0 | + | 13 12 2 0 0 0 0 |
| − | 12 11 1 0 0 0 0 | + | 12 11 1 0 0 0 0 |
| − | 9 8 1 0 0 0 0 | + | 9 8 1 0 0 0 0 |
| − | 8 7 2 0 0 0 0 | + | 8 7 2 0 0 0 0 |
| − | 7 6 1 0 0 0 0 | + | 7 6 1 0 0 0 0 |
| − | 21 40 1 0 0 0 0 | + | 21 40 1 0 0 0 0 |
| − | 40 23 1 0 0 0 0 | + | 40 23 1 0 0 0 0 |
| − | 22 40 1 0 0 0 0 | + | 22 40 1 0 0 0 0 |
| − | 40 24 1 0 0 0 0 | + | 40 24 1 0 0 0 0 |
| − | 2 25 1 0 0 0 0 | + | 2 25 1 0 0 0 0 |
| − | 3 26 1 0 0 0 0 | + | 3 26 1 0 0 0 0 |
| − | 26 27 2 0 0 0 0 | + | 26 27 2 0 0 0 0 |
| − | 7 28 1 0 0 0 0 | + | 7 28 1 0 0 0 0 |
| − | 8 29 1 0 0 0 0 | + | 8 29 1 0 0 0 0 |
| − | 12 30 1 0 0 0 0 | + | 12 30 1 0 0 0 0 |
| − | 13 31 1 0 0 0 0 | + | 13 31 1 0 0 0 0 |
| − | 31 32 1 0 0 0 0 | + | 31 32 1 0 0 0 0 |
| − | 32 33 1 0 0 0 0 | + | 32 33 1 0 0 0 0 |
| − | 33 41 2 0 0 0 0 | + | 33 41 2 0 0 0 0 |
| − | 33 34 1 0 0 0 0 | + | 33 34 1 0 0 0 0 |
| − | 17 35 1 0 0 0 0 | + | 17 35 1 0 0 0 0 |
| − | 35 36 1 0 0 0 0 | + | 35 36 1 0 0 0 0 |
| − | 36 37 1 0 0 0 0 | + | 36 37 1 0 0 0 0 |
| − | 37 38 1 0 0 0 0 | + | 37 38 1 0 0 0 0 |
| − | 37 42 2 0 0 0 0 | + | 37 42 2 0 0 0 0 |
| − | 18 39 1 0 0 0 0 | + | 18 39 1 0 0 0 0 |
| − | S SKP 7 | + | S SKP 7 |
| − | ID BMCCPPHM0004 | + | ID BMCCPPHM0004 |
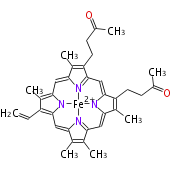
| − | NAME | + | NAME 3- [18- (2-carboxyethyl) -8-ethenyl-3,7,12,13,17-pentamethylporphyrin-22,24-diid-2-yl] propanoic acid; iron(4+) |
| − | FORMULA C35H36FeN4O2 | + | FORMULA C35H36FeN4O2 |
| − | EXACTMASS 600.2187 | + | EXACTMASS 600.2187 |
| − | AVERAGEMASS 600.5311 | + | AVERAGEMASS 600.5311 |
| − | SMILES C(C=84)(=C(C)C(N87)=Cc(c1C=C)n([Fe+2]723)c(C=C(C=6C)N2=C(C6CCC(C)=O)C=c(c(CCC(C)=O)5)n(c(c5C)=C4)3)c(C)1)C | + | SMILES C(C=84)(=C(C)C(N87)=Cc(c1C=C)n([Fe+2]723)c(C=C(C=6C)N2=C(C6CCC(C)=O)C=c(c(CCC(C)=O)5)n(c(c5C)=C4)3)c(C)1)C |
| − | KEGG http://www.genome.jp/dbget-bin/www_bget?compound+C00924 | + | KEGG http://www.genome.jp/dbget-bin/www_bget?compound+C00924 |
M END | M END | ||
| − | |||
Latest revision as of 11:42, 16 June 2010
Copyright: ARM project http://www.metabolome.jp/
42 49 0 0 0 0 0 0 0 0999 V2000
4.4013 0.7407 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.7226 0.9612 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.7226 1.6749 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
4.4013 1.8954 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
4.6126 2.6841 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
5.4013 2.8954 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
5.6218 3.5741 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
6.3355 3.5741 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
6.5560 2.8954 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
7.3447 2.6841 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
7.5560 1.8954 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
8.2347 1.6749 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
8.2347 0.9612 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
7.5560 0.7407 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
7.3447 -0.0480 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
6.5560 -0.2593 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
6.3355 -0.9380 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
5.6218 -0.9380 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
5.4013 -0.2593 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
4.6126 -0.0480 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
4.9786 1.3181 0.0000 N 0 0 0 0 0 0 0 0 0 0 0 0
5.9786 2.3181 0.0000 N 0 0 0 0 0 0 0 0 0 0 0 0
6.9786 1.3181 0.0000 N 0 0 0 0 0 0 0 0 0 0 0 0
5.9786 0.3181 0.0000 N 0 0 0 0 0 0 0 0 0 0 0 0
2.9135 0.3734 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.9135 2.2627 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.0000 1.8559 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
5.0340 4.3831 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
6.9232 4.3831 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
9.2347 1.6749 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
9.0437 0.3734 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
9.9573 0.7802 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
10.7663 0.1924 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
11.6798 0.5991 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
6.9232 -1.7470 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
6.5165 -2.6606 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
7.1043 -3.4696 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
8.0988 -3.3651 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
5.0340 -1.7470 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
5.9786 1.3181 0.0000 Fe 0 2 0 0 0 0 0 0 0 0 0 0
10.6617 -0.8021 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
6.6975 -4.3831 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
4 21 1 0 0 0 0
4 5 1 0 0 0 0
5 6 2 0 0 0 0
6 22 1 0 0 0 0
21 1 1 0 0 0 0
1 20 1 0 0 0 0
20 19 2 0 0 0 0
19 24 1 0 0 0 0
1 2 2 0 0 0 0
2 3 1 0 0 0 0
3 4 2 0 0 0 0
24 16 2 0 0 0 0
16 15 1 0 0 0 0
15 14 2 0 0 0 0
14 23 1 0 0 0 0
23 11 1 0 0 0 0
11 10 2 0 0 0 0
10 9 1 0 0 0 0
9 22 2 0 0 0 0
19 18 1 0 0 0 0
18 17 2 0 0 0 0
17 16 1 0 0 0 0
14 13 1 0 0 0 0
13 12 2 0 0 0 0
12 11 1 0 0 0 0
9 8 1 0 0 0 0
8 7 2 0 0 0 0
7 6 1 0 0 0 0
21 40 1 0 0 0 0
40 23 1 0 0 0 0
22 40 1 0 0 0 0
40 24 1 0 0 0 0
2 25 1 0 0 0 0
3 26 1 0 0 0 0
26 27 2 0 0 0 0
7 28 1 0 0 0 0
8 29 1 0 0 0 0
12 30 1 0 0 0 0
13 31 1 0 0 0 0
31 32 1 0 0 0 0
32 33 1 0 0 0 0
33 41 2 0 0 0 0
33 34 1 0 0 0 0
17 35 1 0 0 0 0
35 36 1 0 0 0 0
36 37 1 0 0 0 0
37 38 1 0 0 0 0
37 42 2 0 0 0 0
18 39 1 0 0 0 0
S SKP 7
ID BMCCPPHM0004
NAME 3- [18- (2-carboxyethyl) -8-ethenyl-3,7,12,13,17-pentamethylporphyrin-22,24-diid-2-yl] propanoic acid; iron(4+)
FORMULA C35H36FeN4O2
EXACTMASS 600.2187
AVERAGEMASS 600.5311
SMILES C(C=84)(=C(C)C(N87)=Cc(c1C=C)n([Fe+2]723)c(C=C(C=6C)N2=C(C6CCC(C)=O)C=c(c(CCC(C)=O)5)n(c(c5C)=C4)3)c(C)1)C
KEGG http://www.genome.jp/dbget-bin/www_bget?compound+C00924
M END